Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Salud Animal
versión impresa ISSN 0253-570X
Rev Salud Anim. v.31 n.2 La Habana Mayo-ago. 2009
Artículo original
EVALUACIÓN DE DIFERENTES MÉTODOS DE EXTRACCIÓN DE ADN DE MICOPLASMAS PARA SU EMPLEO EN EL DIAGNÓSTICO POR PCR
EVALUATION OF DIFFERENT DNA EXTRACTION METHODS FROM MYCOPLASMA TO BE EMPLOYEES IN THE DIAGNOSTIC BY PCR
Yenney Hernández, Evelyn Lobo, Siomara Martínez y Loidy Zamora
Dirección de Microbiología, Grupo Biología Molecular, Laboratorio de Diagnóstico de Micoplasmas (MYCOLAB), Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, La Habana, Cuba. Correo electrónico: yeni@censa.edu.cu
RESUMEN
El objetivo del presente trabajo fue evaluar diferentes métodos de extracción de ADN de micoplasmas, para su empleo en el diagnóstico por PCR. La caracterización de la cepa de Mycoplasma arginini utilizada como control positivo, se realizó con el empleo de los estudios morfológicos y las pruebas bioquímicas y enzimáticas. A continuación se procedió a la obtención de un cultivo homogéneo de la cepa de Mycoplasma arginini, que permitiera evaluar tres métodos de extracción de ADN de micoplasmas para su utilización en el ensayo de PCR. Se probaron los métodos de extracción fenol - cloroformo, centrifugación y centrifugación con choque térmico en muestras clínicas de cultivos celulares, sueros y productos biofarmacéuticos, que habían sido testadas por el método de cultivo y la Hibridación de Ácidos Nucleicos (HAN). Como resultado se obtuvo la cepa de Mycoplasma arginini caracterizada y evaluada. Tanto el método de fenol-cloroformo, como la centrifugación y centrifugación con choque térmico, más sencillos, brindaron similares resultados cuando se realizó la PCR utilizando la cepa tipo de Mycoplasma arginini. Se demostró que los métodos de choque térmico y centrifugación son eficientes para la obtención de ADN de micoplasmas a partir de muestras clínicas con el empleo de la PCR, comparándose estos resultados con los obtenidos por cultivo y la HAN.
Palabras clave: micoplasmas; diagnóstico; cultivos celulares; PCR
ABSTRACT
The aim of this work was to evaluate different DNA extraction methods of mycoplasma with the use of PCR. The characterization of Mycoplasma arginini strain use as positive control in this test was carried out, by means of morphological studies and biochemical and enzymatic tests, in order to obtain a homogenous culture, which allowed evaluating three mycoplasma DNA extraction methods for their use in the PCR test. Methods based on centrifuge and thermal shock with centrifuge were proven for the DNA extraction in cell culture clinical samples, sera and biopharmaceutical products, which had been tested by culture method and Nucleic Acid Hybridization (NAH). Mycoplasma arginini, strain was characterized and evaluated. Either phenol-chloroform extraction as well as two more simple methods describe above, brought the same results when the PCR assay was carried out using M. arginini strain. It has been demonstrated that the methods based on centrifuge and thermal shock with centrifuge are efficient to obtain Mycoplasma DNA from clinical samples with the use of PCR, comparing these results with those obtained by culture method and NAH.
Key words: mycoplasmas; diagnostic; cell cultures; PCR
INTRODUCCIÓN
La necesidad de controlar estrictamente las contaminaciones por micoplasmas en cultivos celulares, sueros y productos biofarmacéuticos de aplicación biomédica, está basada en los efectos indeseables que producen estos microorganismos en los cultivos celulares infectados, así como en el individuo que recibe estos productos contaminados (1). Debido a esto las agencias reguladoras han establecido normas muy estrictas relacionadas con la detección de contaminaciones y entre ellas, específicamente, la detección de micoplasmas (2; 3).
Numerosos métodos se han utilizado para lograr el aislamiento, detección e identificación de los micoplasmas: los cultivos microbiológicos, las pruebas bioquímicas y enzimáticas, tinción fluorescente del ADN y las técnicas moleculares dentro de las que se encuentran la Hibridación de ácidos nucleicos (HAN) y la Reacción en cadena de la polimerasa (PCR) (4).
Los micoplasmas como característica distintiva carecen de pared celular, lo cual ha permitido desarrollar diferentes métodos de extracción de ADN mucho más sencillos, basados en choques térmicos o ruptura mecánica, para ser empleados en los ensayos de PCR, además de tener en cuenta las grandes ventajas que presenta esta técnica como son su gran sensibilidad, especificidad y rapidez en el diagnóstico (5). Por tales antecedentes, recientemente los organismos reguladores internacionales como la Farmacopea Europea, 2007 (3) y el Manual de OIE, 2004 (6) han aprobado esta técnica, como otros de los métodos para el diagnóstico de micoplasmas en cultivos celulares, sueros y productos biofarmacéuticos de aplicación biomédica, que junto al cultivo microbiológico y la Tinción de ADN, permita la liberación de estos productos con la calidad requerida.
Teniendo en cuenta lo anteriormente planteado es que nos propusimos como objetivo evaluar diferentes métodos de extracción de ADN para la detección de micoplasmas, mediante el empleo de un ensayo de PCR.
MATERIALES Y MÉTODOS
CEPAS DE MICOPLASMA UTILIZADAS
• Cepa de Mycoplasma arginin NCTC 10129 (Nacional Colecction of Type Culture, de la Agencia de Protección a la Salud, Inglaterra), empleada como control positivo para la técnica de PCR, según lo recomienda la Farmacopea Europea, 2007 (3).
• Cepa de Mycoplasma gallinarum, cepa de colección del PDRC (Poultry Diagnostic Reseach Center, Atlanta University, USA, utilizada como control positivo en la prueba de la producción de Film y Spot.
Siembra de la cepa: El cultivo de la cepa se realizó según lo descrito por Poveda (7). Se incubó durante 15 días en condiciones de microaereofilia y
anaerobiosis para favorecer el crecimiento de los micoplasmas, realizándose lecturas al microscopio cada 48 horas.
Estudio morfológico: Se siguió como criterio de caracterización la formación de colonias pequeñas, entre 50 a 500 mm de diámetro (7).
Una vez observado el crecimiento de la cepa en forma de huevo frito, se subcultivó en medio Hayflick líquido para su posterior identificación, incubándose a 370C en condiciones de aerobiosis durante 15 días, realizando lecturas diarias para determinar cambios de pH en el medio que indicara crecimiento de micoplasmas.
Pruebas bioquímicas y enzimáticas: Los procedimientos bioquímicos (prueba de la Digitonina, fermentación de la glucosa, hidrólisis de la arginina y producción de Film y Spot) para la identificación de micoplasmas se realizaron según el protocolo descrito por Poveda, 1998 (7) y de acuerdo a los criterios de clasificación del Manual del Bergey (8).
OBTENCIÓN DEL CULTIVO DE LA CEPA A UTILIZAR COMO CONTROL POSITIVO
Se partió de la cepa crecida en placa y se sembró en 5 mL de medio Hayflick líquido y se incubó a 370C por 48 horas hasta que alcanzara DO540nm entre 0.2-0.4 y un pH entre 7.4-7.5. Posteriormente se procedió de igual manera y bajo las mismas condiciones de crecimiento hasta obtener un cultivo crecido en un volumen final de 100 mL (9).
EVALUACIÓN DE TRES MÉTODOS DE EXTRACCIÓN DE ADN DE LA CEPA DE Mycoplasma arginini, UTILIZADA COMO CONTROL POSITIVO, MEDIANTE EL EMPLEO DE UN ENSAYO DE PCR.
Método I: Extracción con Fenol-Cloroformo (10).
Método II: Se tomaron 25 mL del cultivo de micoplasma y se centrifugaron a 12 000 rpm durante 10 minutos, el sedimento se resuspendió en 25 mL de PBS y se centrifugó nuevamente a 12 000 rpm durante 10 minutos. Se eliminó el sobrenadante y se resuspendió en 2.5 mL de agua destilada estéril, se hirvió durante 10 minutos y rápidamente se colocó en hielo. El extracto se conservó a -200C hasta su posterior utilización (11).
Método III: Se hirvieron 25 mL del cultivo de micoplasma durante 10 minutos y posteriormente se centrifugaron a 10 000 rpm por 10 minutos. Se tomó el pellet y se resuspendió en 2.5 mL de agua destilada estéril y se conservó a -200C hasta su posterior utilización (12, 13).
Electroforesis del ADN extraído: Se realizó una electroforesis en gel de agarosa al 0.8% teñido con bromuro de etidio (0.5 ìg/mL), corrida a 90 volts por 35 minutos, para analizar la presencia y calidad del ADN obtenido por los tres métodos.
Cebadores utilizados: Se utilizó un pareja de cebadores reportado por Fernández y Chávez, 1999 (11) que amplifica una región de 1200pb, correspondiente a la región conservada del ARNr 16S de Mollicutes (14). Los cebadores fueron sintetizados en el Centro de Ingeniería Genética y Biotecnología (CIGB).
Cebador MGSO:
5´ AGA GTT TGA TCC TGG CTC AGG A 3´
Cebador GPO-1:
5´ GGT AGG GAT ACC TTG TTA CGA CT 3´
Preparación de la mezcla
La amplificación se realizó en un volumen final de 50 uL donde se utilizó 2.5 uL para la muestra de ADN extraído con fenol cloroformo y 5 uL para las muestras provenientes de los otros dos métodos de extracción descritos. La mezcla de reacción contenía 5 uL de solución tampón 1X (10 mM de KCL, 200 mM de Tris HCI pH 8.8; 2.5 mM de MgCl2; 0.1% de Triton X-100 Promega); 10 mM de los dNTP (Promega); 20 pmoles de cada cebador y 1 ìL de la enzima AmpliCEN (CENSA).
Condiciones de la reacción
Las reacciones se realizaron en un equipo Termociclador de ADN REACTOR ThermoHyBaidTM, utilizando el siguiente programa de amplificación: 1 ciclo de 940C por 4 minutos; 39 ciclos de 940C /1 minuto; 550C/1 minuto; 720C/ 2 minutos y 1 ciclo de 720C por 8 minutos. La electroforesis se realizó en gel de agarosa al 1.2% teñido con bromuro de etidio (0.5 ug/mL), corrida a 90 volts por 35 minutos. El resultado se visualizó en un trasluminador de luz ultravioleta (MacroVue). Se utilizó un patrón de peso molecular con una talla de 150 a 2000pb (Promega).
EVALUACIÓN DE LOS MÉTODOS DE EXTRACCIÓN DE ADN II Y III EN MUESTRAS CLÍNICAS, MEDIANTE LA REALIZACIÓN DEL PCR
Tipo de muestras
Se analizó un total de 56 muestras, que corresponden con las muestras de cultivos y líneas celulares, suero fetal bovino, tripsina, líquido ascítico, productos biofarmacéuticos de aplicación biomédica procedentes del CIGB y CIM. Las mismas habían sido analizadas con anterioridad por el método de cultivo y la HAN.
En la extracción del ADN de micoplasmas provenientes de las muestras clínicas, se empleó el método II y III descritos anteriormente y la preparación de la mezcla y realización del PCR, se realizaron bajo las mismas condiciones y requerimientos que el experimento anterior.
RESULTADOS Y DISCUSIÓN
A pesar del incremento en el empleo de las técnicas moleculares para la caracterización de cepas de micoplasmas, el estudio morfológico y el comportamiento frente a las diferentes pruebas bioquímicas y enzimáticas continúan siendo recomendadas por la literatura especializada para este fin (1).
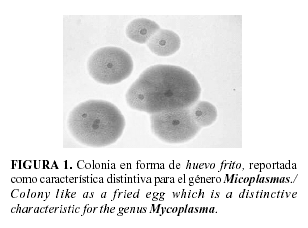
Los resultados obtenidos como parte de la caracterización de la cepa en cuanto al análisis morfológico se puede observar en la Figura 1, donde se muestra la apariencia microscópica de las colonias de este cultivo a las 48 horas de incubación. En la misma se puede apreciar la conocida forma de huevo frito reportada para el género Mycoplasma como característica distintiva de los Mollicutes, consistente en una zona central granular opaca embebida en el agar y una zona traslúcida periférica (7).
El comportamiento de estos microorganismos frente a la fermentación de la glucosa, la hidrólisis de la arginina, la producción de Film y Spot, prueba de la digitonina, entre otras, constituyen una herramienta importante en la caracterización de estos agentes (8). En la Tabla 1 se muestran los resultados obtenidos en la caracterización de la cepa de Mycoplasma arginini utilizada como control positivo, frente a las diferentes pruebas bioquímicas y enzimáticas.

El comportamiento de la cepa frente a las diferentes pruebas, coincide con lo reportado como característico para esta especie, según Manual de Microbiología Clínica (15) y Manual del Bergey (8).
El resultado frente a la prueba de la digitonina confirma su identidad como microorganismo perteneciente al género Mycoplasma. En la Figura 2 se puede observar el halo de inhibición producido por la ausencia de crecimiento en dicha región.

A partir de estos resultados se obtuvo un cultivo con el pH y DO requeridos según lo referido en el Manual de Obtención del Antígeno (9), donde se señala que con valores de pH entre 7.4 y 7.5 y una absorbancia entre 0.2 y 0.4 son los óptimos valores en cultivo para ser utilizado como control positivo en el diagnóstico de micoplasmas. En nuestro caso, el cultivo tenía un pH de 7.4 y una absorbancia de 0.23. Por otra parte, una vez lograda su obtención, dicho cultivo mantuvo sus propiedades morfológicas y bioquímicas estables, lo cual posibilitó contar con un cultivo homogéneo, que permitiera la extracción del ADN de esta especie por los diferentes métodos, partiendo de las mismas condiciones.
El análisis bacteriológico del cultivo de la cepa resultó negativo, lo cual demostró que el mismo no presentaba contaminación bacteriana.
En la Figura 3 se evidencian los resultados obtenidos en la electroforesis analítica realizada al ADN de la cepa Mycoplasma arginini obtenido a partir de los tres métodos de extracción.

En la Figura 3 solo se observa el ADN extraído por el método de fenol/cloroformo, debido a que el material genético se encuentra purificado y separado del resto del material celular, libre de inhibidores y de las histonas que son degradadas por la proteinasa K, lo cual concuerda con lo reportado por Bashiruddin, 1998 (10) y Harasawa, 2004 (16). Por otra parte, se observa la banda de ADN obtenida por este procedimiento, por encima del marcador de peso molecular, lo cual se debe a que la talla del genoma de micoplasmas es de aproximadamente 2 Kb (17).
A diferencia del método de fenol/cloroformo, con los otros dos métodos no se observó ninguna banda que indicara presencia de ADN. Esto se debe a que los mismos se basan en la ruptura mecánica o por choque térmico, obteniéndose ADN geonómico unido al material celular y a proteínas desnaturalizadas y por tanto no se encuentra totalmente expuesto para ser detectado en este tipo de electroforesis analítica, realizada con el objetivo de observar la calidad y pureza del ADN extraído, coincidiendo estos resultados con lo reportado por Bashiruddin, 1998 (10) y Reina, 2003 (1).
Sin embargo, cuando se realizó la PCR utilizando el ADN de Mycoplasma arginini extraído por los tres métodos, se pudo observar la presencia de un producto amplificado de aproximadamente 1200 pb, el cual coincide con el control positivo de la reacción (Figura 4).

Como se muestra en la Figura 4 (a), los tres métodos empleados en la extracción del ADN de la cepa utilizada, permitieron la amplificación del material genético de micoplasmas, coincidiendo con lo reportado por Nissen et al. (18); Uphoff y Drexler (19) y Chaudhry et al. (20) quienes describen que no es necesario tener un ADN puro, es decir expuesto o libre de material celular para que pueda ser detectado por esta técnica, lo que se debe al empleo de cebadores que reconocen una región específica del ADN de estos microorganismos. Unido a esto también se encuentra la capacidad del PCR de amplificar ADN presente en la muestra en muy pequeñas concentraciones, incluso hasta degradado, siempre que no se afecte la región de interés, lo cual avala la gran sensibilidad y especificidad de este sistema (12).
A pesar de la calidad del ADN obtenido por el método fenol/cloroformo, este procedimiento implica una mayor manipulación de la muestra, un mayor tiempo de realización, así como el empleo de reactivos que encarecen el diagnóstico, los cuales en muchas ocasiones suelen dañar la salud del personal. Por tales motivos, se dificulta su empleo para el tratamiento de muestras clínicas en el diagnóstico de rutina de micoplasmas (21).
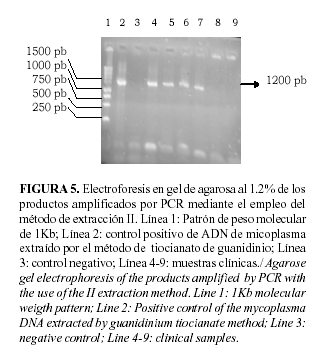
A partir de los resultados obtenidos en la extracción de ADN de la cepa de Mycoplasma arginini, con el sistema de PCR, se decidió emplear el método II y III para la extracción de ADN de micoplasmas, a partir de muestras clínicas de cultivos celulares, sueros y productos biofarmacéuticos de aplicación biomédica. En las Figuras 5 y 6 se observan los resultados con los métodos II y III respectivamente.

Los micoplasmas al carecer de pared celular, son susceptibles a la lisis por temperaturas elevadas, permitiendo por tanto, la accesibilidad del material genético, lo cual justifica la aplicabilidad de los métodos II y III basados en choques térmicos, para su empleo en los ensayos de PCR en el diagnóstico de los micoplasmas, lo cual coincide con lo reportado por Fernández y Chávez (11) y Sung et al. (4). Además, se debe tener en consideración las grandes ventajas de los mismos, que a diferencia de la extracción con fenol/cloroformo, no se necesita gran manipulación, siendo muy sencillos y rápidos y no se requiere del uso de reactivos que hagan más engorroso el trabajo.
Por otra parte en la Tabla 2 se evidencian los resultados obtenidos en el ensayo de PCR frente a las diferentes muestras clínicas procesadas por los métodos de extracción de ADN II y III, en comparación con los resultados registrados por el método de cultivo y la HAN.

Según se observa en la Tabla 2 la PCR detectó mayor cantidad de muestras positivas que el cultivo y la HAN. En comparación con el cultivo, diferentes autores refieren la ventaja que reporta la PCR de detectar organismos no viables en contraposición al cultivo que solo detecta microorganismos vivos (18; 23). Además se debe tener en cuenta que es un método muy rápido, que a diferencia del cultivo, a las 24 horas se puede brindar un resultado con la calidad y confianza requeridas. Una de las razones por la que el cultivo demora tanto es que como parte de la rutina del laboratorio se añaden antibióticos a los cultivos para evitar posibles contaminaciones bacterianas, las cuales tienden a enmascarar la presencia de micoplasmas, impidiendo su crecimiento.
La HAN al igual que la PCR, es capaz de detectar microorganismos no viables, pero solo detecta hasta 104 UFC/mL, a diferencia de la PCR que según Razin, 1994 (22) y Tang et.al , 2000(24) es capaz de amplificar por sistemas simples hasta 102 UFC/mL. En ocasiones, esta sensibilidad puede ser mucho mayor cuando se emplean sistemas como la PCR anidada y/o RT-PCR, llegando a detectar 1 UFC/mL. Además, la HAN implica un mayor tiempo de realización, así como el empleo de isótopos radioactivos que ponen en riesgo la salud del personal.
Como resultado de este trabajo se pudo comprobar que los métodos de extracción II y III empleados fueron capaces de detectar el ADN de micoplasmas en las muestras clínicas analizadas. Por las ventajas que presenta el método III, más sencillo que el método II, proponemos sea empleado en el diagnóstico de micoplasmas por PCR, con el objetivo de brindar un servicio mucho más rápido, menos costoso, confiable y sencillo. Además con este trabajo se pudo corroborar que la PCR constituye una técnica de gran utilidad en el diagnóstico de rutina de micoplasmas en cultivos celulares, sueros y productos biofarmacéuticos.
REFERENCIAS
1. Reina M. Las contaminaciones. Técnicas de estudio de líneas celulares. Proc Soc Exp Biol Med. 2003;122:478.
2. Monitoring Mycoplasma Test Meeting (AMPIS). Responses to the Issues Raised at the VICH-WG Biological Quality. 2001; 11-13.
3. Farmacopea Europea 5.8. Mycoplasmas. Appendix 2.6.7. 2007.
4. Sung H, Hye S, Jin Y, Tae J, Rok Y, Lee C, et al. PCR-Based Detection of Mycoplasmas Species. J. Microbiol. 2006;44(1):42-49.
5. Menéndez S, Pérez E. La PCR múltiple en microbiología clínica. Departamento de Microbiología y Biología Celular. Universidad de la Laguna. Santa Cruz de Tenerife. España. 2004.
6. OIE Manual of Diagnostic Tests and Vaccines for Terrestrial Animals. Validation and quality control of polymerase chain reaction methods used for the diagnosis of infectious diseases. Office International Des. Epizooties. 2004; Chapter 1.1.4. pp: 1-10.
7. Poveda JB. Biochemical Characteristic in Mycoplasma identification. In: Miles RJ, Nicholas RA, editors. Methods in Molecular Biology. Mycoplasma Protocols. New Jersey: Humana Press; 1998. p. 69-79.
8. Manual of Systematic Bacteriology Bergey. 2002. Available from: http://www.bergeys.org.
9. Manual de obtención del antígeno. Protocolos para la producción y estandarización de antígeno. Departamento de agricultura. Universidad de Georgia, Atlanta, USA. 2002.
10.Bashiruddin J. Extraction Mycoplasma Protocols. New Jersey: Humana of DNA from Mycoplasmas. In: Miles RJ, Nicholas RA, editors. Methods in Molecular Biology. Press; 1998. p. 141-144.
11.Fernández C, Chávez Y. Aplicación de la Reacción en Cadena de la Polimerasa para la detección de micoplasmas en cultivos celulares. Rev Salud Anim. 1999;18(1):31-34.
12.Agüero J. Estudios preliminaries para la amplificación de un PCR-Anidado a la detección de Mycoplasma gallisepticum. (Tesis). Centro Nacional de Sanidad Agropecuaria; 2002.
13.Lobo E. Presencia y patogenicidad de Mycoplasma pullorum en Cuba. (Tesis). Centro Nacional de Sanidad Agropecuaria; 2002.
14.Kong F, James G, Gordon S, Zelynski A, Gilbert G. Species- Specific PCR for identification of common
contaminant Mollicutes in cell culture. Appl Environ Microbiol. 2001;67(7):3195-3200.
15.Manual of Clinical Microbiology. 2003. Available from: http://www.mmbiologia.shtml
16.Harasawa R. Mycoplasma imitans a putative transposase gene in its 16S-23S rRNA intergenic spacer region. In: Abstract Book of 15th International Congress of the Internetional Organization of Mycoplasmology (IOM). July 11-16, Georgia, Athens; 2004. p. 137.
17.Mutangadura T, March J, Wiliamson H, Callcutt M. Distribution of ICEC in ¨Mycoplasma mycoides cluster mycoplasmas. In: Abstract Book of 15th International Congress of the Internetional Organization of Mycoplasmology (IOM). July 11-16, Georgia, Athens; 2004. p.145.
18.Nissen E, Vollenbroich D, Pauli G. Comparison of PCR detection methods for Mycoplasma in cell culture. In Vitro Cell Dev Biol Anim. 1998;32:463-464.
19.Uphoff C, Drexler H. Detection of Mycoplasma contaminations in cell cultures by PCR analysis. Human Cell. 1999;12(4):229-236.
20.Chaudhry R, Tabassum I, Nisar I, Dhawan B, Broor S, Khare S. Mycoplasma contamination in cell culture. In: Abstract Book of 15th International Congress of the Internetional Organization of Mycoplasmology (IOM). July 11-16, Georgia, Athens; 2004. p.88.
21.Barbeyrac B, Bébéar C, Taylor Robinson D. PCR: Preparation of DNA from clinical Specimens. In: Tully JG, Razin, S, editors. Molecular and Diagnostic Procedures in Mycoplasmology. Diagnostic Procedures. San Diego: Academic Press, 1996. p. 61-65.
22.Razin S. DNA probes and PCR in diagnosis of mycoplasma infections. Molecular and Cellular Probes. 1994;8:497-511.
23.Sachse K, Hotzel H. Classification of isolates by DNA-DNA Hybridization. In: Miles RJ, Nicholas RA, editors. Methods in Molecular Biology. Mycoplasma Protocols. New Jersey: Humana Press; 1998. p. 189-195.
24.Tang J, Hu M, Lee S, Roblin R. A polymerase chain reaction based method for detecting Mycoplasma/Acholeplasma contaminants in cell culture. J. Microbiol Methods. 2000;39:121-126.
(Recibido 2-1-2007; Aceptado 14-12-2008)
1 ADN de referencias, donadas gentilmente por el Dr. K.E Johansson, Jefe del Instituto Nacional de Veterinaria, Departamento de Bacteriología, Suecia.














