INTRODUCCIÓN
Las neoplasias mieloproliferativas crónicas (NMPC) constituyen un grupo de ocho entidades clínicas dentro de las cuales se incluyen las de fenotipo BCR-ABL negativas; estas se generan por un desorden clonal en las células madre hematopoyéticas, que conduce a una excesiva producción de células maduras, así como su acumulación en sangre periférica. Los linajes afectados en estos desórdenes son el eritroide, el megacariocítico y el granulocítico, los cuales generan policitemia vera (PV), trombocitemia esencial (TE) y mielofibrosis primaria (MFP), respectivamente. En la clasificación más reciente de la Organización Mundial de la Salud (OMS), esta última entidad se ha subdividido a su vez en estado prefibrótico y fibrótico. (1) A nivel mundial, la incidencia global de PV es de 0,84 por cada 100 000 habitantes con variaciones de 0,86 y 0,74 por cada 100 000 habitantes en Europa y Norteamérica, respectivamente. Por su parte, la TE tiene una incidencia global de 1,03 y 0,96 por cada 100 000 habitantes en estos dos continentes. Para la MFP la incidencia global es de 0,47 por cada 100 000 habitantes y no presenta diferencias marcadas entre regiones. Es preciso mencionar que muchos de los reportes han sido realizados en países desarrollados por lo que la incidencia de las NMPC en países subdesarrollados pudiera estar subestimada. (2) En Colombia, no existen datos epidemiológicos publicados para las NMPC. (3
El análisis genético y molecular de las NMPC ha sido de gran importancia en los últimos años al poner en evidencia la heterogeneidad en las bases genéticas de estas entidades. La primera alteración directamente relacionada con estas neoplasias fue la mutación en el gen de la Janus Kinasa 2 (JAK2) la cual tiene un efecto directo en la activación de la vía del JAK-STAT; esta mutación se detecta en el 90 % de los pacientes con PV, el 60 % con TE y el 65 % con MFP. (4
A partir del descubrimiento de mutaciones JAK2 en el año 2005, varios estudios estuvieron dirigidos en la búsqueda de otros genes responsables del desarrollo de NMPC en pacientes negativos para la mutación JAK2; fue así como en el año 2006, se encontró que una mutación somática en el receptor de trombopoyetina (MPL) también era responsable del inicio de la TE y la MFP en el 4-5 % de los casos. (5 La alteración molecular recientemente descrita en las NMPC es la mutación en el gen de calreticulina (CALR), esta fue identificada en el año 2013 por dos estudios simultáneos de Klampfl et al y Nangalia et al, por medio de secuenciación completa de exones y secuenciación dirigida de genes en un grupo de pacientes con NMPC negativos para las mutaciones en JAK2 y MPL; ambos estudios reportaron una frecuencia de mutaciones en CALR de aproximadamente 20-25 % en la TE y 25-30 % en la MFP. 6,7
La calreticulina es una proteína multifuncional de retículo endoplásmico altamente conservada, e involucrada en el control de calidad de las proteínas del retículo endoplásmico; las funciones de actividad como chaperona, la homeostasis del calcio y la apoptosis y muerte celular inmunogénica.
Dada la relación directa que existe entre las diferentes mutaciones en CALR con el desarrollo de las NMPC, así como su alta frecuencia, este gen se ha considerado como el segundo marcador molecular más importante después de JAK2 y, por lo tanto, en la actualización de la OMS del año 2016 se incluyó como criterio mayor para el diagnóstico de TE y MFP. (1 Por lo anterior, a nivel del laboratorio clínico se hace importante el conocimiento y aplicación de diferentes técnicas moleculares que permitan la detección de este marcador las cuales se tratarán con detalle en la presente revisión.
ASPECTOS ESTRUCTURALES Y MOLECULARES DEL CALR
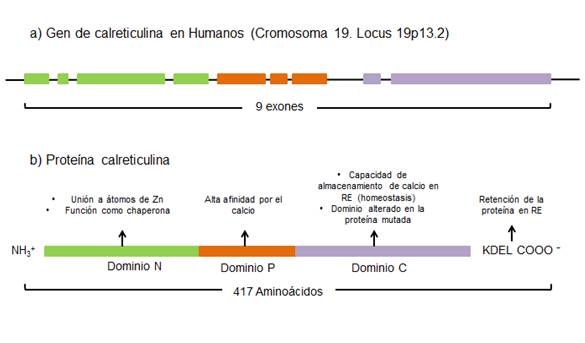
El gen CALR se encuentra ubicado en el brazo corto del cromosoma 19, (19p13.2); tiene una longitud de 5 891 pb y está formado por 9 exones. El ARN mensajero transcrito a partir del gen tiene una longitud de 1 929 pb y genera una proteína de 47 kDa (Fig. 1a). (8
La proteína calreticulina está formada por 417 aminoácidos que se dividen en 3 dominios principales: dominio N terminal, dominio P central y dominio C terminal. La unión a átomos de zinc así como la actividad de chaperona está dada por la presencia de un sitio de unión a lectina en el dominio N terminal; el dominio P central posee una alta afinidad por el calcio y, al igual que el dominio N, le otorga a la proteína funciones de chaperona, lo que le permite contribuir con el correcto plegamiento de proteínas y glicoproteínas recién sintetizadas; por otra parte, este dominio favorece la unión de calreticulina con la proteína de retículo endoplásmico 57 (Erp57). El dominio C terminal es crítico en el almacenamiento del calcio en el retículo endoplásmico, contribuyendo con su homeostasis. Por otra parte, la función como chaperona de calreticulina está estrechamente relacionada con la homeostasis del calcio, en la medida que contribuye con el correcto plegamiento del receptor de la bradiquinina, así como su unión a este; esta molécula induce liberación del calcio del retículo endoplásmico. Otras funciones reportadas para proteína calreticulina son el control de calidad de proteínas celulares, la apoptosis y muerte celular inmunogénica (Fig. 1b). (7,8
Además de los 3 dominios descritos, existe una cuarta región en el extremo C terminal denominada KDEL, formada por una pequeña secuencia de aminoácidos de lisina, ácido aspártico, ácido glutámico y leucina. KDEL constituye un dominio de retención en el retículo endoplásmico para la proteína calreticulina, que permite que después de su procesamiento en el aparato de Golgi, esta sea devuelta al retículo endoplásmico y no sea transportada fuera del citoplasma. En todas las proteínas calreticulina mutadas este dominio se encuentra ausente (Fig. 1b). (9,10
MUTACIONES Y SU UTILIDAD DIAGNOSTICA
Con el advenimiento de la biología molecular, para el año 2013 se estableció una gran influencia de los genes JAK2 y MPL en el desarrollo de estas enfermedades. En el gen JAK2 se describieron mutaciones como la V617F en la que el cambio de nucleótidos conduce a una sustitución de valina por fenilalanina en el codón 617 de la proteína y las del exón 12, descritas inicialmente en la PV. La alteración V617F es la más frecuente en las NMPC con un mayor porcentaje en la PV (95 - 96 %) y de 50 - 60 % en la TE y la MFP. (4,11) El descubrimiento de esta mutación, modificó los criterios de diagnóstico y pronóstico en las NMPC. Luego del descubrimiento de JAK2, se demostró la relación de otras mutaciones con las NMPC, una de estas fue la mutación somática en el receptor de trombopoyetina (MPLW515L), descrita en el 2006 como un evento patogénico importante en pacientes con MFP negativos para JAK2V617F.
Actualmente se conocen varias mutaciones para MPL y al igual que las de JAK2, desencadenan NMPC por activación constitutiva de la vía de señalización JAK-STAT que genera un aumento considerable en la producción de células maduras. 12
La identificación de CALR en el año 2013, fue un avance importante en el conocimiento de las neoplasias mieloproliferativas crónicas a nivel molecular. Cuando se relacionó, por primera vez, su presencia en estas entidades, fueron identificadas más de 30 mutaciones causadas en su gran mayoría por inserciones o deleciones en el exón 9; las frecuencias identificadas fueron muy altas, aproximadamente del 67 al 80 % de los pacientes negativos para mutaciones en JAK2, eran positivos para CALR. En estas alteraciones genéticas, diferentes modificaciones en la secuencia de nucleótidos ocasionan un desplazamiento en el marco de lectura del gen, esto genera la transcripción de un nuevo ARN mensajero alterado el cual es traducido en una proteína con modificaciones en la secuencia aminoacídica del dominio C terminal, que cambia su carga negativa por positiva, fundamentalmente. En la actualidad se conocen más de 50 mutaciones distintas para CALR. (6
Dentro de ellas hay dos variantes principales con mayor frecuencia de presentación, estas se han clasificado como mutación tipo I y tipo II. La mutación tipo I (c.1092_1143del) se genera por una inserción de 52 pb flanqueada por 7 pb de secuencia idéntica y la mutación tipo II (c.1154_1155insTTGTC) resulta de una inserción de 5 pb y representa una duplicación inversa de los 5 nucleótidos que preceden la inserción. (6,7 Estas dos mutaciones se encontraron en frecuencias muy similares en los estudios de Klampfl y Nangalia, con distribuciones de 53,0 % y 45 % para la tipo I y 31,7 % y 41 % para la tipo II, respectivamente; lo que demostró que la mutación tipo I es la de mayor frecuencia. (6,7
Es importante resaltar que diferentes tipos de mutaciones en el exón 9 de CALR fueron detectadas no solo en NMPC, sino también en otros desórdenes hematológicos como: leucemia mieloide aguda y crónica, leucemia mielomonocítica crónica, síndromes mielodisplásicos, anemia refractaria con sideroblastos en anillo asociada con trombocitosis (RARS-T) y anemia refractaria con exceso de blastos. En estas entidades, las mutaciones se identificaron en menor frecuencia. Por el contrario, en neoplasias linfoides y en tumores sólidos, estas mutaciones estuvieron ausentes. (6, 7
El efecto a nivel funcional que generan estas mutaciones fue evidenciado in vitro en células que presentaron la mutación tipo I. Esta condición genera una proliferación celular independiente de citoquinas, la cual se inhibió al tratar tanto las células mutadas como las no mutadas con inhibidores de la JAK2. Este hallazgo sugiere una participación de la vía JAK-STAT en la proliferación celular inducida por CALR. Así mismo se evidenció un incremento en la fosforilación de STAT5 en las células mutadas para CALR en ausencia de interleuquina 3. In vivo se ha demostrado gran efectividad terapéutica a inhibidores de JAK2 en pacientes con NMPC positivos para CALR. 6
Al igual que las otras dos mutaciones conductoras o “drivers”, antes mencionadas, diferentes estudios a nivel clínico han evidenciado que la presencia de mutaciones en CALR se asocia con condiciones clínicas características que tienen implicaciones importantes en la evaluación del pronóstico y curso de la enfermedad de cada paciente. Desde su primera descripción en 2013, los estudios de Nangalia y Klampfl demostraron que los pacientes con TE positivos para CALR, cursaban con menores niveles de hemoglobina, hematocrito y glóbulos blancos, así como con un mayor recuento de plaquetas respecto a los pacientes con mutaciones en JAK2. Del mismo modo, los pacientes con MFP CALR positiva, tenían menores recuentos de leucocitos y mayores recuentos de plaquetas comparados con pacientes JAK2 positivos. (5-7 En diversos estudios realizados con el objetivo de determinar diferencias a nivel clínico de las mutaciones conductoras, se han reportado las mismas características hematológicas para TE con CALR mutado; estos resultados han demostrado que el fenotipo de CALR mutado se asocia con una expansión preferencial del linaje megacariocítico comparado con la expansión eritroide que se manifiesta en NMPC con mutaciones en JAK2. 13-14
Es importante mencionar que se ha relacionado la presencia de mutaciones en CALR con una menor incidencia de trombosis en comparación con JAK2 y MPL con porcentajes de 1,6 %, 14,54 % y 19,46 %, respectivamente. Así mismo, los eventos cardiovasculares ocurren en un menor porcentaje en pacientes con TE CALR positivos. La anterior asociación parecería contradictoria si se tiene en cuenta que CALR se relaciona con mayores recuentos de plaquetas, pero se cree que el evento trombótico ocurre más por la activación de plaquetas y leucocitos, y un alto recuento de leucocitos; que por el aumento único de plaquetas. 14-16
En cuanto a la transformación a MFP posterior a TE, la presencia de la mutación en CALR favorece este evento, comparado con una menor tendencia de transformación en pacientes negativos para CALR. (7,13,17 En pacientes con MFP, la presencia de mutaciones CALR se asocia con menores porcentajes de transformación leucémica en comparación con genotipos JAK2, MPL o triple negativos. (18
Los diferentes estudios clínicos han demostrado que las NMPC CALR positivas tienen un curso clínico más favorable que las JAK2 o MPL positivas y por lo tanto evidencian la gran importancia que tiene la caracterización molecular de esta mutación. La detección de las mutaciones en CALR constituye una herramienta diagnóstica de gran impacto en la medida que orienta no solo el manejo clínico de las NMPC, sino también su curso y progresión.
TECNICAS PARA LA DETECCIÓN DE CALR
La alta frecuencia de presentación de las mutaciones en el gen CALR en NMPC, así como sus implicaciones a nivel clínico; ha puesto en evidencia que su detección constituye una práctica fundamental en los laboratorios clínicos tanto para el diagnóstico como para la predicción de riesgo de progresión de la enfermedad y respuesta al tratamiento. Por esta razón, muchos investigadores se han interesado en el desarrollo y aplicación de técnicas cada vez más sensibles, específicas y costo-efectivas que faciliten el diagnóstico preciso del gran porcentaje de pacientes con NMPC negativos para JAK2, por medio de la detección de CALR.
La primera metodología aplicada por los investigadores Nangalia y Klampfl en el año 2013, fue la secuenciación completa del gen y la secuenciación a nivel de exones, esta técnica ofrece alta especificidad ya que permite identificar de manera precisa el tipo de mutación, además ha sido reconocida como el estándar de oro para la detección de CALR.6,7 Dado que la secuenciación constituye un método costoso y de alta demanda de tiempo, varios autores se vieron en la necesidad del desarrollo de nuevas técnicas mucho más eficientes para la detección de estas mutaciones.
En el 2014, Andrikovics y cols., implementaron la técnica de análisis de fragmentos de alta resolución por medio de electroforesis capilar para indicar la presencia o ausencia de las mutaciones en CALR. Esta se basó en la detección de cambios en la longitud de las secuencias de ADN obtenidas por reacción en cadena de la polimerasa (PCR). En este estudio, las muestras identificadas como positivas para CALR por medio del análisis de fragmentos, fueron analizadas con la secuenciación de Sanger para confirmar el tipo de mutación específico. De 121 pacientes analizados, encontraron 53 % con mutación tipo I, 28 % con mutación tipo II y 23 % con otros tipos de mutaciones. La implementación de esta técnica demostró gran utilidad dada su alta sensibilidad y especificidad. (6,19)
Debido a la simplicidad y sensibilidad de los análisis de fusión de alta resolución (HRM) para identificación de diferentes alteraciones genéticas, esta técnica ha sido ampliamente introducida en el laboratorio con diferentes fines diagnósticos. Para la detección de CALR, varios autores han evaluado la aplicación de esta metodología. El estudio de Bilbao y cols., en 2014, con 153 pacientes con TE y trombocitosis recurrentes, negativos para MPL y JAK2, mostró que el método HRM es útil para la detección de las mutaciones en el exón 9 de CALR así como para la clara discriminación entre los pacientes positivos y los negativos.20 La confirmación de estas alteraciones se realizó por la metodología de secuenciación, no se detectaron falsos positivos o negativos; de este modo se demostró gran reproducibilidad de los análisis de fusión para la detección de CALR. Con este estudio se demostró que el HRM es un método sensible, fiable y rápido para seleccionar con precisión los pacientes positivos para CALR; sin embargo, la caracterización del tipo de mutación requiere la aplicación de métodos más específicos como secuenciación directa o análisis de fragmentos. 20 Al igual que Bilbao, Lim KH y cols., demostraron que el análisis HRM es una técnica de tamizaje, rápida y sensible para la detección de mutaciones CALR. 21
En ese mismo año (2014), Chi J y cols. desarrollaron un nuevo algoritmo, tanto para la caracterización inicial como para la identificación específica y cuantificación de las mutaciones de CALR, a través del uso combinado de HRM y PCR en tiempo real (Q-PCR), respectivamente. 22 Para el análisis HRM, diseñaron cebadores específicos que amplifican el exón 9 de CALR y generan un fragmento de 298 pb; con esta técnica demostraron que todas las muestras positivas para la mutación (ocho tipos diferentes) tenían curvas de fusión características que diferían notablemente de las negativas. Además, evidenciaron la asociación de las curvas de fusión en tres grupos diferentes que se correlacionaban con una deleción de 52 pb (mutación tipo I), una inserción de 5 pb (mutación tipo II) y tipos silvestres. Posterior al tamizaje de las mutaciones, estos autores se interesaron en implementar una alternativa rápida y eficiente para diferenciar específicamente las dos mutaciones más frecuentes en CALR (tipo I y II); para este fin desarrollaron una Q-PCR con cebadores específicos que se unen exclusivamente a las secuencias de interés, esta técnica demostró gran especificidad para la caracterización de las dos variantes. Por otra parte, en el estudio se evidenció que la técnica posee alta sensibilidad para detectar las mutaciones en una carga alélica de 1- 2 %, siendo mayor que la sensibilidad reportada para el análisis por electroforesis capilar y de análisis de fragmentos. Este estudio ofrece un algoritmo rápido, sencillo y útil para la cuantificación y seguimiento de la enfermedad el cual, comparado con la secuenciación Sanger, permite la confirmación del tipo de mutación de una manera mucho más simple. 22 Al igual que Chi J y cols, otros autores han demostrado que la Q-PCR es una técnica sensible y específica, útil en la detección de mutaciones en CALR. 23
En el estudio realizado por Yao QM y cols. en el año 2015, en 152 pacientes diagnosticados con TE y MFP negativos para JAK2, se implementó como técnicas para la detección de CALR la amplificación por PCR, seguida del análisis de longitud de fragmentos con confirmación por secuenciación directa. También realizaron una estimación de la carga alélica con experimentos de dilución. (24 Con la combinación de estas metodologías, fue posible la identificación de un gran número de mutaciones para CALR, a excepción de aquellas delecciones de 1pb; a diferencia del algoritmo propuesto por Chi J, en el cual solo es posible la detección de los dos tipos principales de mutaciones, este estudio provee una mayor especificidad. El análisis de fragmentos desarrollado en este estudio y en estudios de otros investigadores, constituye una técnica rápida altamente sensible y específica para el tamizaje y caracterización de mutaciones de CALR y, potencialmente útil para la detección de niveles bajos de alelo mutado, lo que le da aplicabilidad en el monitoreo de la respuesta al tratamiento. A diferencia de las demás técnicas, es ampliamente adaptable para la identificación de un gran número de cambios en el gen. (24-28
Recientemente, una nueva técnica para la detección de mutaciones tipo I y II fue implementada por Rosso y cols. Esta se basó en la metodología de bloqueo de la PCR mediado por péptidos de ácidos nucleicos para discriminar secuencias de tipo silvestre de las secuencias mutadas. (29 El principio de este método es la competencia de unión para un sitio común en el ADN entre un polipéptido de ácido nucleico (PNA) complementario a la secuencia de tipo salvaje y un cebador de la PCR que es complementario para la secuencia mutada, o viceversa. La unión del PNA al ADN domina sobre la unión del cebador debido a su mayor afinidad por el sitio; así mismo la unión del PNA impide la amplificación del ADN. En este estudio, la secuencia PNA fue diseñada para unirse al ADN de pacientes negativos para la mutación y al de los positivos para la mutación tipo I. Por lo tanto, en estos dos casos, no era posible la amplificación por PCR produciendo bandas débiles de diferentes tamaños. En los pacientes positivos para la mutación tipo II, el cebador era específico para la secuencia de nucleótidos por lo que se favoreció la amplificación por PCR. Con esta nueva metodología se demostró gran eficiencia, sensibilidad y especificidad en la detección de CALR y al igual que los demás métodos probados, este tipo de PCR fue menos costosa y más rápida, comparada con la secuenciación de Sanger. Una desventaja atribuible a esta técnica es que únicamente predice las mutaciones para las cuales se hayan diseñado los cebadores. 29-30
Con el fin de realizar una evaluación comparativa de los principales métodos moleculares descritos, en 2015, Jones y cols. realizaron diluciones de ADN mutado con ADN silvestre para evaluar la carga alélica mínima detectada por cada una de estas metodologías. Concluyeron que entre las técnicas de HRM, análisis de fragmentos y secuenciación de Sanger; las dos primeras son más sensibles con niveles de detección de 5 % y 5 - 10 %, respectivamente; comparados con la secuenciación de Sanger que detecta la mutación solo en niveles altos de 10-25 %.31 Sin embargo, la secuenciación de última generación (NGS, por sus siglas en inglés), tuvo el menor límite de detección con una carga alélica de 1,25 %, siendo la técnica más sensible para el diagnóstico de estas mutaciones.31 En este método, se realiza amplificación por PCR mediante el uso de cebadores que forman apareamiento con secuencias conectoras unidas a un sustrato sólido; tras varios ciclos de PCR, se producen grupos del gen de interés que se secuencian fácilmente mediante el uso de nucleótidos marcados con fluorescencia. A pesar de que los estudios en los cuales se aplica esta técnica son escasos, algunos autores han comprobado su alta sensibilidad para detectar cargas alélicas desde 0,2 %. 32
En 2015, otro estudio de comparación de métodos moleculares para la detección de CALR fue reportado por Park JH. Al igual que en los anteriores, se comprobó que el análisis de fragmentos es el más sensible seguido de HMR y secuenciación de Sanger, con porcentajes de sensibilidad de 98,2 %, 96,4 % y 89,3 %, respectivamente; la especificidad reportada para estos tres métodos fue de 100 %, 96,3 % y 100 %, respectivamente (Tabla 1). Los resultados sugieren que en la práctica del laboratorio de rutina, el análisis de fragmentos tiene un desempeño similar al análisis de fusión por lo que ambos pueden ser utilizados como métodos de detección, de tamizaje de primera línea y además constituyen métodos fáciles y rápidos; en caso de que se obtengan resultados positivos, la secuenciación puede ser utilizada para confirmar y definir el tipo específico de mutación. 33
Clásicamente, todos los métodos diseñados para la identificación de CALR, se enfocan en la detección a nivel molecular de las diferentes mutaciones; en 2016, Stein H y cols. diseñaron una nueva técnica basada en la caracterización de mutaciones en CALR a nivel proteico, más que genómico. En este caso, desarrollaron un anticuerpo monoclonal (CAL2) que reconoce específicamente la región C terminal alterada que se genera en todos los tipos de mutaciones del gen. (34 Luego de la obtención del anticuerpo en modelos murinos, los investigadores desarrollaron una coloración inmunohistoquímica para la identificación directa de las mutaciones CALR en biopsias de medula ósea, la efectividad de este método fue comparada con secuenciación de Sanger y mostró 100 % de correlación en 52 pacientes positivos. La coloración CAL2 fue probada como un método sensible, rápido, simple y específico. (34) En ese mismo año, Nomani y cols. probaron igualmente una alta correlación de la técnica en 8 pacientes positivos para CALR todos con tinciones positivas. (35
Tabla 1 Principales técnicas usadas para la detección de mutaciones en CALR
| Técnica | Descripción de la técnica | Sensibilidad (%) | Especificidad (%) |
|---|---|---|---|
| Secuenciación de Sanger | Técnica basada en la amplificación de ADN mediante el uso de nucleótidos marcados con fluorocromos que determinan la secuencia exacta del ADN | 89,30* | 100* |
| Análisis de fragmentos | En esta técnica se combina la amplificación por PCR con electroforesis capilar de alta resolución para detectar pequeñas diferencias en la longitud de los productos de PCR | 98,20* | 100* |
| Análisis de fusión de alta resolución (HRM) | Las alteraciones de la secuencia se detectan mediante cambio en el perfil de fusión del ADN que depende del contenido de nucleótidos, la longitud y la heterocigocidad del producto de PCR | 96,40* | 96.30* |
| Secuenciación de última generación | Se realiza amplificación por PCR mediante el uso de cebadores que forman apareamiento con secuencias conectoras unidas a un sustrato sólido; generando grupos del gen de interés que se secuencian fácilmente mediante el uso de nucleótidos marcados con fluorescencia | 98 - 99** | 100** |
* Tomado de: Park et al. PLoS One. 2015;10(10):1-7 33)
** Tomado de: Jones et al. Leuk Res. 2015;39(1):82-7 31
CONSIDERACIONES FINALES Y PERSPECTIVAS
Hasta el año 2006, el diagnóstico de las NMPC se centró principalmente en signos clínicos y características hematológicas de los pacientes en sangre periférica y en medula ósea; con el advenimiento de la biología molecular, en los últimos años, ha sido posible la identificación de diferentes marcadores genéticos de gran utilidad para el diagnóstico preciso y rápido de estas neoplasias, los principales marcadores según la frecuencia de presentación son JAK2, CALR y MPL.
El conocimiento de estas mutaciones ha permitido una mayor comprensión de las características moleculares de las NMPC. Con esta revisión se evidencia que las mutaciones en el gen CALR, constituyen nuevos marcadores de gran importancia, los cuales deberían incluirse en la práctica médica como parte de los algoritmos diagnósticos.
Por otra parte, a través de la recopilación de información de las diferentes técnicas moleculares implementadas para la detección de CALR, se puede concluir que después de la NGS, las metodologías de análisis de fragmentos y análisis de fusión, constituyen opciones útiles que pueden ser aplicables en nuestro medio para la identificación de los diferentes tipos de mutaciones puesto que presentan altos porcentajes de sensibilidad, especificidad y eficiencia; además, su implementación representa menores costos en comparación con la secuenciación. Finalmente, es preciso destacar que la detección de mutaciones en CALR en pacientes con NMPC representa un avance importante no solo a nivel diagnóstico sino también a nivel pronóstico Por lo anterior, se destaca la necesidad de la inclusión de este marcador como técnica de rutina en los laboratorios de diagnóstico.