Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión On-line ISSN 2224-4697
Rev. Protección Veg. vol.30 no.3 La Habana sep.-dic. 2015
ARTÍCULO ORIGINAL
Organismos asociados a la pudrición del cogollo de la palma aceitera (Elaeis guineensis Jacq) en San Lorenzo, Ecuador
Organisms associated with the oil palm (Elaeis guineensis Jacq) bud rot in San Lorenzo, Ecuador
Fernando Rivas FigueroaI*, Lidcay Herrera IslaII
IFacultad de Recursos Naturales, Escuela Superior Politécnica de Chimborazo, Riobamba, Código postal 060150, Ecuador.
IIFacultad de Ciencias Agropecuarias, Universidad Central Marta Abreu de Las Villas, Santa Clara, Código postal 50400. Cuba.
RESUMEN
El presente trabajo se realizó con el objetivo de aislar los microorganismos asociados a la enfermedad Pudrición del Cogollo de la palma aceitera. Para ello, se seleccionaron plantas con manifestaciones de los síntomas característicos de la enfermedad y se tomaron fragmentos de los tejidos afectados. Las muestras se lavaron en agua corriente durante 15 minutos, luego se desinfectaron con hipoclorito de sodio al 2% (v/v) por 5 minutos y, finalmente, se lavaron con agua destilada estéril. Las secciones de tejidos se secaron en papel filtro estéril en cámara de flujo laminar y se colocaron en medio Agar Agua estéril al 2% y se incubaron a 25oC± 1. Las estructuras observadas se sembraron en medios estériles PDA 3,8% y Agar Nutriente 2,8% para aislar los hongos y las bacterias, respectivamente. Los resultados mostraron a Fusarium como el género asociado más prevalente, principalmente las especies Fusarium oxysporum Schlechtendahl emend. Snyder & Hansen, Fusarium proliferatum (Matsushima) Nirenberg y Fusarium solani (Martius) Appel & Wollenweber emend. Snyder & Hansen. Además, se aislaron Chalaropsis sp., Diplodia sp. y aislados bacterianos pertenecientes al género Erwinia.
Palabras clave: Chalaropsis, Diplodia, Erwinia, Fusarium, pudrición del cogollo.
ABSTRACT
The present work was aimed to isolate microorganisms associated with the bud rot disease in oil palm. Samples of infected tissues were taken from plants with typical symptoms of the disease. They were washed with tap water for 15 minutes, disinfected with a 2% sodium hypochlorite solution for 5 minutes, and finally washed with sterile distilled water. Tissue fragments were blot-dried on sterile filter paper in a laminar flow cabinet, placed on sterile 2% water agar, and incubated at 25oC ± 1 for 2 to 7 days. The structures observed were transferred to sterile PDA (3.8%) and Nutrient Agar (2.8%) to isolate fungi and bacteria, respectively. Fusarium was the prevalent fungal genus, and the main species identified were Fusarium oxysporum Schlechtendahl emend. Snyder & Hansen, Fusarium proliferatum (Matsushima) Nirenberg, and Fusarium solani (Martius) Appel & Wollenweber emend. Snyder & Hansen. Besides, Chalaropsis sp., Diplodia sp., and bacterial isolates belonging to the genus Erwinia were also identified.
Key words: Chalaropsis, Diplodia, Erwinia, Fusarium, bud rot.
INTRODUCCIÓN
En Ecuador, el cultivo de la palma aceitera tiene gran importancia económica en la producción agrícola; la superficie sembrada de esta especie es de 280 000 hectáreas (1). Dentro de las enfermedades que afectan la producción se encuentra una grave enfermedad conocida como pudrición del cogollo, PC o complejo PC, que ha provocado daños de relevante consideración económica al destruir plantaciones enteras en varios países de América Latina, tales como Panamá, Colombia, Surinam, Ecuador y Brasil (2, 3).
En la Provincia Esmeraldas, en la zona de San Lorenzo, la enfermedad PC ha provocado, en los últimos seis años, pérdidas de más de 4000 hectáreas; lo cual representa una seria amenaza para la producción palmera en San Lorenzo, Eloy Alfaro, la Concordia y Quinindé (4).
La sintomatología de la enfermedad se presenta como clorosis y amarillamiento en las hojas jóvenes, necrosis de los foliolos de la base hacia la parte distal, pudrición descendente del paquete de flechas, colapso y doblamiento de las hojas jóvenes. La pudrición del cogollo puede mostrar un color anaranjado-amarillento o café-rojizo, que se extiende hacia abajo hasta alcanzar el punto de crecimiento y causar la muerte de la palma (5, 6).
La primera investigación detallada y publicada sobre la PC se realizó en las plantaciones de Lever Brothers en el sur este de la República Democrática del Congo (Zaire). Después de trabajos de campo y laboratorio se identificó a la bacteria Erwinia lathyri como el organismo causal, a pesar que puede ser aislada también de plantas sin síntomas (7).
Años más tarde, investigadores colombianos informaron la presencia de varias especies de Fusarium, principalmente Fusarium solani (8) y un complejo fúngico que comprende Fusarium sp., Pythium sp. y Thielaviopsis paradoxa como los agentes asociados a la enfermedad PC (9), en el cual se resalta el papel de T. paradoxa en la patogénesis (10).
En otros estudios realizados en la Zona Oriental de Colombia se han encontrado diferentes microorganismos, entre ellos el hongo T. paradoxa, varias especies de Fusarium, Phytophthora, Pythium y bacterias del género Erwinia, pero no hay evidencias claras de que alguno, o algunos de ellos, sean los responsables de la enfermedad, ya que no ha sido posible reproducirla (11).
Sobre la base de la problemática anterior, el presente estudio tuvo como objetivo aislar e identificar los organismos asociados a la pudrición del cogollo en las plantaciones palmeras de la zona de San Lorenzo, provincia Esmeralda, Ecuador.
MATERIALES Y MÉTODOS
Recolección de muestras de plantas enfermas
En los meses de octubre y diciembre de 2012 se recolectaron muestras de tejidos afectados por la pudrición de cogollo en plantaciones de cinco empresas palmicultoras y plantas de vivero enfermas del híbrido Coarí x La Mé y Elaeis guineensis, de las Empresas Palmeras de Los Andes y Alespalma (Fig.1).
Las palmas enfermas se cortaron transversalmente en la base del tallo, mediante el empleo de una motosierra. Posteriormente, se retiraron todas las hojas del estipe dejando el paquete central de flechas. Luego, se realizó un nuevo corte transversal en el estipe a la altura del último racimo de fruta y, por último, se practicó un corte longitudinal a nivel del cogollo de la planta. Se colectó material de tejido sano y enfermo de la zona de cogollo, la flecha y las raíces. Se colocóó el material en papel kraft con su debida identificación y se transportó en nevera portátil al Laboratorio de Sanidad Vegetal de la Escuela Superior Politécnica de Chimborazo (ESPOCH).
Aislamiento de organismos asociados a la PC
El aislamiento de microorganismos de tejido enfermo, de un primer muestreo, se realizó en el Laboratorio de la Empresa Alespalma y en el Laboratorio de Sanidad Vegetal de la ESPOCH; se efectuó el aislamiento de los organismos asociados de las plantas enfermas de vivero del material híbrido Coarí x La Mé y Elaeis guineensis, así como de tejidos enfermos de un segundo muestreo en las plantaciones comerciales de las empresas palmicultoras seleccionadas.
A nivel de laboratorio se practicaron cortes en las muestras de tejidos enfermos en pequeñas secciones, las cuales se lavaron en agua corriente por 15 minutos; luego, se desinfectaron en hipoclorito de sodio al 2% por 5 minutos; posteriormente, se lavaron con agua destilada estéril. Bajo la cámara de flujo laminar, las secciones de las muestras en estudio se secaron en papel filtro estéril y se practicaron pequeños cortes de 5 a 8 mm en las secciones de tejidos enfermos. Los cortes se sembraron inicialmente en medio de cultivo estéril agar-agua al 2% para promover el desarrollo microbiano y se incubaron las placas Petri a 25oC ± 1 durante 2 a 7 días. Las floras microbiana fúngica y bacteriana presentes se transfirieron a los medios estériles PDA al 3,8% y Agar Nutritivo al 2,8%, respectivamente, y se incubaron a 25o C±1 durante 7 días. Posteriormente, se hicieron reaislamientos de los agentes microbianos fúngicos y bacterianos para obtener cultivos puros (12). Los cultivos puros se registraron y se almacenaron en placas Petri debidamente selladas en gabinetes a temperatura ambiente.
Identificación morfológica-cultural de los aislamientos fúngicos
Para la identificación de los aislados fúngicos se tomaron en consideración las características culturales en medio Papa Dextrosa Agar (coloración del medio de cultivo) y las características de las estructuras reproductivas, utilizando claves taxonómicas morfológicas establecidas para el efecto (13, 14).
Identificación molecular de los agentes fúngicos asociados a la enfermedad
Para los análisis moleculares se tomaron en consideración los géneros fúngicos que aparecieron con mayor frecuencia en las muestras analizadas del híbrido Coarí x La Mé. El ADN de cada aislamiento se obtuvo a partir de cultivos monospóricos. Para ello, se tomó un disco de micelio de 0.5 cm de diámetro de los aislados, provenientes de Placas Petri con 10 ml de medio PDA, donde los aislados crecieron durante 15 días. Los discos se colocaron en Erlenmeyers (250 ml de capacidad) que contenían 100 ml del medio de cultivo líquido B5 (15, 16), suplementado con sacarosa 2% (17). Los mismos se colocaron en agitación (agitador orbital GFL) a 120 rpm y temperatura de 25±10C durante tres semanas. Transcurrido este periodo, se colectó el micelio por filtración con un filtro millipore (45µm) y se conservó en congelación (-20o C) hasta su utilización. La extracción del ADN genómico total se realizó usando el kit de extracción Trizol DNA (Sigma-Aldrich Co., Mo, USA) de acuerdo a las instrucciones del fabricante (Invitrogen) y se conservó el ADN extraído en 50 µl del tampón TE. Las concentraciones de ADN se calcularon por la medición de la absorbancia en la longitud de onda de 260 nm en un espectrofotómetro (Ultrospec Plus Spectrophotometer Pharmacia, LKB), según lo referido (18).
Se amplificó la región ITS del ADN utilizando los primers ITS1-F (5_-CTT GGT CAT TTA GAG GAA GTA A-3) y ITS4 (5_-TCCTCC GCT TAT TGA TAT GC-3), acorde con lo sugerido por White et al. (19). La mezcla de reacción de PCR empleada tuvo un volumen final de 25 µL, estuvo compuesta por Tampón PCR 10 X (tris-HCl 10mM, KCl 50mM., MgCl2 1,5mM), dNTPs 200uM, 100ng de ADN, cebadores ITS4f e ITS4r 10µM de cada uno y 1U de Taq polimerasa (Promega, Madison, WI, USA).
Las amplificaciones se ejecutaron en un termociclador (PTC-100; M J Research, Inc) que desarrolló el siguiente programa: un paso de desnaturalización inicial a 94oC (30 segundos), hibridación a 55oC (30 segundos) y extensión a 72oC (1 minuto); estos pasos se repitieron en 35 ciclos sucesivos y se efectuó un paso de extensión final a 72oC por 7 minutos (20).
Los productos del PCR se purificaron al usar el Kit QIAquick® kit (QIAGEN, Germany), para ello se siguieron las instrucciones del fabricante y se secuenciaron en ambas direcciones en un Secuenciador de DNA, ABI prisma automatizado (modelo 377, versión 2.1.1; Applied Biosystems Warrington, United Kidngdom) con cebadores ITS4f e ITS4r.
Para el análisis de las secuencias de las ITS obtenidas se realizó la comparación con secuencias archivadas en GenBank usando análisis tipo BLAST.
Identificación morfológica y bioquímica de bacterias
La identificación de los aislamientos bacterianos seleccionados se ejecutó en el Laboratorio de Microbiología de la Facultad de Ciencias Agropecuarias de la Universidad Central «Marta Abreu» de Las Villas en Cuba, para lo cual se siguieron los procedimientos morfológicos y bioquímicos descritos (21). Los aislados bacterianos se caracterizaron bioquímicamente sobre la de los siguientes aspectos: crecimiento a 36ºC, producción de ácido a partir de lactosa, producción de indol, prueba oxidativa-fermentativa (O/F), producción de pigmentos fluorescentes en medio King B, hidrólisis de gelatina y pudrición de rodajas de papa, según lo referido por Lelliott y Stead (21). Las colonias bacterianas inoculadas en los distintos medios se incubaron a 28o C por 72 horas. Además, se probó la reducción de la sacarosa con el reactivo de Benedict; para esto, en los tubos de ensayo que contenían medio de cultivo con las diferentes cepas bacterianas puras, se les adicionaron 5 ml del reactivo a cada uno de los tubos y se comparó con un control experimental. Luego de colocar los tubos en vaso de precipitación con agua caliente en baño María por 10 minutos, se observó y se registró la reacción de cada cepa.
RESULTADOS Y DISCUSIÓN
Organismos aislados asociados al complejo PC
Los microorganismos aislados asociados al complejo PC ascendieron a 63 aislamientos fúngicos, de los cuales el 85,71 % correspondió al género Fusarium spp.; 6,34% al género Trichoderma sp.; 1,58% al género Diplodia sp.; 3,17% al género Chalaropsis sp; 1,58% al género Colletotrichum sp. y 1,58% al género Pestalotiopsis sp. (Tabla 1). Además, se obtuvieron 27 aislados bacterianos.
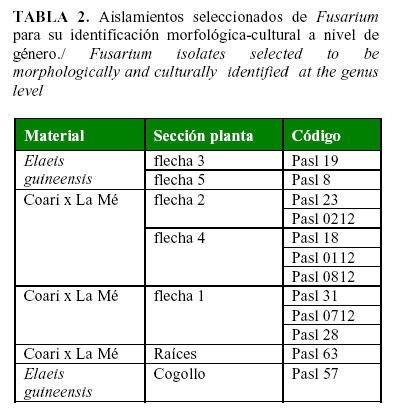
A partir del resultado anterior se seleccionaron 12 aislamientos correspondientes al género Fusarium spp., para la identificación de especie (Tabla 2).
Identificación morfológica cultural de aislamientos fúngicos de Fusarium spp.
Las características morfológicas culturales de los 12 aislamientos del género Fusarium spp., se muestran en la Tabla 3. De ellos, 33.33% correspondió a Fusarium proliferatum, el 50 % a Fusarium oxysporum y 16,67 % a Fusarium solani (Tabla 4; Fig. 2, 3 y 4).
Identificación molecular de 2 aislados del género Fusarium asociados al complejo PC
Las secuencias obtenidas a partir de la amplificación de las regiones ITS permitieron confirmar los 2 agentes del género Fusarium asociados a la enfermedad. Para el caso del aislamiento PASL0712 se identificó como F. proliferatum, con el número de acceso al Gen Bank KJ 025005.1, mostró un porcentaje de homología del 99% y, para el caso del aislamiento PASL 0112, se determinó como F. oxysporum, con el número de acceso del Gen Bank KF 562122.1 mostró un porcentaje de homología de 99%.
Identificación morfológica y bioquímica de bacterias
De un total de 27 aislamiento bacterianos, obtenidos a partir de muestras de plantas en vivero y campo, atacadas por el complejo PC en las diversas empresas palmicultoras, se seleccionaron 7 aislamientos, teniendo en cuenta la reacción de Gram; sus características culturales se muestran en la Tabla 5.
Las pruebas morfofisiológicas demostraron la presencia de bacterias Gram negativas y de morfología bacilar típica del género Erwinia sp. (Fig. 5, Tabla 6).
Del género Fusarium prevalecieron las especies F. oxysporum, F. proliferatum y F. solani, mientras que fue menor la presencia Chalaropis sp. y Diplodia sp., así como las cepas bacterianas del género Erwinia. Estos resultados coinciden con los referidos en otras investigaciones efectuadas en Colombia, el Congo y Ecuador (11, 6).
La presencia de la especie F. proliferatum, dentro del complejo de la PC de los agentes asociados en la zona de San Lorenzo, constituye el primer informe para San Lorenzo, Provincia Esmeraldas. Además, no se detectó la presencia de Phythopthora spp. asociadas con esta enfermedad, lo cual concuerda con lo obtenido por otros autores (6). La presencia de aislados de Erwinia spp. sugiere que esta bacteria puede contribuir con las fases finales del proceso de pudrición del cogollo de la palma aceitera.
AGRADECIMIENTOS
Presentamos nuestros agradecimientos a las empresas Palmeras de Los Andes, Palesema, Energy & Palma, Alespalma y Paipailón establecidas en San Lorenzo y a la Asociación Nacional de Cultivadores de Palma Aceitera (ANCUPA) por su comprometida colaboración con la investigación de campo. Igualmente, agradecemos a la Universidad Central «Marta Abreu» de Las Villas en Cuba y a la Universidad de Sassari en Italia y a la Agencia Ecuatoriana de Aseguramiento de la Calidad del agro, por apoyarnos en actividades de investigación a nivel de laboratorio en la identificación de los organismos asociados con la pudrición de cogollo en la palma aceitera.
REFERENCIAS
1. Ancupa. Ecuador País palmicultor. Revista Palma. Edición Especial. 2010;23:34-42.
2. De Franqueville H. Oil Palm Bud Rot in Latin America. Cambridge University Press. 2003;39:225-240.
3. Santos AE. Análisis de las últimas investigaciones sobre pudrición del cogollo en palma de aceite (Elaeis guineensis Jacq). Trabajo de grado para optar al título de especialista en cultivos perennes industriales, Bogotá, Colombia. 2010;39p.
4. El cogollo mantiene en ajetreo a los palmicultores ecuatorianos. Consultado en agosto de 2011. http://www.hoy.com.ec.2011.
5. Laing D. Causa de la pudrición de cogollo en la palma de aceite papel del calcio en una hipótesis abiótica-edáfica. XII Congreso Ecuatoriano de la Ciencia del Suelo. Santo Domingo, Ecuador. 17 -19 Noviembre 2010: 24.
6. Ronquillo M, Estévez de Jensen C, BernalG. Fusarium spp. asociados a la pudrición del cogollo de la palma aceitera (Elaeis guineensis Jacq) en Ecuador. J Agric Univ P.R. 2013;97(3-4):135-148.
7. Duff ADS. The but rot/Little leaf disease of the oil palm. Journal of Waifor. 1963;4(14):176-190.
8. Chinchilla C. Las pudriciones de cogollo en palma aceitera: La complejidad del desorden y una guía de convivencia ASD. Costa Rica (citado 2012, ago 18). Disponible en: http://www.asd-cr.com/paginas/español/articulos/Guia-PC.html.; 2010.
9. Nieto PLE. Síntomas e identificación del agente causal del complejo pudrición del cogollo de palma de aceite, Elaeis guineensis Jacq. Revista Palmas (Colombia). 1996;71(2):57-60.
10.Gómez PL, Ayala L, Munévar F. Characteristics and management of Bud rot, a disease of oil palm. Proc Of the International Planters Conference, 17-20 May 2000: 545-553.
11.Sarria GA, Torres GA, Aya HA, Ariza JG, Rodríguez J, Vélez DC. Microorganismos asociados a la Pudrición del cogollo de la Palma de aceite y su inoculación en palmas de vivero. Edición Especial, Palmas. 2008;29(3):27.
12.Agrios GN. Plant Pathology. Deparment of Plan Phatology University of Florida. 5th Edition. Elsivier Academia Press, USA. 2005. P. 921.
13.Gerlach W, Nireemberg H. Institute Fur Mikrobiologie, Berlin-Dahlem. The genus Fusarium a Pictorial Atlas. Berlim. 1982: 386.
14.Barnett HL, Hunter BB. Ilustrated genera of imperfect fungi. ASP Press. 1998; p.218.
15.Gamborg OL, Millar RA, Ojiva K. Nutrients requirements of suspension cultural of soybean root cells. Exp Cell Res. 1968;50:148-151.
16.Jannatul F, Hanafi MM, Rafii MY, Kharidah M. A quick DNA extraction protocol: Without liquid nitrogen in ambient temperature. African Journal of Biotechnology. 2012;11(27):6956-6954.
17.Spanu P. HCf-1 a hydrophoben gene of Cladosporium fulvum, does not affect pathogenicity in tomato. Physiol Mol Plant Pathol. 1998;52:323-334.
18.Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor. 1989.
19.White TJ, Bruns T, Lee S, Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR protocols: A guide to methods and applications. 1990;18:315-322.
20.Mishra PK, Fox RTV, Culham A. Development of a PCR-based assay for rapid and reliable identification of pathogenic Fusaria. Fems Microbiology letters 218 (2003). Elsevier Science B.V. 2002; 329-332.
21.Lelliott RA, Stead DE. Methods for the Diagnosis of Bacterial Diseases of Plants. Methods in Plant Pathology, Volumen 2, Series editor: TF Preece. Publised on behalf of the British Society for Plant Pathology by Blakwell Scientific Publications. Oxford London. 1987; 37-200 p.
22.Balmas V. Identificación morfológica de aislados de Fusarium spp. Comunicación personal. 20 abril, 2014.
Recibido: 2-12-2014.
Aceptado: 6-6-2015.
* Autor para correspondencia: Fernando Rivas Figueroa. Correo electrónico: frivas@espoch.edu.ec.