INTRODUCCIÓN
Los ríos son importantes fuentes de agua dulce y un recurso imprescindible para el desarrollo de diferentes actividades sociales y económicas; sin embargo, el incremento poblacional y la contaminación producida por el ser humano, las actividades industriales y agrícolas, constituyen importantes factores para el rápido deterioro de los ecosistemas dulceacuícolas. La determinación de la diversidad microbiana debe realizarse como primer paso en los procesos de recuperación ambiental, para entender el papel de los microorganismos como responsables de los ciclos biogeoquímicos y como sensores de cambios ambientales, considerando al mismo tiempo su valor como fuente abundante de recursos biotecnológicos. En la actualidad el estudio de la biodiversidad constituye uno de los temas más importantes en la investigación científica en Cuba, ya que permite conocer microorganismos con potencialidades en la producción de compuestos con interés ecológico, biotecnológico e industrial. Estudios de caracterización de la diversidad bacteriana en Cuba, solo se han realizado en aguas oceánicas y costeras; sin embargo, no existen antecedentes de este tipo de análisis en ecosistemas dulceacuícolas. El objetivo de esta investigación fue determinar la relación entre la estructura y función de las comunidades bacterianas y la calidad química y microbiológica en 2 ecosistemas dulceacuícolas del occidente de Cuba.
MÉTODOS
Evaluación de la calidad microbiológica y química de las aguas de los ríos Almendares y San Juan
Se tomaron muestras puntuales de 3 estaciones de muestreo del río Almendares (Río Cristal, Paila y Puente de Hierro) y de 3 estaciones del río San Juan (presa El Palmar, presa San Juan y Baños del San Juan) durante el periodo 2013-2017. Estas estaciones se seleccionaron de acuerdo a caracterizaciones previas realizadas en ambos ríos.1,2 La recolección de las muestras, así como la determinación de la temperatura, pH, conductividad eléctrica, sólidos totales disueltos (STD), oxígeno disuelto, nutrientes (PO43--P, NH4+-N, NO2--N, NO3--N), demanda química de oxígeno (DQO), indicadores bacterianos (Escherichia coli, enterococos y coliformes termotolerantes) se realizaron según Larrea et al.3
Análisis de las comunidades bacterianas de los ríos Almendares y San Juan mediante electroforesis en gel con gradiente desnaturalizante y pirosecuenciación 454
La extracción del ADN a partir de las muestras de agua y posterior análisis de las comunidades bacterianas mediante la electroforesis en gel con gradiente desnaturalizante Denaturing Gradient Gel Electrophoresis (DGGE por sus siglas en inglés).3 La pirosecuenciación 454 del gen ADNr 16S se empleó como técnica complementaria de la DGGE para el análisis de las comunidades bacterianas de ambos ríos. Se utilizaron los cebadores 27F (5´-GAGTTTGATCNTGGCTCAG-3´) y 519R (5´-GWNTTACNGCGGCKGCTG-3´) para abarcar las regiones variables V1-V3 del gen ADNr 16S del dominio bacteria. Esta técnica se realizó en el Laboratorio de Investigación y Ensayo (RTL) de Lubbock, Texas, Estados Unidos. Las secuencias generadas en este estudio se pueden descargar a partir de los archivos de lectura de secuencias del Centro Nacional para la Información Biotecnológica (NCBI, por sus siglas en inglés National Center for Biotechnology Information), con el número de acceso SRP063983.
Determinación de la actividad proteolítica extracelular y la actividad fosfatasa ácida extracelular en las aguas de los ríos Almendares y San Juan
En todas las estaciones de muestreo de los ríos Almendares y San Juan se determinó la actividad proteolítica y la actividad fosfatasa ácida a partir de las muestras de agua. La metodología empleada se describe en Izquierdo et al.4 y Larrea et al.5
Caracterización micromorfológica-tintorial, determinación de la producción de enzimas extracelulares y caracterización molecular de los aislados del río Almendares
Para el aislamiento de bacterias productoras de enzimas extracelulares se colectaron muestras de la columna de agua y de plantas procedentes de las 3 estaciones del río Almendares. La metodología empleada para la caracterización micromorfológica-tintorial, la determinación de la producción de enzimas extracelulares y la caracterización molecular de los aislados se describe en Larrea et al.6
Análisis biométricos
Para investigar la relación entre la composición de la comunidad bacteriana y los indicadores físico-químicos e indicadores bacterianos de contaminación fecal en ambos ríos, se realizó el análisis de redundancia (RDA) utilizando el paquete estadístico CANOCO, versión 4.5 para windows. El RDA se seleccionó después de que se comprobó mediante el análisis de correspondencia sin tendencia (DCA) que los datos de la comunidad bacteriana exhibían una respuesta lineal a los gradientes de las variables físico-químicas y de contaminación fecal. Se realizó una selección a priori de los indicadores físico-químicos e indicadores bacterianos significativos (p < 0,05) mediante la prueba de permutación de Monte Carlo (999 permutaciones no restringidas) bajo el modelo reducido. Las variables correlacionadas se eliminaron antes de realizar el RDA. Para representar los gráficos triplots entre las estaciones de muestreo, las variables físico-químicas e indicadores de contaminación y las clases bacterianas se utilizó el programa CANODRAW incluido en el paquete estadístico CANOCO 4.5.7
Para evaluar la correlación entre los indicadores físico-químicos, los indicadores de contaminación fecal, la actividad proteolítica, la actividad fosfatasa ácida y la concentración de bacterias proteolíticas se calculó el coeficiente de correlación de Pearson (r, método paramétrico) para los casos en los cuales se cumplieron con las premisas. En los que no se cumplieron, se realizó la prueba no paramétrica de correlación lineal de Spearman. Los análisis estadísticos se realizaron en el programa statistica versión 8.0.8
RESULTADOS Y DISCUSIÓN
Identificación de las comunidades bacterianas de los ríos Almendares y San Juan mediante electroforesis en gel con gradiente desnaturalizante y pirosecuenciación 454
A partir de la DGGE obtenida con las muestras del río Almendares, se cortaron 10 bandas, las cuales se reamplificaron y se secuenciaron. La identificación de las bandas secuenciadas evidenció el predominio de la clase Gammaproteobacteria, siendo identificadas 5 bandas como pertenecientes a esta clase. Además, se identificaron 2 bandas pertenecientes a la clase Betaproteobacteria, 1 banda relacionada con la clase Flavobacteria, 1 banda relacionada con el filo actinobacteria y 1 banda relacionada con el filo Chloroflexi.3 En el caso del río San Juan se seleccionaron 12 bandas. Se obtuvieron 6 bandas relacionadas con la clase Actinobacteria, 5 bandas relacionadas con la clase Betaproteobacteria y 1 banda relacionada con la clase Bacilli.3
Mediante la pirosecuenciación 454 se obtuvieron en las estaciones del río Almendares un total de 35 872 secuencias y se apreció que los filos Proteobacteria (52,7 %), Actinobacteria (24,6 %), Bacteroidetes (17,5 %) y Firmicutes (3,96 %) eran los más abundantes en el periodo de estudio con más de 500 secuencias (figura 1A).

Fig. 1 Distribución porcentual taxonómica de los diferentes filo/clase en las estaciones de muestreo de los ríos Almendares (A) y San Juan (B). Estaciones del río Almendares: RC, Río Cristal; P, Paila; PH, Puente de Hierro. Meses: Feb2, febrero 2013; Abr2, abril 2013; Jun2, junio 2013. Estaciones del río San Juan: S-1, presa El Palmar; S-2, presa San Juan; S-3, Baños del San Juan. Meses: Feb2, febrero 2013; Abr2, abril 2013; Jul2, julio 2013.
Dentro del filo Proteobacteria las clases Gammaproteobacteria (76 %), Betaproteobacteria (15 %) y Alphaproteobacteria (9 %) fueron las más frecuentes. En el río San Juan se obtuvieron un total de 47 305 secuencias y se apreció que los filos Actinobacteria (39,1 %), Proteobacteria (30,9 %), Cyanobacteria (18,9 %), Bacteroidetes (5,1 %), Firmicutes (3,6 %) y Deinococcus-Thermus (1,1 %) eran los más abundantes en el periodo de estudio. Dentro del filo Proteobacteria las clases Alphaproteobacteria (63 %), Betaproteobacteria (22 %) y Gammaproteobacteria (15 %) fueron las más frecuentes (figura 1B).
Tomando en consideración la clasificación taxonómica hasta el nivel de familia, se identificaron en el río Almendares un total de 73 familias pertenecientes a 44 órdenes. Particularmente dentro de la clase Gammaproteobacteria, se identificaron con mayor frecuencia las familias: Moraxellaceae, Xanthomonadaceae, Pseudomonadaceae, Methylococcaceae y Enterobacteriaceae (figura 2). En cambio, en el río San Juan se identificaron un total de 60 familias pertenecientes a 38 órdenes, y específicamente dentro de la clase Alphaproteobacteria, se identificaron con mayor frecuencia las familias: Sphingomonadaceae y Caulobacteraceae (figura 2).

Fig. 2 Distribución porcentual taxonómica de las diferentes familias identificadas en los ríos Almendares y San Juan.
De las familias identificadas en ambos ríos 43 constituyen nuevos informes para ecosistemas dulceacuícolas del Caribe. Por primera vez para el Caribe se identifican los miembros de las comunidades bacterianas de ecosistemas dulceacuícolas con diferente grado de contaminación hasta el nivel de familia, de los cuales 43 familias constituyen nuevos informes para ecosistemas dulceacuícolas de esta área, lo que representa un aporte al conocimiento de la diversidad bacteriana. Además, por primera vez se analiza la comunidad bacteriana de un ecosistema dulceacuícola en una reserva de biosfera como Sierra del Rosario, lo cual contribuye al incremento del valor biológico de esta área protegida.
Determinación de la relación entre la estructura de las comunidades bacterianas y la calidad química y microbiológica en los ecosistemas dulceacuícolas Almendares y San Juan
En el río Almendares, las altas concentraciones de amonio, fosfatos, enterococos, Escherichia coli y coliformes termotolerantes fueron los factores que más influyeron en la variabilidad de la composición de las comunidades bacterianas en el río Almendares (p < 0,01); todos estos indicadores muestran un mismo fenómeno: la contaminación fecal.
De acuerdo al análisis de redundancia, el porcentaje de la varianza acumulada explicada por la relación especies-variables ambientales indicó que el primer y segundo ejes canónicos describen el 79,5 % de la varianza. Las correlaciones especies-ambiente de cada eje fueron de 0,991 y 0,981 para el primer y segundo eje respectivamente (figura 3).
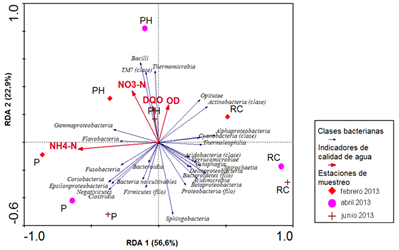
Fig. 3 Triplot del análisis de redundancia (RDA) de la composición de la comunidad bacteriana de las estaciones de muestreo del río Almendares en el año 2013 utilizando indicadores físico-químicos. Las flechas rojas señalan la dirección en la que se incrementa el valor de cada indicador de calidad de agua. Las flechas azules representan las diferentes clases bacterianas. La longitud de las flechas indica el grado de correlación con el eje representado. La posición de las estaciones de muestreo relativa a las flechas se interpreta por la proyección de los símbolos sobre las flechas, e indica el grado de influencia del indicador de calidad de agua representado por la flecha roja sobre la composición de la comunidad bacteriana de la estación de muestreo. NO3-N (nitratos), NH4-N (amonio), OD (oxígeno disuelto), DQO (demanda química de oxígeno), RC, Río Cristal; P, Paila; PH, Puente de Hierro.
Las clases bacterianas se distribuyeron de acuerdo a las características significativas de las estaciones de muestreo del río Almendares. Las clases Thermoleophilia, Cyanobacteria, Alphaproteobacteria, Actinobacteria, Sphingobacteria, Acidobacteria, Verrucomicrobiae, Cytophagia, Spirochaetia, Deltaproteobacteria, Betaproteobacteria, Acidimicrobia, Opitutae y bacterias no cultivables pertenecientes a los filos Proteobacteria y Bacteroidetes, se asociaron a la estación Río Cristal caracterizada por las bajas concentraciones de amonio, DQO y nitratos. Las clases Bacilli, TM7 y Thermomicrobia se asociaron a la estación Puente de Hierro, caracterizada por altas concentraciones de nitrato y DQO. Las clases Gammaproteobacteria y Flavobacteria se asociaron a las estaciones Paila y Puente de Hierro, ambas caracterizadas por las altas concentraciones de amonio. Las clases Fusobacteria, Coriobacteria, Epsilonproteobacteria, Negativicutes, Clostridia, Bacteroidia, bacterias no cultivables pertenecientes al filo Firmicutes y bacterias no cultivables dentro del dominio bacteria se asociaron a la estación Paila, caracterizada por las altas concentraciones de amonio y bajas concentraciones de oxígeno disuelto.
En el río San Juan, la concentración de oxígeno disuelto y las bajas concentraciones de nutrientes fueron los factores que más influyeron en la variabilidad de la composición de las comunidades bacterianas (p < 0,05).9 De acuerdo al análisis de redundancia, el porcentaje de la varianza acumulada explicada por la relación especies-variables ambientales indicó que el primer y segundo ejes canónicos describen el 70,6 % de la varianza. Las correlaciones especies-ambiente de cada eje fueron de 0,986 y 0,989 para el primer y segundo eje respectivamente (figura 4).

Fig. 4 Triplot del análisis de redundancia (RDA) de la composición de la comunidad bacteriana de las estaciones de muestreo del río San Juan en el año 2013 utilizando indicadores físico-químicos. Las flechas rojas señalan la dirección en la que se incrementa el valor de cada indicador de calidad de agua. Las flechas azules representan las diferentes clases bacterianas. La longitud de las flechas indica el grado de correlación con el eje representado. La posición de las estaciones de muestreo relativa a las flechas se interpreta por la proyección de los símbolos sobre las flechas, e indica el grado de influencia del indicador de calidad de agua representado por la flecha roja sobre la composición de la comunidad bacteriana de la estación de muestreo. OD, oxígeno disuelto; PO4-P, fosfatos; DQO, demanda química de oxígeno; STD, sólidos totales disueltos; S-1, presa El Palmar; S-2, presa San Juan; S-3, Baños del San Juan.
Las clases bacterianas también se distribuyeron de acuerdo a las características significativas de las estaciones de muestreo. Las clases Flavobacteria, Opitutae, Alphaproteobacteria, Betaproteobacteria, Gammaproteobacteria, Acidimicrobia, Actinobacteria, Chloroflexi, Clostridia, Cytophagia y las bacterias no cultivables pertenecientes al filo Bacteroidetes se asociaron a la estación Baños del San Juan caracterizada por un pH ligeramente alcalino y altas concentraciones de fosfato y altas concentraciones de STD. Las clases Epsilonproteobacteria, Thermomicrobia, Thermoleophilia, Deinococci, Bacilli, Sphingobacteria, Acidobacteria, Cyanobacteria y las bacterias no cultivables pertenecientes a los filos Acidobacteria y Verrucomicrobia se asociaron a la estación presa San Juan, caracterizada por bajas concentraciones de DQO, altas concentraciones de oxígeno disuelto, bajas concentraciones de fosfato y pH ligeramente ácido. En el caso particular de la clase Cyanobacteria, también se asoció a la estación presa El Palmar caracterizada por bajas concentraciones de fosfatos, altas concentraciones de oxígeno disuelto y pH ligeramente ácido. Por primera vez en el Caribe se realiza el análisis de la relación entre factores ambientales naturales y antrópicos y la composición de las comunidades bacterianas.
Determinación de la actividad proteolítica y la actividad fosfatasa ácida en los ríos Almendares y San Juan y su relación con indicadores físico-químicos y microbiológicos
La mayor actividad proteolítica se detectó en el río San Juan en comparación al río Almendares lo que indica que en ecosistemas más contaminados la actividad enzimática proteolítica disminuye respecto a otro sistema menos impactado. Además, se evidenció la variación temporal anual de la actividad proteolítica en ambos ríos.4 La actividad enzimática proteolítica no siempre se corresponde con la presencia de bacterias proteolíticas, lo que indica que su actividad depende de diversos factores bióticos y abióticos como la composición de la comunidad microbiana, la disponibilidad de materia orgánica disuelta y la concentración de nutrientes. En los análisis de correlación en el río San Juan se obtuvo relación entre la actividad proteolítica, la concentración de bacterias productoras de proteasas y los indicadores físico-químicos de la calidad del agua. Esto indica que la actividad proteolítica está relacionada con contaminantes de origen natural procedentes de la descomposición de las hojas, los escurrimientos o los productos de la actividad fitoplanctónica.4 En cambio, en el río Almendares no se observó esta relación, lo que indica que la actividad proteolítica se encuentra afectada por otros contaminantes vertidos a sus aguas no evaluados en este estudio.4 Además, se obtuvo que la mayoría de las aislados bacterianos proteolíticos en ambos ríos eran bacilos grampositivos formadores de endosporas, lo cual demuestra el predominio de especies pertenecientes a la clase Bacilli.10,11
La mayor actividad fosfatasa ácida se detectó en el río Almendares en comparación al río San Juan, lo cual puede deberse a los altos valores de materia orgánica encontrada en el río Almendres y el pH ligeramente alcalino observado en el río San Juan.5 Con anterioridad, en sistemas dulceacuícolas con un mayor impacto antropogénico se han detectado incrementos de la actividad enzimática fosfatasa. Además, se evidenció la variación espacio-temporal de la actividad fosfatasa ácida en ambos ríos siendo significativas las diferencias entre el periodo lluvioso y el periodo poco lluvioso en el río San Juan.
En los análisis de correlación en el río San Juan se obtuvo una relación positiva entre la actividad fosfatasa ácida, la clorofila a, la concentración de Eschericia coli y la concentración de heterótrofos totales y una relación negativa con el pH. En cambio, en el río Almendares se observó una relación positiva con la demanda química de oxígeno y la concentración de nitratos.5 Estos resultados muestran que, en los ecosistemas de agua dulce con pH neutro o ligeramente alcalino, la actividad potencial de la fosfatasa ácida puede estar presente y también juega un papel importante en el reciclaje de materia orgánica en estos entornos. Además, en sistemas de agua dulce contaminados, esta actividad está más influenciada por factores contaminantes (ej. concentración de nitratos, materia orgánica) en comparación con sistemas menos impactados donde depende de las condiciones naturales (ej. precipitación, pH, actividad del fitoplancton) por lo que la actividad de la fosfatasa ácida en la columna de agua puede ser un buen indicador del impacto antropogénico sobre el funcionamiento microbiano en ecosistemas de agua dulce.
Por primera vez en Cuba se relaciona la actividad enzimática proteolítica y la actividad fosfatasa ácida con la calidad de agua de ecosistemas dulceacuícolas, lo que evidencia la factibilidad del uso de las actividades enzimáticas extracelulares como indicadores del estado ecológico de estos sistemas.
Caracterización micromorfológica-tintorial, determinación de la producción de enzimas extracelulares y caracterización molecular de los aislados del río Almendares
En el presente estudio, los bacilos grampositivos formadores de endosporas predominaron entre los aislados de muestras de agua y rizosedimento del río Almendares, ecosistema contaminado con altas concentraciones de materia orgánica como se apreció a través del indicador DQO.6,10,11 Teniendo en cuenta que los miembros de la clase Bacilli son bacilos grampositivos, aerobios y formadores de endosporas se puede inferir que la mayoría de los aislados de esta investigación pertenecen a esta clase. Se observó la producción de proteasas y amilasas a partir de las bacterias formadoras de endosporas aisladas.10 A partir de la producción de las enzimas proteasas y amilasas, se seleccionaron 36 aislados, los cuales fueron representativos de las 3 estaciones de muestreo del río Almendares, así como de las diferentes plantas colectadas. Con respecto a los aislados del rizosedimento, no existen informes previos de aislamientos a partir del rizosedimento de las plantas Eichhornia crassipes, Pistia startiotes, Commelina sp., Hydrocotyle sp., Cabomba sp. y Bidens pilosa L. en Cuba.
Mediante el sistema API ZYM, se constataron las capacidades metabólicas de los aislados seleccionados, los cuales produjeron fosfatasas, esterasas, aminopeptidasas y glicosil hidrolasas.6 Este resultado brinda una información aproximada de la composición polimérica de las fuentes de materia orgánica que pudieran estar presentes en el río Almendares, así como de las capacidades de los microorganismos aislados de este ecosistema. De esta forma, los microorganismos aislados contribuyen a la degradación de la materia orgánica en descomposición en el río Almendares participando en el reciclaje de nutrientes como el carbono, el fósforo y el nitrógeno a la biosfera.
A partir de la producción de enzimas extracelulares detectadas mediante el sistema API ZYM se seleccionaron 15 aislados bacterianos. Los aislados seleccionados se identificaron a través de la secuenciación del gen ADNr 16S. El análisis de la comparación de las secuencias del gen ADNr 16S de cada cepa con las secuencias de referencia en el Centro Nacional de Información Biotecnológica de Estados Unidos (NCBI), reveló que 10 mostraron similitud con el grupo Bacillus cereus sensu lato, 2 con Bacillus megaterium, 1 con Bacillus pumilus y 2 con Bacillus subtilis. Las cepas pertenecientes al grupo B. cereus se identificaron también a través de la secuenciación del gen gyrB, lo cual confirmó los resultados obtenidos con el gen ADNr 16S, pero no permitió la diferenciación de las cepas dentro del grupo. Sin embargo, la presencia de cristales proteicos permitió identificar las cepas como Bacillus thuringiensis.6 Las cepas identificadas fueron depositadas en la colección de la Facultad de Biología de la Universidad de La Habana bajo los números CCMFB-B225 al CCMFB-B239. Por primera vez en Cuba se identifican cepas de Bacillus sp. aisladas de la columna de agua y el rizosedimento de las plantas Eichhornia crassipes, Pistia stratiotes, Hydrocotyle sp., Bidens pilosa L. y Commelina sp. de un ecosistema dulceacuícola contaminado, productoras de enzimas extracelulares con interés ecológico.
Conclusiones
Esta investigación permitió obtener evidencias concretas sobre la utilidad práctica del empleo de las comunidades bacterianas como indicadoras del estado ecológico de aguas superficiales de clima tropical. Los resultados obtenidos contribuyen a la sostenibilidad medioambiental del complejo turístico Las Terrazas, pues brindan información oportuna sobre el estado actual de la calidad del agua del río San Juan (Las Terrazas, Artemisa), el cual se encuentra ubicado en la Reserva de la Biosfera Sierra del Rosario. La información obtenida en este trabajo avala la factibilidad del Programa de Desarrollo Sostenible que se lleva a cabo en la comunidad de Las Terrazas. Además, estos resultados han contribuido a mitigar los efectos de la contaminación en este ecosistema, ya que a partir de los datos brindados a la Estación Ecológica Sierra del Rosario se han podido acometer medidas correctivas para mejorar la calidad ambiental y la preservación del recurso agua. Los resultados que se presentan constituyen un aporte al conocimiento de la diversidad bacteriana de ecosistemas dulceacuícolas cubanos. Se brinda información acerca de las diferentes familias bacterianas presentes en los ríos Almendares y San Juan, lo cual constituye la base para cualquier acción de recuperación que se lleve a cabo en el río Almendares y contribuye a aumentar el valor biológico del área protegida donde se localiza el río San Juan. Por primera vez para el Caribe se identifican los miembros de las comunidades bacterianas de ecosistemas dulceacuícolas con diferente grado de contaminación hasta el nivel de familia, de los cuales 43 familias constituyen nuevos informes para ecosistemas dulceacuícolas de esta área, lo que representa un aporte al conocimiento de la diversidad bacteriana.














