Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.56 n.3 Ciudad de la Habana sep.-dic. 2004
Comparación entre 5 métodos para la extracción de ADN de Triatomíneos: su utilización en la técnica de ADN polimórfico amplificado al azar (RAPD)
Lic. Jorge Fraga Nodarse,1 Lic. Jinnay Rodríguez,2 Lic. Omar Fuentes,3 Lic. Mayda Castex4 y Lic. Aymé Fernández-Calienes5
Resumen
Se evaluó la aplicabilidad de 5 protocolos útiles para la extracción del ADN genómico de Triatomíneos, y se describió el método del acetato de potasio modificado, como un método con el que se obtiene un alto rendimiento y pureza del ADN en el menor tiempo y costo, para su utilización como molde en la técnica de ADN polimórfico amplificado al azar (RAPD [la cual es un método simple para detectar el polimorfismo genético del ADN]) y probablemente en otras técnicas moleculares basadas en la amplificación por la reacción en cadena de la polimerasa. La calidad del ADN constituye un elemento crucial para esta técnica, la cual necesita un método de extracción de ADN lo más estandarizado posible con el que se obtenga un ADN puro, no degradado, libre de ARN y de inhibidores de la reacción en cadena de la polimerasa: porque cambios en la pureza afectan los perfiles de amplificación y esto se manifiesta en la presencia de bandas falsas y en la poca reproducibilidad del ensayo.
Palabras clave: RAPD, extracción de ADN, Triatomíneos.
Los triatomíneos desempeñan un papel importante en la transmisión de la enfermedad de Chagas. En Cuba existe el riego de la enfermedad vectorial al encontrarse en la isla 4 especies de triatomíneos: Triatoma flavida (Neiva, 1911), Triatoma bruneri (Usinger, 1944), Bolbodera scabrosa (Valdés, 1990) y Triatoma rubrofasciata (De Geer, 1973). La especie T. flavida es considerada una especie selvática que presumiblemente es atraída a las casas por la luz; de las especies encontradas en Cuba es la más abundante, y posee características que aumentan su peligrosidad como vector potencial.1
El análisis de las variaciones genéticas de las poblaciones de triatomíneos utilizando métodos moleculares constituye un elemento esencial en estudios taxonómicos, biosistemáticos, epidemiológicos, de genética poblacional y de evolución.2,3 La técnica del ADN polimórfico amplificado al azar (RAPD) representa un ensayo para el estudio del polimorfismo genético del ADN. Este consiste en la amplificación de segmentos de ADN genómico mediante la tecnología de la reacción en cadena de la polimerasa (RCP), utilizando como cebadores oligonucleótidos cortos diseñados al azar.4 El RAPD requiere de cantidades muy pequeñas de ADN y no es necesario el clonaje, la secuenciación o la hibridización, lo que constituye una ventaja sobre otras técnicas moleculares usadas en la caracterización genómica. Sin embargo diferentes estudios han demostrado que varios factores afectan los perfiles obtenidos, manifestándose en la presencia de bandas falsas y la poca reproducibilidad del ensayo.5,6
Uno de los factores que afectan los patrones del RAPD es la calidad del ADN en la reacción.7 En este trabajo se reportó el uso de un método rápido de extracción de ADN genómico, con elevada pureza y rendimiento, que facilita la caracterización genética y molecular de poblaciones de triatomíneos en Cuba utilizando la técnica del RAPD.
Métodos
Se utilizaron en el estudio 10 ejemplares de T. flavida colectados en la cueva Caimanera, Península de Guanahacabibes, Pinar del Río, Cuba. A los 10 triatomas colectados se le aisló el ADN genómico a partir de una pata, utilizando 5 métodos de extracción diferentes.
Método de calentamiento: la pata fue homogeneizada de forma manual en nitrógeno líquido y 50 µL de agua destilada. Se incubó a 93 °C durante 15 min. Se realizó una centrifugación a 8 000 g por 10 min y el sobrenadante se conservó a - 20 °C.
Método fenol-cloroformo:3 la pata fue homogeneizada de forma manual en nitrógeno líquido y 1 mL de tampón de lisis (tris-HCl 50mM pH 8,25; EDTA 50 mM; NaCl 50 mM; SDS 1 %). La suspensión se incubó con 2 mg/mL de proteinasa K (Boehringer Mannheim) toda la noche a 37 °C y seguidamente se realizaron 3 extracciones de proteínas con igual volumen de fenol, fenol-cloroformo-alcohol isoamílico (25:24:1) y cloroformo-alcohol isoamílico (24:1), con sus respectivas centrifugaciones a 8 000 g por 10 min a 4 °C. El ADN se precipitó con 2 volúmenes de etanol absoluto y 0,1 volumen de acetato de sodio 3 M pH 5,3; durante 30 min a - 20 °C. El precipitado de ADN genómico que se obtuvo por centrifugación a 10 000 g durante 20 min se lavó con etanol 70 % y se secó a temperatura ambiente; fue resuspendido finalmente en 50 mL de tampón tris-EDTA (TE) (tris-HCl 1 mM pH 8,0; EDTA 1 mM pH 8,0). El ARN presente en la muestra se digirió con la adición de RNasa H (Boehringer Mannheim, Germany), incubándose durante 1 h a 37 °C. Seguidamente se realizó una extracción empleando un volumen de cloroformo-alcohol isoamílico (24:1) y el sobrenadante se conservó a - 20 °C.
Método del acetato de potasio:8,9 la pata fue homogeneizada de forma manual en 100 µL de tampón de lisis (NaCl 0,1 M; sacarosa 0,2 M; EDTA 50 mM; tris-HCl 100 mM pH 8,25; SDS 0,05 %). La suspensión se incubó durante 30 min a 65 °C. Los ácidos nucleicos se extrajeron adicionando 14 µL de acetato de potasio 8 M incubándose en hielo durante 15 min, seguido de una centrifugación a 8 000 g durante 10 min a 4 ºC donde se colectó la fase acuosa. El ADN se precipitó a - 20 ºC durante 30 min en presencia de 2 volúmenes de etanol absoluto que contenía acetato de sodio 0,3 M. Posteriormente, se centrifugó la mezcla a 10 000 g durante 20 min y el precipitado se lavó con etanol (70 %). Después de secar a temperatura ambiente, el ADN se disolvió en 50 µL de tampón TE. El ARN restante se eliminó con RNAsa H (Boehringer Mannheim), la suspensión se incubó a 37 ºC durante 1 h. Después de extraer con igual volumen de cloroformo-alcohol isoamílico (24:1), la fase acuosa se conservó a - 20 °C.
Método del acetato de potasio modificado: la pata fue homogeneizada de forma manual en nitrógeno líquido y 150 µL de tampón de lisis (tris-HCl 20 mM pH 8,25; EDTA 25 mM; NaCl 25 mM; SDS 1 %). La suspensión se incubó con 100 µg/mL de proteinasa K (Boehringer Mannheim) durante 1 h a 56 °C. Los ácidos nucleicos se extrajeron adicionando 100 µL de acetato de potasio 3 M incubándose en hielo durante 1 h, seguido de una centrifugación a 8 000 g durante 10 min a 4 ºC donde se colectó la fase acuosa. El ADN se precipitó a - 20 ºC durante 30 min en presencia de 2 volúmenes de etanol absoluto que contenía acetato de sodio 0,3 M. Posteriormente, se centrifugó la mezcla a 10 000 g durante 20 min y el precipitado se lavó con etanol (70 %). Después de secar a temperatura ambiente, el ADN se disolvió en 50 µL de tampón TE. El ARN restante se eliminó con RNAsa H (Boehringer Mannheim), la suspensión se incubó a 37 ºC durante 1 h. Después de extraer con igual volumen de cloroformo-alcohol isoamílico (24: 1), la fase acuosa se conservó a - 20 °C.
Método de bromuro de cetil-trimetil amonio (CTAB):10 la pata fue homogeneizada de forma manual en nitrógeno líquido y 500 µL de tampón de lisis (tris-HCl 20 mM pH 8,25; EDTA 25 mM; NaCl 25 mM; SDS 1 %). La suspensión se incubó con 100 µg/mL de proteinasa K (Boehringer Mannheim) durante 1 h a 56 °C. Los ácidos nucleicos se extrajeron adicionando 150 µL de NaCl 5 M y 1/10 del volumen de 10 % de CTAB, incubándose durante 10 min a 65 ºC, seguidamente se realizaron 2 extracciones de proteínas con igual volumen de fenol-cloroformo-alcohol isoamílico (25:24:1) y cloroformo-alcohol isoamílico (24:1) con sus respectivas centrifugaciones a 8 000 g por 10 min a 4 °C. El ADN se precipitó con 2 volúmenes de etanol absoluto y 0,1 volumen de acetato de sodio 3 M pH 5,3; durante 30 min a - 20 °C. El precipitado de ADN genómico que se obtuvo por centrifugación a 10 000 g durante 20 min se lavó con etanol 70 % y se secó a temperatura ambiente, y fue resuspendido finalmente en 50 mL de tampón TE. El ARN presente en la muestra se digirió con la adición de RNasa H (Boehringer Mannheim, Germany), incubándose durante 1 h a 37 °C. Seguidamente se realizó una extracción empleando un volumen de cloroformo-alcohol isoamílico (24:1) y el sobrenadante se conservó a - 20 °C.
La concentración de ADN extraído por los diferentes métodos se estimó espectrofotométricamente mediante su absorbancia a 260 nm. La pureza de la muestra se examinó mediante la relación de las absorbancias a 260 nm y 280 nm y por electroforesis en gel de agarosa (0,8 % en tampón TBE (tris-borato 0,045 M; EDTA 0,001 M) que contenía bromuro de etidio (0,5 mg/mL) y utilizando un transiluminador UV (Macrovue 2011, LKB) para su visualización.11
El ADN extraído por los diferentes métodos (25 ng) se amplificó en un volumen de reacción de 25 mL, utilizando los cebadores OPA-1, OPA-2 y OPA-3 (Kit A, Operon Technologies, USA) manteniendo constante el resto de los componentes de la reacción (2,5 mL de tampón de amplificación 10 x (tris-HCl 100 mM pH 8,3; KCl 500 mM; gelatina 0,01 %) (Promega, USA), 200 mM de cada deoxinucleótido trifosfato (Promega, USA), 2,5 mM de MgCl2, 5 pmol de cebador, 2 U Taq ADN polimerasa (Promega, USA)) con el objetivo de determinar la calidad del ADN para ser utilizado en la técnica de RAPD. La amplificación se realizó en un termociclador (Perkin Elmer, USA) con el siguiente perfil: desnaturalización inicial a 94 °C por 5 min, seguido de 45 ciclos de: desnaturalización a 94 °C por 1 min, hibridación a 36 °C por 1 min y extensión 72 °C por 2 min, con una extensión final después del último ciclo a 72 °C por 15 min. Para la detección del producto se analizaron 20 mL de cada mezcla resultante en electroforesis en gel de agarosa 1,2 %, preparado en tampón TBE 0,5 x con bromuro de etidio 0,5 mg/mL. La visualización de los productos de amplificación se realizó mediante luz ultravioleta.
Los experimentos se repitieron 3 veces en diferentes días con el objetivo de chequear la reproducibilidad de los resultados del RAPD.
Análisis estadístico: se determinaron las medias y las desviaciones estándares de los valores: material de partida, concentración de ADN y rendimiento, para cada uno de los métodos de extracción analizados.12 Los resultados fueron procesados con ayuda del paquete estadístico para Windows (GraphPad Prism versión 3.03 para Windows, GraphPad Software, San Diego California, 1999). Se utilizó la prueba paramétrica de ANOVA para comparar las medias entre los parámetros analizados. Se consideraron significativos los valores de p menores que 0,01.
Resultados
En la tabla se observa la media de la cantidad de material de partida (mg) para cada uno de los métodos analizados, no se encontraron diferencias significativas (p= 0,9988) al comparar las medias del material de partida utilizado para la extracción de ADN por cada uno de los métodos.
Tabla. Comparación del tiempo de procesamiento, concentración, rendimiento y pureza entre las muestras de ADN extraídas por los diferentes métodos
| Método de extracción | Tiempo | Material de partida (mg)a | Concentración de ADN (µg/µL)b | Rendimiento (µg ADN/mg de material de partida)c | Purezad |
| Método calentamiento | 30 min | 1,88 (0,34) | 0,5785 (0,111) | 15,81 (4,15) | 0,79 |
| Método del fenol-cloroformo | 19 h | 1,89 (0,36) | 0,1305 (0,039) | 3,49 (0,92) | 1,96 |
| Método del acetato de potasio | 4 h | 1,85 (0,20) | 0,1414 (0,027) | 3,81 (0,77) | 1,93 |
| Método del acetato de potasio modificado | 4 h | 1,86 (0,32) | 0,2675 (0,028) | 7,38 (1,52) | 1,94 |
| Método del CTAB | 4 h | 1,85 (0,38) | 0,1650 (0,025) | 4,67 (1,48) | 1,90 |
a: valor medio del material de partida para la extracción de ADN por lo diferentes métodos, con la desviación estándar entre paréntesis (n= 10); b: valor medio de la concentración de ADN obtenida con la desviación estándar entre paréntesis(n= 10); c: valor medio del rendimiento con la desviación estándar entre paréntesis (n= 10); d: valor medio de la razón DO 260 nm/ DO 280 nm (n= 10).
La pureza del ADN extraído por cada uno de los métodos de extracción de ADN se corroboró mediante electroforesis en gel de agarosa. Se obtuvo ADN libre de contaminación con ARN, no degradado, correspondiéndose la intensidad de la banda de ADN con la concentración de las muestras medidas espectrofotométricamente en todos los métodos analizados; exceptuando el método de calentamiento, donde no se observa un patrón de ácidos nucleicos (datos no mostrados). Con este método se obtienen los mayores valores de concentración y rendimiento en el menor tiempo, sin embargo la pureza del mismo es baja (tabla).
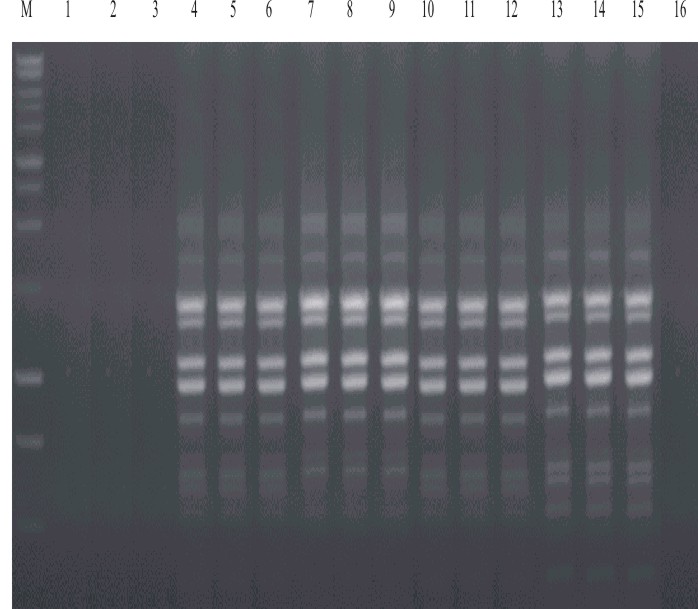
El ADN obtenido con 4 de los 5 métodos de extracción analizados (fenol-cloroformo, acetato de potasio, acetato de potasio modificado y CTAB) amplifica patrones de RAPD reproducibles con todos los cebadores analizados. Sin embargo, no se obtienen productos de amplificación a partir del ADN extraído por el método de calentamiento, lo que indica la posibilidad de una contaminación con inhibidores de la RCP. En la figura se muestra el patrón de amplificación obtenido con el cebador OPA-2.
Carril M: marcador de peso molecular 1 kb (Promega, USA): Carriles 1-3: ADN obtenido utilizando el método del calentamiento; Carriles 4-6: ADN obtenido por el método del acetato de potasio; Carriles 7-9: ADN obtenido utilizando el método del acetato de potasio modificado; Carriles 10-12: ADN obtenido utilizando el método del CTAB; Carriles 13-15: ADN obtenido utilizando el método del fenol cloroformo;Carril 13: control negativo.
Fig. Patrones genéticos de RAPD repetidos 3 veces en diferentes días utilizando el cebador OPA-2 y 25 ng de ADN obtenido por los diferentes métodos de extracción, de uno de los triatomíneos utilizados en el estudio.
Al comparar las medias de la concentración de ADN para cada uno de los 4 métodos con los que se obtienen patrones de amplificación de RAPD, se encontraron diferencias significativas entre los métodos (p< 0,001) al igual que al comparar las medias del rendimiento obtenido para cada uno de estos (p< 0,001). El método del acetato de potasio modificado resultó ser el mejor para estos propósitos, al posibilitar la extracción de ADN en menor tiempo, con elevada concentración, rendimiento y pureza.
Discusión
El desarrollo de los métodos moleculares ha llevado a la necesidad de crear métodos de extracción de ADN más simples y eficientes, sin embargo, variaciones en la eficiencia de la lisis, el rendimiento y la pureza del ADN pueden afectar los resultados de técnicas moleculares como la RCP, la hibridización y el clonaje.13 La técnica de RAPD necesita un método de extracción de ADN genómico lo más estandarizado posible con el que se obtenga un ADN puro, no degradado, libre de ARN y de inhibidores de la RCP para de esa forma obtener patrones reproducibles de RAPD.
La técnica de RAPD ha proporcionado las bases moleculares para reevaluar las relaciones taxonómicas entre subfamilias de triatomíneos,3,14 así como en estudios epidemiológicos.3 Se ha usado, además, en la caracterización genética de poblaciones de Rhodnius prolixus y Rhodnius columbiensis,13 Psammolestes tertius,15,Triatoma dimidiata,16 Triatoma braziliensis.17,18 En estos estudios se utilizaron fundamentalmente los métodos de extracción de ADN del fenol-cloroformo y del acetato de potasio. El método que se propone aquí ofrece ventajas sobre el método del fenol-cloroformo, la principal desventaja de este último es la cantidad de pasos que lleva, lo que implica mayor manipulación y muy largo el tiempo del proceso y resulta tedioso cuando se analiza un gran número de muestras.14 En contraste con los métodos del acetato de potasio y el método del CTAB, se disminuye la cantidad de pasos y el tiempo total del proceso, minimizándose las posibles contaminaciones accidentales de las muestras durante la extracción, con el cual se obtiene mayor concentración y rendimiento (tabla). La diferencia fundamental entre los métodos del acetato de potasio, lo constituye la utilización de un macerado inicial con nitrógeno líquido y el uso de la proteinasa K, en el caso del procedimiento que aquí se propone, por lo que la combinación de estos pasos al protocolo son los responsables de los mejores resultados obtenidos.
Los resultados de este estudio sugieren el uso del método de extracción de ADN genómico del acetato de potasio modificado, porque constituye un método rápido con el cual se obtiene un ADN genómico de triatomíneos con elevada calidad y pureza para su análisis por la técnica de RAPD y probablemente, para otras técnicas moleculares basadas en la amplificación por la RCP.
Agradecimientos
Este trabajo fue parcialmente financiado por la ECLAT (European Community Latin American Triatominae Research Network). Al doctor Fidel Ángel Nuñez, por su ayuda en el análisis estadístico de los resultados y por la revisión crítica del manuscrito.
Summary
The applicability of 5 protocols useful for the extraction of genomic DNA of triatominae was evaluated and the modified potassium acetate method was described as a method with which a high yield and purity of DNA is obtained in the shortest time and at the lowest cost to be used as a mold in the random amplified polymorphic DNA technique (simple method to detect the DNA genetic polymorphism) and probably in other molecular techniqeus based in the PCR amplification. The DNA quality is a crucial element for this technique, which needs a DNA extraction method as standardized as possible to obtain a pure non degraded DNA free of RNA and of PCR inhibitors, because changes in the purity can affect the amplification profiles and this is manifested in the presence of false bands and in the little reproducibility of the assay.
Key words: RAPD, DNA extraction, Triatominae
Referencias bibliográficas
- Jiménez-Ozete H. Observaciones sobre la biología de Tritoma flavida, Neiva, 1911 en Cuba. Rev Cubana Med Trop 1981;33:42-50.
- Borgues EC, Pires HHR, Barbosa SE, Nunes CMS, Pereira MH, Romanha AJ, Diotaiuti. Genetic variability in brazilian triatomines and the risk of domiciliation. Mem Inst Oswaldo Cruz 1999;94(suppl 1):371-3.
- García AL, Carrasco HJ, Schofield CJ, Russell J, Frame IA, Valente SAS, Miles MA. Random amplified polymorphic DNA as a tool for taxonomic studies of Triatomine Bug (Hemiptera: Reduviidae). J Med Entomol 1998;35(1):38-45.
- Williams JGK, Kubelik AR, Livak KJ, Rafalski JA, Tingey SV. DNA polymorphisms amplified by arbitrary primers are useful as genetics markers. Nucleic Acids Res 1990;18:6531-5.
- Ellsworth DL, Rittenhouse KD, Honeycutt RL. Artifactual variation in rndomly aplified plymorphic DNA banding patterns. Biotechniques 1993;14:214-7.
- Atienzar F, Evenden A, Jha A, Savva D, Depledge M. Optimized RAPD analysis generates high quality genomic DNA profiles at high annealing temperature. Biotechniques 2000;28:52-4.
- Muralidharan K, Wakeland EK. Concentration of primer and template qualitatively affects products in random amplified polymorphic DNA-PCR. Biotechniques 1993;14:362-4.
- Coen E, Thoday J, Dover G. Rate of turnover of structural variants in the rDNA gene family of Drosophila melanogaster. Nature (London) 1982;295:564-68.
- Black W, Mustermann L. Molecular Taxonomy and systematics of arthropod vectors. En: Beaty B, Marquardt W (eds). The biology of disease vector. Colorado:University Press of Colorado, Niwot, Co. 1996.p. 438-70.
- Moeller EM, Bahnweg G, Sandermann H, Geiger HH. A simple and efficient protocol for isolation of high molecular weight DNA from filamentous fungi, fruit bodies and infected plant tissues. Nucleic Acids Res 1992;22:6115-6.
- Deng MQ, Cliver DO. Rapid DNA extraction methods and new primers for randomly amplified polymorphic DNA analysis of Giardia duodenalis. J Microbiol Methods 1999;37:193-200.
- Krsek M, Wellington EMH. Comparison of different methods for isolation and purification of total community DNA from soil. J Microbiol Methods 1999;39:1-16.
- Jaramillo C, Montaña MF, Castro LR, Vallejo GA, Guhl F. Differentiation and genetic analysis of Rhodnius prolixus and Rhodnius colombiensis by rDNA and RAPD amplification. Mem Inst Oswaldo Cruz 2001;96:1043-8.
- Breniére SF, Taveira B, Bosseno MF, Ordoñez R, Lozano-Kasten F, Magallón-Gastélum E, et al. Preliminary results of Random Amplified Polymorphic DNA among Triatominae of the phyllosoma complex (Hemiptera, Reduviidae). Mem Inst Oswaldo Cruz 2003;98:1033-938.
- Soares RPP, Barbosa SE, Borges EC, Melo Júnior TA, Romanha AJ, Dujardin JP, et al. Genetic studies of Psammolestes tertius (Hemiptera: Reduviidae: Triatominae) using male genital morphology, morphometry, isoenzymes and random amplified polymorphic DNA. Biochem Genet 2001;39:1-13.
- Dorn PL, Melgar S, Rouzier V, Gutierrez A, Combe C, Rosales R, et al. The Chagas vector, Triatoma dimidiata (Hemiptera: Reduviidae), is panmictic within and among adjacent villages in Guatemala. J Med Entomol 2002;40:436-40.
- Borges EC, Romanha AJ, Diotaiuti L. Uso do Random Amplified Polymorphic DNA (RAPD) no estudo populacional do Triatoma brasiliensis Neiva, 1911. Cad Saúde Pública 2000;16:97-100.
- Borges EC, Dujardin JP, Schofield CJ, Romanha AJ, Diotauti L. Genetic variability of Triatoma brasiliensis (Hemiptera: Reduviidae) populations. J Med Entomol 2000;37:872-7.
Recibido: 20 deagosto de 2004. Aprobado: 16 de septiembre de 2004.
Lic. Jorge Fraga Nodarse. Instituto de Medicina Tropical Pedro Kourí, Apartado Postal 601, Marianao 13, Ciudad de La Habana, Cuba. Correo electrónico: fraga@ipk.sld.cu
1 Licenciado en Bioquímica. Aspirante a Investigador.
2 Licenciada en Biología. Aspirante a Investigadora.
3 Licenciado en Biología. Investigador Auxiliar.
4 Licenciada en Biología. Investigadora Agregada.
5 Licenciada en Bioquímica. Investigadora Agregada.