Introducción
Se estima que existe 257 millones de personas con infección crónica por el virus de la hepatitis B (VHB) globalmente. Para tener una idea de la magnitud, en 2015, la hepatitis B ocasionó 887 000 muertes, la gran mayoría debido a sus complicaciones (cirrosis y el carcinoma hepatocelular) según cifras de la Organización Mundial de la Salud (OMS).1 En el 2015, los Estados Miembros de la Organización Panamericana de la Salud (OPS) aprobaron el primer Plan de acción para la prevención y el control de las hepatitis virales a escala regional, en el que se promueven políticas e intervenciones de salud pública encaminadas a eliminar las hepatitis virales en los Estados Miembros hacia el 2030. En el caso de la hepatitis B para lograr esta meta es fundamental contar con directrices para la prevención, el diagnóstico y la atención médica de la infección por el VHB.2,3
Una de las herramientas moleculares para el estudio de la infección por el VHB es la detección cuantitativa del ADN del VHB o carga viral. La carga viral es imprescindible para definir la existencia de replicación del VHB en pacientes con un perfil serológico compatible con infección crónica, para investigar la presencia de infección oculta por el VHB (HBsAg negativo) y para monitorear la respuesta al tratamiento antiviral y la definición de respuesta virológica.4 Además, es fundamental para estudiar a las embarazadas con HBsAg positivo con el propósito de instaurar terapia antiviral. Lo anterior coadyuvaría a la eliminación de la transmisión materno-infantil de la hepatitis B y el logro de las metas de impacto 2020 y hacia la meta del 2030.
El estuche SUMASIGNAL VHB (un paso) propuesto por el Centro de Inmunoensayo (CIE), Cuba, es un método cuyo principio se basa en una reacción en cadena de la polimerasa en tiempo real (RCP-TR). Dicho método fue evaluado internamente en el CIE, resultando ser un sistema de RCP-TR simple, rápido, específico, preciso y exacto para la cuantificación del ADN del VHB en suero y plasma humanos, útil para el diagnóstico y el manejo de la enfermedad.5 El presente trabajo tiene como objetivo evaluar el desempeño analítico de SUMASIGNAL VHB (un paso).
Métodos
Muestras clínicas
Se conformó un panel de sueros, que se conservó en condiciones adecuadas (-20 ºC). El panel comprendía 80 muestras residuales de sueros con las siguientes características: 20 muestras negativas al HBsAg y al ADN-VHB; 10 muestras negativas al HBsAg y positivas a otros agentes infecciosos que pueden co-infectar junto al VHB o poseen cuadros clínicos similares como: virus de la hepatitis A (IgM anti-VHA, 2 muestras), virus de la hepatitis C (VHC, 2 muestras), virus de la hepatitis E (VHE, 1 muestra), virus de la inmunodeficiencia humana (VIH, 2 muestras), citomegalovirus (CMV, 2 muestras) y virus dengue (IgM anti-dengue, 1 muestra). Además, 50 muestras positivas al ADN-VHB analizadas previamente por los estuches COBAS® TaqMan® HCV Test v2.0 (Roche), Artus RG HBV PCR kit (Qiagen) y una RCP-TR estandarizada y evaluada en el Laboratorio de referencia de hepatitis virales del Instituto de Medicina Tropical Pedro Kourí (IPK),6 dichas muestras abarcaban todo el rango lineal de cuantificación del ensayo SUMASIGNAL VHB (un paso) desde 5,0E+02 a 5,0E+09 UI/mL.
Consideraciones éticas
En el presente estudio se utilizaron muestras residuales de suero que fueron enviadas al laboratorio del IPK y del Instituto de Gastroenterología para diagnóstico molecular del VHB y que se encontraban adecuadamente conservadas en congelación. Se utilizaron muestras enumeradas respetando la identidad de estas. No se utilizaron con otros fines y los resultados obtenidos formaron parte del estudio de validación solamente.
Estándar Internacional
Se utilizo el Tercer Estándar Internacional de la OMS para las técnicas de amplificación de ácidos nucleicos del VHB (National Institute for Biological Standards and Control, NIBSC código 10/264) (EI) para determinar la exactitud y linealidad. Este reactivo es de genotipo A2 serotipo adw2 y posee una concentración de 850 000 UI/mL (~5,93 log10 UI/mL), cuando se reconstituye en 0,5 mL de agua libre de nucleasas.7
Evaluación de la prueba inicial de desempeño
Se determinaron las características del ensayo tales como especificidad clínica, especificidad analítica (reactividad cruzada), rango lineal o linealidad y exactitud, precisión intraensayo y comparación con un ensayo de referencia.8
Especificidad clínica o diagnóstica: Se determinó con el análisis de muestras negativas al HBsAg y al ADN-VHB.
Especificidad analítica o reactividad cruzada: Se determinó con el análisis de las muestras negativas al HBsAg y positivas a otros agentes infecciosos que pueden co-infectar junto al VHB o que poseen cuadros clínicos similares.
Linealidad y exactitud: Se utilizó el EI diluido en 0,250 mL de agua libre de nucleasas, para obtener una concentración de 1700E+06 UI/mL. Se realizaron diluciones seriadas en base 10 hasta 1:1000, 5 réplicas de cada uno y se incluyó además el estándar puro. Este experimento se realizó en un mismo día. Los resultados en UI/mL se transformaron en Log10 para su procesamiento final.
Precisión: Fue calculada con el EI en cuatro diluciones, desde 1 700 000 UI/mL hasta 1700 UI/mL para asegurar diferentes concentraciones de ADN (alta, media y baja) con 3 réplicas de cada una.
Estudio de comparación entre SUMASIGNAL VHB (un paso) y un ensayo de referencia (Artus® HBV RG PCR Kit): Se analizaron por ambos métodos paralelamente 50 muestras positivas al ADN-VHB que abarcasen todo el rango lineal de cuantificación del ensayo SUMASIGNAL VHB (un paso). De ellas, el 38 % de las muestras tenía una carga viral (CV) en Log10 entre 3 y 5, 34 % estaban entre >5 y 7 y el 28 % eran de Log10> 7.
Ensayo a evaluar
SUMASIGNAL VHB (un paso)
Este estuche diagnóstico es una prueba in vitro de amplificación de ácidos nucleicos para la cuantificación del ADN del VHB en suero o plasma humano. El ensayo utiliza una solución de lisis que permite la lisis rápida de la partícula viral y la liberación del ADN del VHB a partir de suero o plasma humano. A través de una RCP-TR es posible realizar la cuantificación del ADN del VHB por variaciones en la fluorescencia que se producen al ocurrir dicha reacción. En la mezcla de PCR están presentes dos cebadores que flanquean una región conservada en el genoma del VHB (gen S) y una sonda fluorescente específica que permite la detección cuantitativa. Se elimina la extracción del ADN de las muestras pues solo es necesario añadir el suero o plasma humano directamente al tubo de PCR y mezclarlo con la solución de lisis para amplificar posteriormente el ADN liberado. El sistema utiliza además un control interno para detectar posibles inhibiciones de la RCP, evitando la aparición de resultados falsos negativos. Incorpora también un marcador de referencia (ROX) para eliminar las variaciones que pudieran existir entre los diferentes tubos a través del análisis automático de la relación entre la fluorescencia de las muestras y del marcador ROX, lo que permite una cuantificación más exacta. El sistema de detección utiliza la enzima Uracil-N-Glicosilasa (UNG), que degrada completamente los restos del producto amplificado de reacciones anteriores, lo que evita la obtención de resultados falsos positivos. El límite de detección de este ensayo es de 377,47 UI/mL utilizando el estándar internacional de la OMS.5
Cada tubo de reacción de la RCP-TR contó con un volumen final de 50 μL que consistió en 40 μL de una mezcla de RCP lista para su uso y 10 μL de cada una de las referencias cuantitativas (A-D) los controles positivos y negativos o las muestras con la solución de lisis después de incubadas durante 10 min a temperatura ambiente (20-25 ᵒC). Los parámetros de la reacción consistieron en 2 min. a 50 ᵒC y 2 min a 94 ᵒC, seguido de 45 ciclos repetitivos de 94 ᵒC por 5 s y 57 ᵒC por 30 s. El programa de la PCR-TR se configuró en el equipo SLAN 96-P (Shanghai Hongshi Medical Technology Co., Ltd, Shanghai, China) según manual del estuche.
Ensayos de referencia
Para la extracción del ADN del VHB se utilizó el estuche QIAamp DNA Blood Mini Kit (Qiagen, Alemania) de forma manual, según instrucciones del fabricante. Para la cuantificación del ADN se utilizó el estuche artus® HBV RG PCR Kit (Qiagen, Alemania) según instrucciones del fabricante. El programa de la PCR-TR se configuró en el equipo Rotor Gene Q (Qiagen, Alemania) según manual del estuche.
Análisis estadístico
Para el análisis de los resultados se confeccionó una base de datos en Microsoft® Excel. Se calcularon medidas de estadística descriptiva tales como la distribución de frecuencias, promedios y la desviación estándar. Se calculó el coeficiente de correlación de Pearson (r) y el análisis de regresión lineal para los ensayos de exactitud y linealidad con el EI. Para evaluar la precisión intraensayo se determinó el promedio, la desviación estándar (DE) y el coeficiente de variación (CV= (DE/Promedio) x 100) para cada concentración evaluada del EI dentro de un mismo ensayo. Para comparar el método a evaluar y un ensayo de referencia se determinó el coeficiente de correlación.
Resultados
La especificidad clínica y especificidad analítica o reactividad cruzada del ensayo SUMASIGNAL VHB (un paso) fueron del 100 %. En el ensayo de linealidad y exactitud con el EI se alcanzaron resultados satisfactorios para una r2= 0,9954 y una diferencia de los log10 esperado vs. observado de menos de 0,5 en todas las diluciones, respectivamente (Fig. 1, Tabla 1).
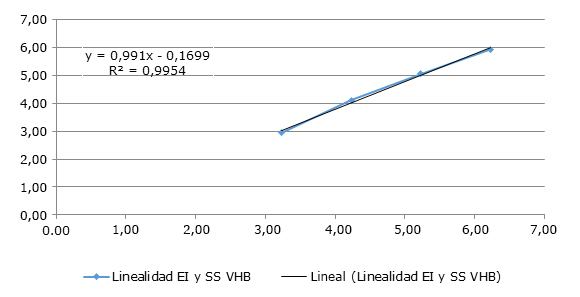 Los resultados están expresados en Log10. El coeficiente de regresión r2= 0,9954 comprueba la linealidad en el rango analizado.
Los resultados están expresados en Log10. El coeficiente de regresión r2= 0,9954 comprueba la linealidad en el rango analizado. Fig. 1 Resultados del experimento de linealidad entre los resultados esperados del EI y los obtenidos con SUMASIGNAL VHB (un paso).
Tabla 1 Comparación entre los títulos esperados de las diluciones del EI y los títulos obtenidos con el estuche SUMASIGNAL VHB (un paso)
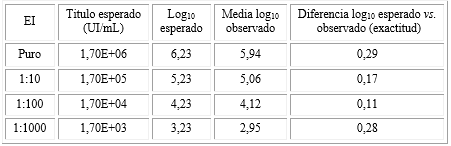
EI: estandar internacional
El coeficiente de correlación entre ambos resultados fue de r= 0,9977.
En la tabla 2 se muestran los resultados de la evaluación de la precisión intraensayo, donde el CV fue de menos del 3 %.
Tabla 2 Precisión intraensayo del ensayo SUMASIGNAL VHB (un paso) utilizando el EI para cuantificar el ADN del VHB

EI: estándar internacional, DE: desviación estándar, CV: coeficiente de variación.
Se cuantificó el ADN-VHB en 50 muestras por ambos métodos, el objeto de evaluación y el de referencia; 25 (50 %) muestras obtuvieron diferencias Log10≤ 0,5; 13 (26 %) muestras obtuvieron una diferencia de logaritmo de entre 0,51 y 1; 7 (14 %) muestras entre ˃1 y 1,5; y 5 (10 %) muestras ˃1,5 (Fig. 2). En el diagrama de dispersión (Fig. 3) se muestran los Log10 de las concentraciones de ADN obtenidas por SUMASIGNAL VHB (un paso) y Artus HBV RG PCR kit. Se obtuvo un coeficiente de correlación r= 0,8882.

Fig. 2 Comportamiento de las diferencias entre los ensayos SUMASIGNAL VHB (un paso) y Artus HBV RG PCR (Log10).
Discusión
En el presente estudio se evaluó el desempeño analítico del ensayo SUMASIGNAL VHB (un paso), un método propuesto por el CIE para cuantificar el ADN-VHB basado en el principio de RCP-TR; el cual tiene como característica la eliminación del paso de extracción del ADN de las muestras, pues solo necesita añadir la misma directamente al tubo de RCP y mezclarlo con la solución de lisis para amplificar posteriormente el ADN liberado. La RCP-TR se considera el estándar de oro para la cuantificación de ácidos nucleicos. Además, es una herramienta diagnóstica poderosa que permite obtener resultados de forma rápida, reproducibles y exactos reduciendo al mínimo el riesgo de contaminaciones.9,10
La especificidad clínica y especificidad analítica o reactividad cruzada obtenidas fueron excelentes, comparable con la obtenida por Armas Cayarga y otros al evaluar internamente dicho ensayo,5 y es similar a otros ensayos de su tipo, en los que se han obtenido resultados de más del 97 %, utilizando muestras seronegativas al VHB.6,9,10,11,12
La utilización del Tercer Estándar Internacional de ADN-VHB de la OMS, reconocido como el reactivo de referencia para las técnicas de detección de ácidos nucleicos, permite obtener resultados más sólidos y exactos, y su uso ha resuelto el problema en la estandarización de los ensayos cuantitativos al unificar los resultados en UI/mL.(7,10,13 Además, ha permitido comparar y armonizar resultados entre diferentes laboratorios y diferentes ensayos y el establecimiento de límites regulatorios y guías de tratamiento.7
En el presente estudio se obtuvieron diferencias mínimas entre los log10 esperados en relación a los obtenidos, sin superar diferencias de más de 0,5 Log10 (valor de corte empleado para evaluar una diferencia clínicamente significativa) lo cual brinda seguridad en el diagnóstico, al apuntar que la variabilidad de los resultados de la prueba no pueda ser confundida como un cambio de la carga viral del paciente.8,14 Asimismo, la ecuación lineal mostró muy buenos resultados (pendiente cercana al valor de 1 e intercepto cercano al valor 0) con un coeficiente de regresión r2= 0,9954, en los que todos los puntos evaluados indicaron una línea recta, lo cual revela que conociendo uno de los valores (esperado) se puede predecir el resultado (observado) perfectamente.
Lograr una buena precisión es esencial en los ensayos de cuantificación de ácidos nucleicos. Se ha planteado que en los ensayos de RCP-TR se puede lograr un CV de 1-3 % para el valor del Ct.15 Algunos autores apuntan a que la imprecisión no debe ser mayor que el 30 %.8 El sistema evaluado logró un excelente CV intraensayo, con mínima dispersión de los resultados al realizar réplicas del estándar de la OMS en diferentes concentraciones y es muy similar al obtenido por otros autores a nivel nacional e internacional.5,6,11,12,16,17
Al comparar la técnica evaluada con el ensayo de referencia, el 76 % de las muestras clínicas obtuvieron resultados con diferencias Log10≤ 1. Si bien las diferencias de 0,5 son las calificadas dentro del intervalo de tolerancia clínica, el coeficiente de correlación de Pearson fue de 0,8882, lo cual indica una fuerte correlación entre las pruebas y gran precisión utilizando 50 muestras clínicas de suero que abarcaban todo el rango de cuantificación de SUMASIGNAL VHB (un paso), lo que permite compararla con otras técnicas comerciales. Si tenemos en cuenta que estos sistemas disponibles comercialmente poseen un costo elevado, SUMASIGNAL VHB (un paso) podría ser una alternativa para los países de bajos recursos.
La técnica evaluada utiliza controles internos, que descarta la posibilidad de inhibidores en las muestras que inhiben la RCP originando falsos negativos. La inhibición de los ensayos moleculares suele ocurrir por alteraciones en el pH, la concentración de iones, o la viscosidad o por inhibición directa de la enzima polimerasa.8,18 Este es un aspecto importante a tener en cuenta ya que asegura la calidad de los resultados.
La limitación de este estuche diagnóstico consiste en su límite de detección definido en 377,47 UI/mL5 debido obviamente a su método de extracción, lo cual impide su uso como estuche de pesquisa en bancos de sangre (identificar muestras en "periodo de ventana"), para determinar casos de infecciones ocultas, en que la carga viral es usualmente baja (< 200 UI/mL) o para definir respuesta virológica en pacientes tratados con análogos de nucleos(t)idos (AN), ya que esta se define justamente con la no detección del ADN-VHB, precisándose ensayos diagnósticos de bajos límites de detección.4,19
Comúnmente se ha estimado la indicación de terapia antiviral de la hepatitis B crónica cuando los niveles de ADN-VHB> 20 000 UI/mL, entre otros marcadores bioquímicos, histológicos y factores del huésped (edad, duración de la infección, estadio de la enfermedad, entre otros). Cuba adopta igualmente este criterio para indicación de tratamiento, proponiendo como procedimiento preferencial monoterapias con AN de alta barrera de resistencia a largo plazo: Entecavir (ETV), Tenofovir alafenamida (TAF) y Fumarato de disoproxilo de tenofovir (TDF).4,19,20 No obstante, en la práctica clínica, se ha visto que algunos pacientes con hepatitis B crónica tienen niveles fluctuantes de ADN-VHB, que pueden variar desde valores indetectables hasta >2 000 000 UI/mL,21 de ahí que el monitoreo seriado de la carga viral es más importante que un único valor de corte arbitrario para la necesidad de tratamiento.19 De acuerdo con estos planteamientos, SUMASIGNAL VHB (un paso) podría usarse en la red diagnóstica identificando a los pacientes con infección crónica tributarios de tratamiento antiviral y a las embarazadas en cuya pesquisa se confirme la presencia del HBsAg.
En conclusión, el estuche SUMASIGNAL VHB (un paso) es un ensayo fácil de realizar manualmente y rápido, ya que se obtienen los resultados entre 1,5 a 2 h aproximadamente (entre procesamiento de las muestras, RCP-TR e interpretación de los resultados). Incluye reactivos de extracción de ácidos nucleicos, asegurando su uso sin necesidad de comprar reactivos comerciales de extracción. Teniendo en cuenta la validez del método para el uso previsto, el estuche SUMASIGNAL VHB (un paso) puede ser recomendado para su introducción en el diagnóstico, la vigilancia y la indicación de tratamiento en los pacientes con hepatitis B crónica.















