Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Hematología, Inmunología y Hemoterapia
versión On-line ISSN 1561-2996
Rev Cubana Hematol Inmunol Hemoter v.16 n.1 Ciudad de la Habana ene.-abr. 2000
Artículos Originales
Instituto de Hematología e InmunologíaEstudio molecular del gen MLL en 30 pacientes con leucemias agudas
Lic. Raquel Levón Herrera, Dr. Alejandro González Otero, Dr. Edgardo Espinosa Martínez, Dr. Porfirio Hernández Ramírez y Lic. Gisela Martínez AntuñaRESUMEN
Los reordenamientos del gen MLL en la banda cromosómica 11q23 son frecuentes en leucemias agudas (LA) en niños y en las LA secundarias desarrolladas después de la terapia con inhibidores de la enzima topoisomerasa II. En menor medida también se aprecia en adultos con LA. La presencia de estos reordenamientos se considera un indicador de mal pronóstico asociado con resultados clínicos desfavorables, por ello es muy importante realizar su determinación en las LA. En este trabajo mostramos los resultados preliminares de la introducción del estudio del gen MLL en nuestro país mediante la técnica de Southern. Analizamos ADN de 30 pacientes con LA, incluidos niños y adultos, que en el momento del estudio se encontraban al debut o en recaída. El estudio molecular se realizó con la sonda FA4, que es un inserto genómico del gen MLL. Sólo uno de los 30 pacientes mostró bandas de reordenamiento con 2 enzimas de restricción diferentes, el resto mostró el gen MLL en configuración germinal. Es interesante destacar que el paciente con el reordenamiento era un niño con leucemia mieloblástica aguda subtipo M5b, lo cual concuerda con la literatura, donde se describe que estos reordenamientos están estrechamente correlacionados con los subtipos mielomonocítico (M4) y monocítico (M5) de leucemia mieloide aguda (LMA).Descriptores DeCS: REORDENAMIENTO GENICO; ENFERMEDADES HEMATOLOGICAS; LEUCEMIA LINFOCITICA.
Las células malignas de muchos pacientes que padecen de leucemia, linfoma u otro neoplasma hematológico, presentan anormalidades cromosómicas adquiridas en forma clonal, que muchas veces están estrechamente o únicamente asociadas con subtipos clínicamente distintos de leucemia o linfoma, y que influyen en la respuesta temprana al tratamiento.
La detección de estas anormalidades recurrentes, puede ser útil para establecer el diagnóstico correcto, para el seguimiento de la enfermedad y en algunos casos tienen valor pronóstico. Por otra parte, estas anormalidades pueden brindar también información sobre la patogénesis de la enfermedad.
En este trabajo estudiamos el gen MLL (del inglés meloid-lymphoidleukemia o mix-lineage-leukemia), también conocido como ALL-1 HRX o Htrx-1, por su homología con el gen Trithorax de Drosophila. La alteración de este gen es un evento patogénico recurrente en leucemias con aberraciones en la banda cromosómica 11q23 (translocaciones recíprocas, deleciones intersticiales, duplicaciones parciales, inversiones u otras) que se caracteriza por una extrema diversidad, ya que se observa con un gran número de parejas cromosómicas diferentes (al menos 25) y en una gran variedad de leucemias, entre las que se incluyen la leucemia mieloide aguda (LMA), la leucemia linfoide aguda (LLA), la leucemia mieloide crónica en crisis blástica (LMCcb), las leucemias agudas (LA) secundarias inducidas por tratamiento con inhibidores de la enzima topoisomerasa II y otras entidades como la enfermedad de Hodgkin, los linfomas no hodgkinianos y los síndromes mielo-displásicos (SMD).1-6 En todas estas enfermedades, como consecuencia de las diferentes translocaciones, el gen MLL se une a distintos genes y origina un nuevo gen híbrido o quimérico.
Este gen codifica una proteína de 431 kD que se localiza en estructuras nucleares y se expresa en muchos órganos y tejidos humanos, incluyendo cerebro, cerebelo, corteza cerebral, cordón espinal, colon, hígado, bazo, timo, amígdalas, riñón, corazón, glándula tiroides, pulmón, testículos y músculo esquelético.7,8
Se ha demostrado que el gen MLL es un factor de transcripción activo durante el período embrionario que está involucrado en la regulación de los genes Hox, los cuales están implicados tanto en el patrón de desarrollo como en la diferenciación hematopoyética.7,9
Las leucemias agudas con alteraciones cromosómicas en 11q23, que involucran al gen MLL, tienen características biológicas inusuales y manifestaciones clínicas agresivas que les confieren un peor pronóstico.10,11 Son particularmente frecuen-tes en niños menores de un año, con una incidencia del 75 % o más en la LA infantil, mientras que en niños mayores de un año y en adultos su incidencia es del 5 %.9,12 También están asociadas con indicadores de alto riesgo de fallo al tratamiento como: gran masa tumoral, demostrada por hiperleucocitosis (>100x109/L); infiltración extramedular frecuente, con hepato-esplenomegalia y toma del sistema nervioso central. Se originan en una célula mul-tipotente común, de ahí que pueden ser de derivación mieloide o linfoide y la evolución clínicohematológica es muy desfavorable, ya que estas leucemias responden pobremente a la terapia convencional y están asociadas con un riesgo incrementado de recaída cuando se comparan con leucemias similares que carecen de anormalidades en MLL.13,14
Estas características nos motivaron a introducir en nuestro país el estudio molecular de este gen mediante la técnica de Southern blot, para usarla como un indicador de pronóstico en las LA, y por lo tanto, como una guía para que el hematólogo adopte, oportunamente, las estrategias terapéuticas más adecuadas con este subgrupo de pacientes. En el presente trabajo mostramos los resultados preliminares de dicho estudio.
MÉTODOS
Pacientes
Iniciamos el estudio con ADNs criopreservados, obtenidos a partir de pacientes con LA que en el momento de la toma de la muestra se encontraban al debut o en recaída. Posteriormente, el estudio se ha seguido realizando a pacientes con LA recién diagnosticados.Se estudió un total de 30 pacientes con diagnóstico de LA, de los cuales 27 (90 %) eran niños (18 LLA, 5 LMA, 2 LA híbridas y 2 LA indiferenciadas) en edades que oscilaron entre 0,9 y 14 años, con un promedio de 5,9 años, y 3 casos (10 %) eran adultos (2 LMA y 1 LA híbrida) con edades entre 23 y 27 años y un promedio de 25,6 años. En el momento del estudio, 22 se encontraban al debut y 8 en recaída.
De las 18 LLA estudiadas, 17 eran de estirpe B y una era de estirpe T. Las 7 LMA estudiadas se distribuyeron según la clasificación FAB de la siguiente forma: 3 M2, 1 M4, 2 M5 y 1 M7.
Técnica de SOUTHERN BLOT
El ADN se obtuvo a partir de leuco-citos de muestras de médula ósea o sangre periférica según el método convencional.15 Ocho microgramos de ADN de alto peso molecular fueron digeridos con las siguientes enzimas de restricción: Hind III, Bam HI, Eco RI y BGI II; sometidos a electroforesis en geles de agarosa al 1 %; desnaturalizados por 20 minutos en NaOH 0,4 N; transferidos con una solución 10x SSPE a filtros de nylon cargados positivamente del tipo Hybond-TM-N+ (Amersham International plc), fijados al filtro durante 2 horas en horno a 80 °C y prehibridados al menos por 2 horas a 65 °C en una solución que contiene los búfferes 5xSSPE, 5xBFP, 0,5 % SDS y 3 mg/mL de ADN de esperma de salmón desnaturalizado. A continuación fueron hibridados durante toda la noche, en la misma solución, con una sonda de ADN marcada previamente por el método de nick translation.15 Dicha sonda fue donada gentilmente por el Dr. G.Cimino de la Universidad La Sapienza de Roma, y es un inserto genómico de 480 pb del locus MLL9 que nos permite explorar una región de 13,7 kb entre 2 sitios de restricción para la enzima Hind III. Esta sonda designada como FA4 está localizada entre los exones 8 y 9 del gen MLL (fig. 1).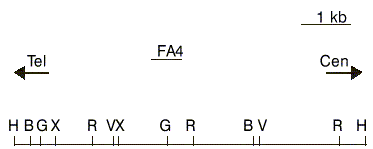
FIG. 1. Mapa de restricción parcial del locus MLL con la localización de la sonda FA4 usada
Los filtros fueron lavados 2 veces durante 5 minutos en 2xSSPE a temperatura ambiente y posteriormente fueron lavados en una solución 0,2xSSPE; 0,1 % SDS durante 30 minutos a 65 °C y expuestos a autorradiografía con placas de rayos X del tipo Hiperfilm -MP (Amersham International plc) y con pantalla intensificadora a -70 °C por una semana antes de realizar el revelado.
RESULTADOS
En la tabla se muestran algunos datos clínicos y los resultados del estudio molecular de los pacientes. Sólo uno de los 30 pacientes (3,3 %) mostró una banda de reordenamiento al menos en 2 digestiones con 2 enzimas diferentes (Hind III y Bam HI), lo cual excluye la posibilidad de existencia de una digestión parcial o de un polimorfismo en la longitud de los fragmentos de restricción (RFLP). Los casos restantes mostraron el gen MLL en configuración germinal con las diferentes enzimas utilizadas.En la figura 2 se observan los patrones de restricción representativos del estudio molecular del gen MLL con las enzimas Bam HI y BgI II y en la figura 3 se pueden ver además con las enzimas Hind III y Eco RI.
| | | |||||||
| No. | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
| | | | | | | | | |
H: Hind III; B: Bam HI; E: Eco RI; Bg: Bgl II; G: germinal; ind: indiferenciado; ND: no determinado; hib: híbrida; R: reordenado; NU: no útil.

FIG. 2. Patrones de restricción representativos con las enzimas Bam HI y Bgl II. La flecha indica la banda
de reordenamiento.

FIG. 3. Patrones de restricción representativos con las enzimas Eco RI, Bam HI e Hind III.
DISCUSIÓN
La frecuencia total del reordenamiento del gen MLL encontrada por nosotros (3,3 %) es muy similar a la obtenida por un grupo de investigadores argentinos (Hospital Garrahan, Buenos Aires) que encontraron en un total de 577 pacientes con leucemia aguda (453 LLA y 124 LMA), 23 pacientes con alteración molecular en la banda 11q23, lo que representa el 3,9 % del total de pacientes estudiados.16Es interesante destacar que el paciente con reordenamiento del gen MLL (No. 17) era un niño con una LMA subtipo M5, lo cual concuerda con la literatura, donde se señala que estos reordenamientos están estrechamente correlacionados con los subtipos mielomonocítico (M4) y monocítico (M5) de LMA.1,4,5
Era de esperarse la ausencia de reordenamiento en la LLA tipo T (No. 6) y en las LMA subtipo M2 (Nos. 18, 19 y 27), ya que la alteración de este gen en estos tipos de leucemia es inusual.5,6,12,14,17,18
Las LA con MLL reordenado y mal pronóstico en ocasiones suelen ser indiferenciadas o híbridas.5,19 En nuestros pacientes incluidos en estas categorías, sin embargo, no se detectó reordenamiento del gen MLL (Nos. 3 y 30) y (Nos. 4,5,11), respectivamente.
Consideramos que es de gran importancia disponer de esta técnica, ya que nos permite determinar la presencia de reordenamientos del gen MLL incluso en pacientes donde existen alteraciones submicroscópicas en dicha región y que tienen un cariotipo aparentemente normal.11 Dicha presencia constituye siempre un indicador de muy mal pronóstico en pacientes afectados por LA, independientemente de la edad o la estirpe celular de la leucemia.5 En un modelo multivariado desarrollado por Cimino y otros11 para consignar mejor la relevancia clínica y pronóstica de la configuración del gen MLL, que incluyó otros factores de riesgo conocidos como edad, sexo, conteos leucocitarios y morfología; el estado del gen MLL fue un marcador pronóstico independiente de sobrevida libre de eventos y su lesión genética mostró ser la variable más importante que afecta negativamente el pronóstico. Esto reafirma la necesidad de incluir este factor en los esquemas de clasificación de riesgo en las LA y de tener en cuenta su influencia en la elección de estrategias de tratamiento adaptadas al riesgo en este subtipo de leucemia.6,9-11,20,21
AGRADECIMIENTOS
Agradecemos al Dr. G.Cimino de la Universidad La Sapienza de Roma por habernos suministrado la sonda necesaria para la realización de este trabajo.DISCUSIÓN
La frecuencia total del reordenamiento del gen MLL encontrada por nosotros (3,3 %) es muy similar a la obtenida por un grupo de investigadores argentinos (Hospital Garrahan, Buenos Aires) que encontraron en un total de 577 pacientes con leucemia aguda (453 LLA y 124 LMA), 23 pacientes con alteración molecular en la banda 11q23, lo que representa el 3,9 % del total de pacientes estudiados.16Es interesante destacar que el paciente con reordenamiento del gen MLL (No. 17) era un niño con una LMA subtipo M5, lo cual concuerda con la literatura, donde se señala que estos reordenamientos están estrechamente correlacionados con los subtipos mielomonocítico (M4) y monocítico (M5) de LMA.1,4,5
Era de esperarse la ausencia de reordenamiento en la LLA tipo T (No. 6) y en las LMA subtipo M2 (Nos. 18, 19 y 27), ya que la alteración de este gen en estos tipos de leucemia es inusual.5,6,12,14,17,18
Las LA con MLL reordenado y mal pronóstico en ocasiones suelen ser indiferenciadas o híbridas.5,19 En nuestros pacientes incluidos en estas categorías, sin embargo, no se detectó reordenamiento del gen MLL (Nos. 3 y 30 y Nos. 4,5,11), respectivamente.
Consideramos que es de gran importancia disponer de esta técnica, ya que nos permite determinar la presencia de reordenamientos del gen MLL incluso en pacientes donde existen alteraciones submicroscópicas en dicha región y que tienen un cariotipo aparentemente normal.11 Dicha presencia constituye siempre un indicador de muy mal pronóstico en pacientes afectados por LA, independientemente de la edad o la estirpe celular de la leucemia.5 En un modelo multivariado desarrollado por Cimino y otros11 para consignar mejor la relevancia clínica y pronóstica de la configuración del gen MLL, que incluyó otros factores de riesgo conocidos como edad, sexo, conteos leucocitarios y morfología; el estado del gen MLL fue un marcador pronóstico independiente de sobrevida libre de eventos y su lesión genética mostró ser la variable más importante que afecta negativamente el pronóstico. Esto reafirma la necesidad de incluir este factor en los esquemas de clasificación de riesgo en las LA y de tener en cuenta su influencia en la elección de estrategias de tratamiento adaptadas al riesgo en este subtipo de leucemia.6,9-11,20,21
AGRADECIMIENTOS
Agradecemos al Dr. G.Cimino de la Universidad La Sapienza de Roma por habernos suministrado la sonda necesaria para la realización de este trabajo.SUMMARY
Rearrangements of MLL gen in llq23 chromosomal band are frequents in childhood type of acute leukemia (AL) and in secondary AL, developed after therapy with II topoisomerase enzyme. To a lesser extent also is seen in adults with AL. Presence of theses rearrangements is considered to be a worse prognosis indicator, associated with unfavourable clinical results, that is why it is very important to carry our its assessment in AL. In this paper authors present preliminary results from introduction of study on MLL gen in our country through Southern technique. DNA from 30 patients was analized, including children and adults, that at the very moment when study was performed, they were in the onset or in relapse. Molecular study was carried out using FA4 probe, which is a genomic insertion of MLL gen. Only one out of 30 patients, presented rearrangement band with 2 distinct restriction enzymes, remainder showed MLL gen in germinal form. It is interesting to underline that patient with rearrangement was a child presenting subtype M5b acute myeloblastic leukemia, which agree with literature showing that these rearrangements are closely correlated to subtype myelomonocytic (M4) and monocytic (M5) of acute myeloid leukemia (AML).Subject headings: GENE REARRANGEMENT; HEMATOLOGIC DISEASES; LEUKEMIA, LYMPHOCYTIC.
REFERENCIAS BIBLIOGRÁFICAS
1. Kearney L, Bower M, Gibbons B, Das S, Chaplin T, Nacheva E, et al. Chromosome 11q23 translocations in both infant and adult acute leukemias are detected by in situ hibridization with a yeast artificial chromosome. Blood 1992;80:1659-65.
2. Hunger SP, Thachuk DC, Amylon MD, Link MP, Canroll AJ, Welborn JL, et al. HRX involvement in de novo and secundary leukemias with diverse chromosome 11q23 abnormalities. Blood 1993;81:3197-203.
3. Kobayashi H, Espinosa R, Thirman MJ, Gill HJ, Fernald AA, Díaz MO, et al. Heterogeneity of breakpoints of 11q23 rearrangements in hematologic malignancies identified with fluorescence in situ hibridization. Blood 1993;82:547-58.
4. Paul HB, Chen SS, Smith FO, Arthur DC, Dumor PH, Bernstein ID. Molecular rearrangements o MLL gen are present in most cases of infant acute leukemia and are stongly correlated with monocytic (M5) or Myelomonocytic (M4) phenotypes. J Clin Invest 1994;93:429-37.
5. LeBeau MM, Larson RA. Cytogenetic and neoplasia. En: Hoffman R, Benz EJ, Shattil SJ. Hematology. Basic principles and practice. 2 ed. New York:Chrchill Livingstone, 1995:878-94.
6. Hernández P. Alteraciones moleculares en las leucemias agudas. Rev Cubana Hematol Inmunol Hemoter 1995;11:75-113.
7. Yagi H, Deguchi K, Aono A, Toni Y, Kishimoto T, Komari T. Growth disturbance in fetal liver hematopoiesis of MLL mutant mice. Blood 1998;92:108-17.
8. Butler LH, Slany R, Cui X, Cleary ML, Mason DY. The HRX protooncogene product is widely expressed in human tissues and localizes to nuclear structures. Blood 1997;89:3361-70.
9. Cimino G, LoCoco F, Biondi A, Elia L, Luciano A, Croce CM, et al. ALL-1 gene at chromosome 11q23 is consistently altered in acute leukemia of early infancy. Blood 1993;82:544-6.
10. Chen SC, Sorensen PHB, Domer PH, Reaman GH, Korsmeyer SJ, Heerema NA, et al. Molecular rearrangements on chromosome 11q23 predominate in infant acute lymphoblastic leukemia and are associated with specific biologic variables and poor outcome. Blood 1993;81:2386-93.
11. Cimino G, Rapanotti MC. Prognostic relevance of ALL-1 gene rearrangements in acute leukemias. Leukemia 1995;9:391-5.
12. Raimondi SC. Current status of cytogentic research in childhood acute lymphoblastic leukemia. Blood 1993;81:2237-44.
13. Kaneko Y, Maseki T, Takasaki N, Sakurai M, Hayashi Y, Nakazawa S, et al. Clinical and hematological characteristics in acute leukemia with 11q23 translocation. Blood 1986;67:484-7.
14. Raimondi SC, Frestedt JL, Pui CH, Downing JRm Head DR, Keasey JH, et al. Acute lymphoblastic leukemias with deletion of 11q23 or a novel inversion (11) (p13q23) lack MLL gene rearrangements and have favorable clinical features. Blood 1995;86:1881-6.
15. Maniatisatis F, Fristsh EF, Sambrook J. Molecular cloning: a laboratory manual. New York:Laboratory Cold Spring Harber, 1982.
16. Felice M, Zubizarreta P, Alfaro E, Chantada G, Gallego M, Babalardo E, et al. 11q23 rearrangements in childhood acute leukemias. Analysis of 23 patients observed in a single institution [abstract]. Blood 1996;88(Suppl 1 10 Pt 2):3345A.
17. Lukens JN. Classification and differenciation of the acute leukemias. En: Lee GR, Bithel TC, Forester J, Athens JW, Lukens JN. Wintrobe's clinical hematology. 9 ed. Pennsylvania:Lea and Febiger, 1993:1873-91.
18. Collins SJ. Pathobiology of AML. En: Hoffman R, Benz EJ. Shattil SJ. Hematology. Basic principles and practice. 2 ed. New York:Churchill Livingstone, 1995:983-91.
19. Poirel H, Rack K. Incidence and characterization of MLL gen (11q23) rearrangements in acute myeloid leukemia M1 and M5. Blood 1996;87:2496-505.
20. Rubnitz JE, Behm FG, Cursio Brint AM, Pinheiro VRP. Molecular analysis of t(11;19)breakpoints in childhood acute leukemias. Blood 1996;87:4804-8.
21. Look AT. Pathobiology of ALL. En: Hoffman R, Benz EJ, Shattil SJ. Hematology. Basic principles and practice. 2 ed. New York: Churchill Livingstone, 1995:992-1052.Recibido: 2 de abril de 1999. Aprobado: 27 de agosto de 1999.
Lic. Raquel Levón Herrera. Instituto de Hematología e Inmunología. Apartado 8070, CP 10800, Ciudad de La Habana, Cuba. Teléf: (537) 578268. Fax: (537) 338979. e mail:ihidir@hematol.sld.cu














