Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Habanera de Ciencias Médicas
versión On-line ISSN 1729-519X
Rev haban cienc méd vol.16 no.5 La Habana set.-oct. 2017
CIENCIAS BÁSICAS BIOMÉDICAS
Asociación de los polimorfismos T352C y A16974C con la lepra lepromatosa en pacientes cubanos
Association of T352C and A16974C polymorphisms with lepromatose leprosy in cuban patients
Estela Morales PeraltaI, Yadira Hernández PérezII, Kilenda Peñalver MoralesIII, Yulié Lamas TorresIV, Raisa Rumbaut CastilloV, Teresa Collazo MesaVI
IDoctor en Ciencias Médicas. Profesor e Investigador Titular. Universidad de Ciencias Médicas de La Habana. Facultad de Ciencias Médicas "10 de Octubre". Departamento de Medios Diagnósticos. La Habana, Cuba. fornaris@infomed.sld.cu
IILicenciada en Tecnología de la salud, perfil microbiología. Investigadora. Centro Nacional de Genética Médica. La Habana, Cuba. yhperez@cngen.sld.cu
IIIEspecialista Primer Grado en Genética Clínica y Medicina General Integral. Centro Nacional de Genética Médica. La Habana, Cuba. kilenda@infosol.gtm.sld.cu
IVEspecialista Primer Grado en Genética Clínica y Medicina General Integral. Centro Nacional de Genética Médica. La Habana, Cuba. ltyulie@cngen.sld.cu
VMáster en Ciencias. Especialista en Epidemiología. Profesora Auxiliar. Jefa de la Comisión Técnica de Lepra. Ministerio de Salud Pública. La Habana, Cuba. higssv@infomed.sld.cu
VIDoctora en Ciencias. Licenciada en Bioquímica. Investigadora. Centro Nacional de Genética Médica. La Habana, Cuba. tcollazo@infomed.sld.cu
RESUMEN
Introducción: La Lepra es una enfermedad infecciosa causada por el Mycobacterium leprae. Los patrones dermatoglíficos de pacientes cubanos con lepra lepromatosa mostraron indicios probatorios de que existe predisposición genética para el desarrollo de esta enfermedad, que sugiere la búsqueda de la asociación con polimorfismos moleculares, de mayor precisión. Entre estos, unos de los más estudiados son el T352C del gen del receptor de la vitamina D y el A16974C del gen IL12p40, cuya utilidad relativa depende de la población.
Objetivo: Determinar si existe asociación entre la presencia de los polimorfismos T352C y A16974C con la lepra lepromatosa en pacientes cubanos.
Material y Métodos: Se realizó un estudio observacional, analítico, de tipo caso-control de asociación genética donde se estudiaron pacientes con lepra lepromatosa y controles. Fueron identificados los genotipos relacionados con los polimorfismos T352C y A16974C en cada grupo. La prueba Chi-cuadrado de Pearson fue utilizada para determinar si los controles se hallaban en equilibrio de Hardy Weinberg, así como si existía relación entre los polimorfismos y la presencia de la enfermedad.
Resultados: Los pacientes estudiados fueron 32 para el polimorfismo T352C y 44 para el A16974C. Los controles fueron 64 y 44, respectivamente; estos se hallaron en equilibrio Hardy-Weinberg. No se detectó asociaciónentre los polimorfismos A16974C y T352C con la lepra lepromatosa.
Conclusiones: Los polimorfismos T352C y A16974C no son útiles como factor de riesgo predisponente en el grupo de pacientes cubanos con lepra lepromatosa estudiados.
Palabras claves: Lepra, polimorfismo genético, predisposición genética a la enfermedad, Cuba, equilibrio de Hardy-Weinberg, alelos.
ABSTRACT
Introduction: Leprosy is an infectious disease caused by Mycobacterium leprae. Dermatoglyphic patterns of Cuban patients with lepromatose leprosy showed evidential signs of the existence of genetic predisposition to the development of this disease, which suggests a search for the association with molecular polymorphisms of higher degree of accuracy. Among them, some of the most studied are the T352C vitamin D receptor gene and the A16974Cof the IL12p40 gene, which relative usefulness depends on the population.
Objective: To determine whether there is an association between the T352Cand A16974C polymorphisms with lepromatose leprosy in Cuban patients.
Material and methods: An observational analytical case-control type genetic association study was conducted where patients with lepromatose leprosy and controls were studied. Genotypes related to T352Cand A16974C polymorphisms were identified in each group. Pearson´s chi square test was used to determine whether the controls were in Hardy-Weinberg equilibrium, and also whether there was a relation between polymorphisms and the presence of diseases.
Results: There were 32 patients under study for T352C polymorphism, and 42 for A16974C. The controls were 64 and 44, respectively; and these were in Hardy-Weinberg equilibrium. No association between T352Cand A16974C polymorphisms with lepromatose leprosy was detected.
Conclusions: T352Cand A16974C polymorphisms are not useful as a predisposing risk factor in the group of Cuban patients with lepromatose leprosy studied.
Keywords: Leprosy, genetic polymorphism, genetic predisposition to disease, Cuba, Hardy-Weinberg equilibrium, alleles.
INTRODUCCION
La lepra es una enfermedad infecciosa causada por el Mycobacterium leprae (ML). En Cuba se han logrado reconocidos avances en su prevención y control. A partirde 1993, cuando se alcanzó una reducción de su tasa de prevalencia por debajo de uno por cada 10 000 habitantes, se excluyó como problema de salud.1,2 Sin embargo, esta enfermedad aún no ha sido eliminada en nuestro país, y se impone la búsqueda de nuevas medidas preventivas.3,4
Se ha comprobado que menos de 10% de los infectados con ML padecen lepra. Incluso entre los afectados, su cuadro clínico puede diferir de un paciente a otro; desde la forma tuberculoide a la lepromatosa, esta última la más grave. Esto se ha explicado por interacciones complejas entre factores genéticos predisponentes -incluyendo el genotipo de uno o más loci- y una variedad de eventos ambientales que provocan, aceleran o exacerban el desarrollo de la enfermedad.5,6,7
La identificación de tales factores genéticos permitiría una mejor comprensión del papel que estos desempeñan en la predisposición a la lepra y, especialmente, el diseño de medidas preventivas. Ello se puede lograr con la aplicación de estudios genéticos para identificar variantes genotípicas asociadas con la enfermedad que incluyen desde herramientas semiológicas, como los dermatoglifos, hasta la aplicación de técnicas de Biología Molecular.8-12
A través del estudio de los dermatoglifos de pacientes cubanos con lepra lepromatosa (LL) fueron hallados indicios probatorios de predisposición genética para el desarrollo de esta enfermedad.13
La disponibilidad de técnicas moleculares ha estimulado la búsqueda de variantes polimórficas asociadas con la susceptibilidad, o la resistencia, a la lepra, de mayor eficacia que las semiológicas.14,15 Entre los polimorfismos más estudiados se incluyen: el T352C del gen del receptor de la vitamina D (RVD) y el A16974C, del gen IL-12p40, que codifica para la interleucina-12.16-18
El gen RVD codifica un factor de transcripción, que a través de la activación de la vitamina D, modula diversos procesos biológicos, incluyendo la función inmune.19
Mientras, el gen IL-12p40, codifica la síntesis de la interleucina-12, clave en la promoción de la inmunidad celular y, por tanto, en la resistencia inicial a infecciones producidas por micobacterias, entre ellas el ML.
Los resultados de los estudios de asociación de estos dos polimorfismos con la LL han sido contradictorios.20 Ante tal discrepancia es importante determinar qué ocurre en nuestro medio, a fin de disponer de una herramienta más precisa para la identificación de personas cubanas que presenten un riesgo elevado de desarrollar esta enfermedad en Cuba, al presentar predisposición genética; a fin de proponer medidas preventivas en aquellos expuestos al agente.
OBJETIVO
Determinar si existe asociación entre la presencia de los polimorfismos T352Cy A16974C con la lepra lepromatosa en pacientes cubanos.
MATERIAL Y MÉTODOS
Se realizó un estudio analítico tipo caso-control de asociación alélica, desde enero de 2013 hasta junio del 2016, en dos etapas; en cada una de las cuales se estudió uno de los polimorfismos mencionados.
El universo de los casos quedó constituido por los pacientes con diagnóstico de lepra lepromatosa registrados en el programa nacional de lepra del Ministerio de Salud Pública.
Los casos se seleccionaron siguiendo los siguientes criterios:
Criterios de inclusión
Individuos diagnosticados con LL, según la clasificación operacional de la OMS, basada en el número de lesiones con trastornos de la sensibilidad, con 5 años de evolución21 y que dieran su disposición de colaborar con la investigación, lo cual quedó demostrado a través de la firma del consentimiento informado.
Criterios de exclusión
Pacientes con LL que padecieran otras enfermedades en las que se ha demostrado vinculación con los polimorfismos, que fueron objeto de esta investigación (enfermedad de Crohn, colitis ulcerativa, esclerosis múltiple, Diabetes Mellitus tipo 1, tuberculosis, malaria cerebral fatal y/o espondilitis anquilosante, cáncer de próstata, urolitiasis grave y vitiligo). Además de pacientes menores de 18 años; y aquellos pacientes con LL que tuvieran otro familiar afectado incluido en el estudio; para evitar sesgos de inclusión de personas con constitución genética semejante.
Criterio de salida
Quienes manifestaron su deseo de retirar su participación en la investigación, cualquiera que fuera el momento, aún después de haber dado su autorización previa.
El universo de los controles se obtuvo del banco de ácido desoxirribonucleico (ADN) del Centro Nacional de Genética, todos provenientes del estudio poblacional de longevidad.Para su selección se tuvo en cuenta que no presentaran lepra ni alguna de las enfermedades vinculadas a los polimorfismos (que aparecen en el criterio de exclusión número 1); así como el hecho de que, en el consentimiento informado del estudio referido, aceptaron que sus muestras podrían ser utilizadas en investigaciones futuras.
Fueron identificados los alelos de cada polimorfismo según se describe:
- Alelo T, cuando existía el polimorfismo T352C del gen RVD, en caso contrario se consideró el alelo t.
- Alelo A, cuando se identificó el polimorfismo A16974C del gen IL-12p40, alelo C, cuando no estuvo presente.
Se tuvieron en cuenta las siguientes variables:
- Genotipo relacionado con el polimorfismo T352C del gen RVD
TT u homocigótico para el polimorfismo: cuando solo presentó el alelo T
tt o negativo: si apareció únicamente el alelo t
Tt o heterocigótico: si se detectaron ambos alelos.
- Genotipo relacionado con el polimorfismo A16974C del gen IL-12p40
AA u homocigótico para tal polimorfismo: cuando se observó solo el alelo A
CC o negativo: si apareció únicamente el alelo C
AC o heterocigótico: si se detectaron ambos alelos.
A todos los casos se les extrajo 10 mL de sangre periférica, colectada en 250 µL de solución de EDTA (56 mg/mL). El ADN se obtuvo de los leucocitos, a través de la técnica de precipitación salina.22
Los procederes utilizados para identificar los polimorfismos T352C y A16974C se basaron en los métodos de reacción en cadena de la polimerasa (PCR) descritos por los grupos liderados por Taylor y Seegers, respectivamente,22-24 con las siguientes condiciones:
Por cada muestra se preparó un volumen total de reacción de 25 μL, compuesto por: ADN genómico 100 ng, 0,75 U Taq ADN polimerasa, 0,75 μL de cada cebador descrito en la tabla 1 (10 pM/μL), 2,5μL dNTP (1 mM), 1,5 μL MgCl2 (15 mM), buffer 1X de la enzima Taq Pol: 2,5 μL y H2O: volumen suficiente para completar 25 μL por muestra. De forma comprobatoria, como control de contaminación, se utilizó una muestra "blanco" que contenía todos los reactivos, excepto el ADN.
Las amplificaciones se realizaron en un termociclador MJ Research del siguiente modo: desnaturalización primaria (10 min a 94 ºC), seguida de 30 ciclos de: desnaturalización (30 s a 94 ºC), hibridación (30 s a 65 ºC) y elongación: 30 s a 72 ºC. Extensión final 72 °C por 10 minutos.
A fin de comprobar cada amplificación, se hizo una corrida electroforética en gel de agarosa a 2 %, teñidos con bromuro de etidio y buffer TBE 0,5 X, a voltaje fijo de 250 V. Los fragmentos obtenidos fueron de 740 pares de bases (pb) y 1046 pb, de acuerdo correspondieran a los genes RVD y IL-12p40, respectivamente.
Estos se sometieron a digestión con la enzima de restricción Taq I bajo las condiciones del fabricante, que recomiendan que se utilice: 10 µl del producto de la PCR, 2 µl (20U) de la enzima Taq I, 3.5 µl del buffer B y 19.5 µl de H2O.
La incubación se llevó a cabo a 65°C durante toda la noche.
Al producto del proceder anterior se sometió a una corrida en gel de agarosa a 2% durante 30 minutos a 250 volts. Los genotipos que se obtuvieron, relacionados con los polimorfismos, fueron:
- Para el polimorfismo T352C del gen del receptor de la vitamina D:
TT (bandas de 495 y 245 pb), Tt (bandas de 495, 290, 245 y 205 pb) y tt (las bandas fueron 290, 245 y 205 pb).
- Relativos al polimorfismo A16974C:
Fragmento de 1046 pb íntegro, se correspondió con el genotipo homocigótico para el alelo A. En el caso del genotipo homocigótico para el aleloC se observaron dos fragmentos, uno de 906 y otro de 140 pb y los tres fragmentos (1046, 906 y 140 pb) para el heterocigótico.
Procesamiento, análisis, resumen y presentación de la información
Las frecuencias de cada alelo fueron calculadas a partir de los genotipos resultantes de los estudios por PCR. Para definir que los grupos controles se encontraban en equilibrio génico de Hardy-Weinberg (EHW) se utilizó la prueba Chi cuadrado de Pearson. Este mismo proceder estadístico se utilizó para relacionar los genotipos de cada polimorfismo con la enfermedad. El nivel de significación estadístico utilizado fue de α=0,05.
Aspectos Bioéticos
Esta investigación se suscribió a los acápites de la Declaración de Helsinki,25 que establece los principios éticos para las investigaciones médicas en seres humanos y cumplió las normas éticas para la protección de la información genética de los ciudadanos cubanos.26 Fue aprobada por el Comité de Ética de la Investigación del Centro Nacional de Genética Médica.
RESULTADOS
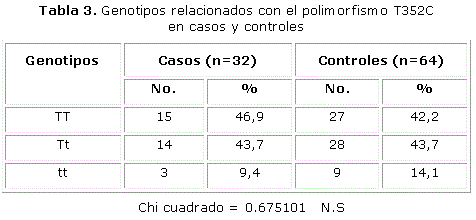
En el estudio del polimorfismoT352C se incluyeron 32 casos y 64 controles.
En la Tabla 2 se muestra que las frecuencias genotípicas relacionadas observadas con el polimorfismo T352C en controles, se corresponden con las esperadas, según la Ley de Hardy Weinberg.
En la Tabla 3 se observan los genotipos relacionados al polimorfismo T352C en pacientes con LL y controles. Aunque el genotipo TT (homocigótico para este polimorfismo) fue el más encontrado en los casos, no tuvo una diferencia notoria con el hallado en los controles. (p=.785785 Chi cuadrado = 0.675101).
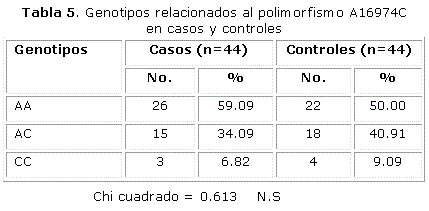
Para el polimorfismo A16974C del gen IL-12p40, el grupo de casos estuvo constituido por 44, al igual que el de los controles.
En la Tabla 4 se nota que las frecuencias observadas se correspondieron con las esperadas, para los genotipos relacionados con este polimorfismo en el grupo control, cumpliendo de este modo el equilibrio de Hardy Weinberg.
En la Tabla 5 se muestran los genotipos, tanto en casos como en controles, para el polimorfismo A16974C, obsérvese que el genotipo AA u homocigótico para este polimorfismo fue el que se presentó en mayor proporción en ambos grupos, mientras el CC fue el menos frecuente, tanto en los pacientes como en los controles.
Al aplicar la prueba Chi cuadrado, no se aprecia una diferencia estadísticamente significativa.
DISCUSIÓN
Los análisis de asociación alélica, tal como el que presentamos, permiten identificar variantes genéticas o polimorfismos relacionados con enfermedades que no muestran un patrón de herencia mendeliano, incluidas las infecciosas. Tienen una base poblacional, y se realizan a través de estudios de casos y controles. Sus resultados se corresponden con la composición genética de la población donde se realizó, y solo son aplicables en ella.7
Esta es una investigación con resultados negativos, en que en los pacientes estudiados los polimorfismos T352C del gen del receptor de la vitaminaD y el A16974C del gen IL-12p40, no representaron un factor de predisposición para la lepra lepromatosa.
Los grupos controles, de ambos estudios, cumplen con los criterios de otros investigadores, al ser personas del mismo país sin la enfermedad, en su forma lepromatosa.27-29
El hecho de que los controles se encontraban en EHW, para los polimorfismos en cuestión, es un dato clave en una investigación de tipo caso-control, de estrategia poblacional de la Epidemiología Genética, tal como la desarrollada, y sirve para evaluar la calidad del estudio.30-32 Si los controles no hubiesen estado en EHW para los polimorfismos a estudiar, hubiese sugerido que ocurrió una estratificación de la población o un error de genotipificación.30
En relación con el polimorfismo T352C, hay autores que han descrito en sus estudios una mayor frecuencia tanto del genotipo TT, en los pacientes con LL, y han demostrado un evidente nexo de este polimorfismo con la LL, con lo cual sugieren que este polimorfismo permite identificar el mayor riesgo de enfermar por lepra. Así se describió en el estudio realizado en Culiacán, México, por Velarde y colaboradores, en un grupo de 71 pacientes y 144 controles.19
Sin embargo, los resultados de esta investigación difieren, lo que puede estar relacionado con que la muestra estudiada fue menor o la composición de una y otra población sean distintas.
En un estudio realizado en Brasil de 102 pacientes y 68 controles no consanguíneos, se evidenció no existir ninguna asociación entre este polimorfismo y la susceptibilidad a enfermar de lepra, lo que coincide con los hallados en esta investigación.32
Los hallazgos, relativos al polimorfismo A16974C, coinciden con los encontrados por Morahan y colaboradores que concluyeron que no existía asociación del polimorfismo A16974C del gen IL-12p40 con la enfermedad.27
Sin embargo, otros autores han observado resultados que difieren a los que acá se presentan. En un estudio realizado en la India, donde se incluyeron un total de 2 345 individuos, el polimorfismo A16974C del gen IL-12p40 se mostró en asociación con la lepra; y se halló además que el riesgo se incrementaba cuando se presentaba junto a otros factores genéticos.28 Este resultado fue similar a otro realizado en el Sudeste asiático en el que se examinaron muestras de ADN de 347 individuos.29
Esta investigación puede tener limitaciones que expliquen las diferencias observadas con otros estudios. Una de ellas es que el tamaño del grupo de casos fue pequeño, en relación con otras investigaciones, y la población de referencia no es la misma, ni tan siquiera similar a estudios que se replican.
El hecho de que la lepra no constituya un problema de salud en Cuba hace que sean pocos los casos a estudiar, especialmente los que muestran la forma lepromatosa. Además, existen diferencias étnicas entre las poblaciones, donde se identificó asociación de estos polimorfismos, con respecto a la cubana, debido a la constitución genética. De hecho, la frecuencia de estos polimorfismos varía según el origen y en algunas de ellas los polimorfismos A16974Cy T352C son útiles para identificar la susceptibilidad a adquirir lepra en otros sitios;33 en los casos estudiados en esta investigación no representaron factores de riesgo.
CONCLUSIONES
Los polimorfismos T352C y A16974Cno son utilidad como factor de riesgo predisponente en el grupo de pacientes cubanos con lepra lepromatosa estudiados. Se impone la búsqueda de otros marcadores moleculares que permitan desarrollar medidas preventivas, especialmente en la población expuesta.
REFERENCIAS BIBLIOGRÁFICAS
1. Moreira Ríos I, Moreno Díaz EN, Sotolongo Castillo A, Rivera Moreira A, Carballea Suárez Y. Enfoque de los factores de riesgo de la lepra con las determinantes sociales de la salud. Rev Cubana Higiene Epidemiol. 2014; 52(1):4-14.
2. Pastrana Fundora F, Ramírez Albajez CR, Moredo Romo E, Ramírez Ramírez H, Alemañy Díaz-Perera C. Impacto de la lepra en la historia. Folia Dermatol Cuba [Internet]. 2012; 6(1). [Consultado: 2017 Jun].Disponible en: http://bvs.sld.cu/revistas/fdc/vol6_1_12/fdc06112.htm
3. Beldarraín-Chaple E. Historical Overview of Leprosy Control in Cuba. MEDICC Rev. 2017 Jan; 19(1):23-30.
4. Ruiz-Fuentes JL, Díaz-García A, Suárez Moreno O, Torres P, Acosta Soto L. Evaluation of different samples for Mycobacterium leprae molecular detection in Cuba. Rev Cubana Med Trop [Internet]. 2015 Ago; 67(2). [Consultado: 2017 Jun 23]. Disponible en: http://scielo.sld.cu/scielo.php?script=sci_arttext&pid=S0375-07602015000200006&lng=es
5. Trautman J. A brief history of Hansen's disease. The stara. 1990; 50:3-5. [Consultado: 2 de Mayo 2017]. Disponible en: http://cdm16313.contentdm.oclc.org/cdm/compoundobject/collection/p15140coll52/id/8065/rec/1
6. Franco-Paredes, C, Hare A, Del Río C. Leprosy in Latin America and the Caribbean: Burden of Disease and Approaches for Elimination. En: Carlos Franco-Paredes, Anna Hare, Carlos del Rio. Neglected Tropical Diseases-Latin America and the Caribbean; Springer Vienna: 2015, p.175-184.
7. Nussbaum RL, Mclnnes RR, Willar D. Genética en Medicina. 8va ed. Philadelphia: Editorial Elsevier; 2016, p. 369-382.
8. Strande NT, Berg JS. Defining the clinical value of a genomic diagnosis in the era of next-generation sequencing. Annual review of genomics and human genetics. 2016; 17:303-332.
9. Pagon RA. Genetic testing: when to test, when to refer. Am Fam Physician. 2005 Jul 1; 72(1):33-4.
10. Roberts JS, Uhlmann WR. Genetic susceptibility testing for neurodegenerative diseases: ethical and practice issues. Prog Neurobiol. 2013 Nov; 110:89-101.
11. Wolfberg AJ. Genes on the Web-direct-to-consumer marketing of genetic testing. N Engl J Med. 2006; 355(6):543-5.
12. Roche PA, Annas GJ. DNA testing, banking, and genetic privacy. N Engl J Med. 2006; 355(6):545-6.
13. Fariñas Rodríguez L, Padilla Preval J, Peñalver Morales K, Rumbaut Castillo R, Brito Chávez M, Morales Peralta E. Patrones dermatoglíficos en pacientes cubanos con lepra lepromatosa. Rev Cubana Genet Comunit. 2014; 8(2):37-43.
14. Aguilar-Medina M, Escamilla-Tilch M, Frías-Castro LO, Romero-Quintana G, Estrada-García I, Estrada-Parra S, et al. HLA Alleles are Genetic Markers for Susceptibility and Resistance towards Leprosy in a Mexican Mestizo Population. Ann Hum Genet. 2017 Jan; 81(1):35-40.
15. Xiong JH, Mao C, Sha XW, Jin Z, Wang H, Liu YY, Ning Y. Association between genetic variants in NOD2, C13orf31, and CCDC122 genes and leprosy among the Chinese Yi population. Int J Dermatol. 2016 Jan; 55(1):65-9.
16. Huang D, Cancilla MR, Morahan G. Complete primary structure, chromosomal localization, and definition of polymorphisms of the gene encoding the human interleukin-12 p40 subunit. Genes Immun. 2000 Dec; 1(8):515-20.
17. Schurr E, AlcaÏs A, de Léséleuc L, Abel L. Genetic predisposition to leprosy: A major gene reveals novel pathways of immunity to Mycobacterium leprae. Semin Immunol. 2006 Dec; 18(6):404-10.
18. Cardoso CC, Pereira AC, De Sales Marques C, Moraes MO. Leprosy susceptibility: genetic variations regulate innate and adaptive immunity, and disease outcome. Future Microbiol. 2011;6(5):533-49. [Consultado: 4 Feb 2015]. Disponible en: http://www.ncbi.nlm.nih.gov/pubmed/21585261
19. Velarde FJS, Cázarez Salazar SG, Castro Velázquez R, Rendón Maldonado JG, Rangel Villalobos H. Association between the TaqI polymorphism of Vitamin D Receptor gene and lepromatous leprosy in a Mexican population sample. Salud pública Méx [Internet]. 2009 Feb; 51(1): 59-61. [Consultado: 2017 Mar 11]. Available from: http://www.scielosp.org/scielo.php?script=sci_arttext&pid=S0036-36342009000100011&lng=en
20. Online Mendelian Inheritance in Man. Leprosy in: McKusick VA. Johns Hopkins University 2007. [Consultado: 13 de Enero 2017]. Disponible en: http://www.ncbi.nlm.nih.gov/omim
21. Rodríguez Junior IA, Gresta LT, Noviello Mde L, Cartelle CT, Lyon S, Arantes RM. Leprosy classification methods: a comparative study in a referral center in Brazil. Int J Infect Dis. 2016 April; 45:118-22.
22. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res. 1988 Feb 11; 16(3):1215.
23. Taylor JA, Hirvonen A, Watson M, Pittman G, Mohler JL, Bell DA. Association of prostate cancer with vitamin D receptor gene polymorphism. Cancer Res. 1996 Sep 15; 56(18):4108-10.
24. Seegers D, Zwiers A, Strober W, Peña AS, Bouma G. A Taq I polymorphism in the 3’UTR of the IL-12 p40 gene correlates with increased IL-12 secretion. Genes Immun. 2002 Nov; 3(7):419-23.
25. General Assembly of the World Medical Association. World Medical Association Declaration of Helsinki: ethical principles for medical research involving human subjects. J Am Coll Dent. 2014 Summer; 81(3):14-8.
26. Gaceta oficial de le República de Cuba. Normas éticas para la protección de la información genética de ciudadanos cubanos; 2007. [Consultado: 6 Abril 2017]. Disponible en: www.sld.cu/galerias/pdf/sitios/genetica/resolucion_219
27. Morahan G, Kaur G, Singh M, Rapthap CC, Kumar N, Katoch K, et al. Association of variants in the IL12B gene with leprosy and tuberculosis. Tissue Antigens. 2007 Apr; 69 Suppl 1:234-6.
28. Ali S, Srivastava AK, Chopra R, Aggarwal S, Garg VK, Bhattacharya SN et al. IL12B SNPs and copy number variation in IL23R gene associated with susceptibility to leprosy. J Med Genet. 2013; 50:34-42. [Consultado: 30 May 2015]. Disponible en: http://jmg.bmj.com/content/50/1/34.abstract
29. Sodsai P, Nakkuntod J, Kupatawintu P, Hirankarn N. Distribution of cytokine gene polymorphisms in Thai population. Tissue Antigens. 2011;77:593-597
30. Lardoeyt Ferrer R. Fundamentos de genética poblacional. La Habana: Editorial Ciencias Médicas; 2016, p: 248-268.
31. Mira MT, et al. Susceptibility to leprosy is associated with PARK2 and PACRG. Nature Journal of science. [Revista en la Internet]. 2004 Feb; 427:636-640.
32. Oliveira AL, Chaves AT, Menezes CA, Guimarães NS, Bueno LL, Fujiwara RT, Rocha MO. Vitamin D receptor expression and hepcidin levels in the protection or severity of leprosy: a systematic review. Microbes Infect. 2017 Mar ;(17).
33. Santana N, Rêgo J, Oliveira JM, Almeida Lucas F, Braz Marcos LM, et al. Polymorphisms in genes TLR1, 2 and 4 are associated with differential cytokine and chemokine serum production in patients with leprosy. Mem. Inst. Oswaldo Cruz [Internet]. 2017 Apr; 112(4): 260-268. [Consultado: 2017 June 23]. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0074-02762017000400260&lng=en
Recibido: 18 de mayo de 2017.
Aprobado: 22 de Agosto de 2017.