Introducción
Las Rickettsias son un género de bacterias intracelulares obligadas transmitidas por artrópodos hematófagos como las garrapatas, piojos, pulgas y ácaros.1) En las últimas décadas ha habido un aumento de nuevas especies de Rickettsia de patogenicidad desconocida, muchas de ellas aisladas de garrapatas. Algunas, previamente fueron consideradas no patógenas; pero recientemente demostraron serlo para humanos, tal es el caso de Rickettsia slovaca, Rickettsia aeschlimannii, Rickettsia massiliae y Rickettsia monacensis en Europa. Además, R. parkeri perteneciente al grupo de las fiebres manchadas reportado por primera vez en 1939, demostró 65 años más tarde ser patógena. Estos hechos demuestran que cualquier nueva Rickettsia descrita en huéspedes invertebrados, especialmente garrapatas, debería ser considerada potencialmente patógena para los humanos.2,3)
La ecología de las infecciones causadas por las Rickettsias está determinada por factores ambientales y la presencia de vectores específicos que condicionan el establecimiento y la epidemiología en diferentes regiones del mundo.3 Las rickettsiosis son una problemática mundial, cuyo cuadro clínico se caracteriza por un síndrome febril agudo con manifestaciones cutáneas y sistémicas. La evolución puede variar desde una enfermedad febril indiferenciada a manifestaciones hemorrágicas con afectación sistémica de carácter grave. La sospecha clínica y el tratamiento oportuno modifican la evolución de la enfermedad.4)
En Latinoamérica, a pesar de que existen muy buenas descripciones de estas afecciones desde la primera mitad del siglo pasado, se puede decir que ha existido un silencio en las agendas de salud pública posterior a su estudio y descripción. Sin embargo, con el reporte de nuevos casos de infección por Rickettsia rickettsii en Centroamérica y otras zonas de Sudamérica después de muchos años sin su ocurrencia, y la emergencia de casos humanos provocados por otras especies de Rickettsia, se ha despertado un gran interés por estas infecciones, debido a que son potencialmente letales, y no distinguirlas o sospecharlas puede conducir a la muerte.5
En la actualidad se están describiendo candidatos a nuevas especies de Rickettsia, que, aunque no han probado su patogenicidad, deben considerarse potencialmente como tales, por lo que resulta fundamental una alta sospecha clínica y la instauración precoz de un tratamiento adecuado, porque el diagnóstico microbiológico puede tardar días o semanas.6
El objetivo de este estudio es detectar la presencia de Rickettsia spp del grupo de las fiebres manchadas en garrapatas colectadas en el departamento del Meta, Colombia.
MATERIALES Y METODOS
Se realizó un estudio descriptivo de corte transversal, en el cual se recolectaron garrapatas mediante un muestreo no probabilístico por conveniencia entre los años 2013 y 2014, a partir de animales domésticos (caninos, bovinos y equinos) de seis municipios del Meta (Acacias, Puerto Gaitán, Puerto López, Restrepo, Cumaral, Villavicencio). En la capital, Villavicencio, se recolectaron garrapatas en seis veredas pertenecientes al área rural (El Carmen, Servitá, Pipiral, Barcelona, Cocuy, La Cecilia) y en barrios del área urbana. La captura se realizó sobre el hospedero (fase parasítica) de forma manual y con pinzas entomológicas.
Área de estudio
El departamento del Meta está localizado en la región centro oriental de Colombia, en el pie de monte de la cordillera oriental entre 04º54’25’’ - 01º36’52’’ N, y 71º4’38’’ - 74º53’57” W (Figura 1). Esta región está comprendida en la zona de confluencia intertropical, la temporada de sequía va desde diciembre a marzo y la época de lluvias de abril a noviembre.
Para la recolección de garrapatas sobre vegetación o en vida libre (fase no parasítica), se utilizaron los métodos de arrastre y bandereo en zonas de potrero y matorrales (única por cada sitio de muestreo).7
Las garrapatas capturadas se depositaron en tubos plásticos de 1,5 ml con alcohol al 70 % y rotulados individualmente, teniendo en cuenta el hospedero animal o sitio en el cual se realizó la captura. Las garrapatas se enviaron al Instituto de Investigaciones Biológicas del Trópico (lIBT) de la Universidad de Córdoba, donde se realizó su identificación por género y especie con el empleo de claves taxonómicas fenotípicas.8) y la detección y caracterización molecular de las rickettsias.
Las garrapatas se prepararon según el protocolo diseñado por el IIBT, que consiste en sacar las garrapatas del alcohol y triturarlas con ayuda de una cuchilla estéril, colocarlas nuevamente en un tubo seco, llevarlas al baño de agua a 56ºC, durante 20 minutos, adicionar 250 µl de PBS al 1x a cada tubo con los macerados de garrapatas y volver a macerar hasta obtener un aspecto lechoso, sin dejar pedazos grandes de garrapatas. El macerado se utilizó para el procedimiento de extracción de ADN, para el cual se empleó el estuche comercial de Purelink DNA mini kit (Invitrogen, California) y se siguieron las recomendaciones del proveedor. El ADN purificado se conservó a -20 ºC hasta su uso.
Reacción en Cadena de la Polimerasa a tiempo real para detección del gen gltA de Rickettsia spp.
Las muestras obtenidas del proceso de extracción fueron analizadas por PCR en tiempo real (PCR-RT), en un termociclador LightCycler 1.5 de tres canales (530, 640 y 705) de la casa comercial Roche. Se utilizaron los iniciadores CS-5 (5’-GAG AGA AAA TTA TAT CCA AAT GTT GAT-3) y CS-6 (5’-AGG GTC TTC GTG CAT TTC TT-3), descritos por Labruna.9 Estos iniciadores son específicos para un fragmento de 147 pb del gen gltA, que se encuentra en todas las especies del género Rickettsia spp. y codifica la enzima citrato sintetasa. Adicionalmente, se empleó una sonda de hidrólisis (6-FAM-CATTGTGCCATCCAGCCTACGGT- BHQ-1), cuyo análisis de detección en la emisión de luz de la longitud de onda fue realizada en el canal de 530 del termociclador.
La mezcla de reacción de PCR se preparó a un volumen final de 20 µL. Cada reacción presentó los siguientes componentes: 4 µL de Master TaqMan (compuesta por FastStar Taq ADN polimerasa, dNTPs y MgCl2 en concentración óptima) (Roche Diagnostic, Applied Science, Mannheim, Alemania), 0,6 µL de cada uno de los iniciadores [0,5 µM]; 1 µL de sonda [0,2 µM]; 8,8 µL de agua calidad biología molecular y 5 µL de muestra de ADN. Las condiciones de termociclado fueron las siguientes: 1 ciclo de 95°C por 10 minutos, 40 ciclos de 95ºC por 10 segundos, 55ºC por 15 segundos y 72ºC por 15 segundos. Un ciclo final de 40ºC por 30 segundos.
Reacción en cadena de la polimerasa convencional para detección de genes gltA, ompB y ompA de Rickettsia spp.
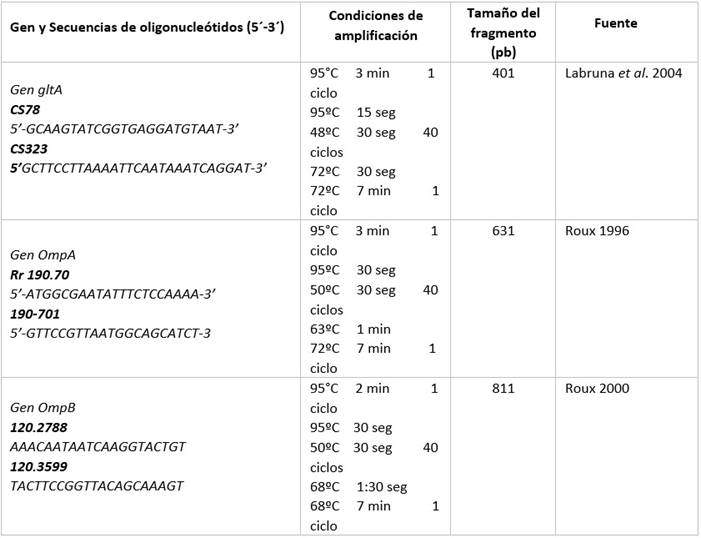
Las muestras positivas por PCR-RT fueron amplificadas posteriormente por PCR convencional, se usaron los iniciadores CS-78 y CS-323 que amplifican un fragmento de 401 pb del gen gltA. Así mismo, se utilizaron los iniciadores 120.2788 y 120.3599 que amplifican un fragmento de 811 pb del gen ompB que se encuentra en la mayoría de especies de Rickettsia; y los iniciadores Rr190-70 y 190-701 que amplifican un fragmento de 631 pb del gen ompA, el cual se encuentra solo en las especies que pertenecen al grupo de las fiebres manchadas.9
Las secuencias de los oligonucleótidos utilizados para PCR convencional se describen en la (tabla 1), así como las condiciones para los programas de amplificación.
Tabla 1 Secuencias de oligonucleótidos y condiciones de amplificación para los genes gltA, ompA y ompB.
Las reacciones de PCR para la amplificación de los diferentes genes se prepararon de acuerdo con los protocolos de identificación de Rickettsia del IIBT. Las mezclas de reacción se prepararon a volúmenes finales de 50 µL. Para la amplificación, se utilizó la enzima Taq ADN polimerasa recombinante (GenTaq, Colombia). La PCR se desarrolló en un termociclador PTC-100TM (MJ Research, Inc, Watertown, USA). Para cada reacción se incluyeron un control negativo (agua grado biología molecular) y un control positivo (ADN de R. amblyommii proporcionada por el IIBT).
Visualización de los productos amplificados
Para la visualización de los productos amplificados se realizó una electroforesis en gel de agarosa al 1,5 %. Posteriormente, los geles se examinaron en un transiluminador (BioRad, CA, EUA). El tamaño de los amplicones se determinó por comparación con el marcador de peso molecular de escalera de 100 pb (Invitrogen, CA, EUA).
Secuenciación de los productos amplificados
Los productos de las PCR se purificaron mediante el empleo del estuche de extracción PureLinkTM Quick Gel (Invitrogen) y se siguieron las instrucciones de la casa manufacturera. Las secuenciaciones se llevaron a cabo en la compañía de biotecnología Macrogen Inc en Corea del Sur y los análisis filogenéticos se realizaron con MEGA (molecular evolutionary genetics analysis) versión 6; las secuencias resultantes se compararon con las depositadas en el GenBank (GenBank/EMBL/DDBJ) mediante la herramienta BLAST (National Center for Biotechnology Information, Bethesda, MD, USA).10,11)
Resultados
Las garrapatas incluidas en este estudio fueron clasificadas en tres géneros: Riphicephalus, Dermacentor y Amblyomma.
Detección molecular de Rickettsia spp. por reacción en cadena de la polimerasa en tiempo real.
De 657 garrapatas incluidas en este estudio, se conformaron 169 grupos que fueron analizados por PCR-RT; en dos de ellos (grupos 18 y 19) se detectó ADN de Rickettsia spp. (1,18 %). El grupo 18 estaba conformado por 16 larvas y una ninfa de garrapata del género Amblyomma recolectadas en una pradera de la vereda Barcelona del municipio de Villavicencio. El grupo 19 por seis larvas y una ninfa de garrapata del género Amblyomma, recolectada en una pradera del municipio de Puerto López.
Detección de los genes gltA, ompB y ompA de Rickettsia spp. por Reacción en cadena de la polimerasa convencional.
La PCR convencional para detectar el fragmento del gen gltA mostró amplificación en los dos grupos de garrapatas de interés. (Figura 2).

Fig. 2 Electroforesis en gel de agarosa al 1,5 % para la detección del gen gltA en los grupos de garrapatas. La línea Mp corresponde al marcador de peso molecular (escalera de 100 pb). Las líneas 18 y 19 corresponden a los grupos de garrapatas donde se observa amplificación, la línea marcada con C- corresponde al control negativo y la C+ al control positivo
Amplificación del gen ompB con los iniciadores 120.2278 y 120.3599.
La PCR para la detección de un fragmento del gen ompB solo mostró amplificación en el grupo 19, que se corresponde a garrapatas Amblyomma recolectadas en una de las praderas del municipio de Puerto López.
Amplificación del gen ompA con los iniciadores Rr190.70 y 190-701.
La PCR convencional para el gen ompA, de igual manera a la anterior, solo amplificó a partir (Figura 3)

Fig. 3 Electroforesis en gel de agarosa al 1,5% para la detección del gen ompA y ompB en el grupo 19 de garrapatas. Las líneas MP50 corresponden al marcador de peso molecular (escalera de 50 pb). La amplificación de 631 pb corresponde al gen ompA y la de 811 pb al gen ompB
Identificación de las especies de Rickettsia en las garrapatas recolectadas
La secuenciación de los productos de PCR positivos para el gen gltA indican que la secuencia obtenida para el grupo 18 presento 99% de identidad con Rickettsia cepa Colombianensi y con Rickettsia sp clon Necoclí. (Figura 4)

Fig. 4 Relaciones filogenéticas del gen gltA de la muestra 18 (consenso 03 gltA). La historia evolutiva fue inferida por el método de máxima verosimilitud basado en el modelo del 3 parámetro de Tamura.10,11 Se calcularon los valores bootstrap para 1000 réplicas. Los Análisis evolutivos se realizaron en MEGA6. 9 La secuencia incluida se muestra en el recuadro rojo
Las secuencias nucleotídicas del gen gltA de los grupos 18 y 19 fueron idénticas entre sí. La secuencia nucleotídica del gen ompB del grupo 19 fue 100% idéntica con Rickettsia sp. cepa Colombianensi reportada en Córdoba (acceso Genbank JF905456 y JF905457).
Discusión
Los miembros más conocidos del genero Rickettsia son patógenos humanos que se encuentran asociados a artrópodos que se alimentan de sangre y dentro de estos artrópodos tal vez las más importantes sean las garrapatas ya que ellas son huéspedes ancestrales para las rickettsias.12
En el presente estudio se detectó infección por rickettsias, solamente en el género Amblyomma, lo cual se corresponde con los resultados reportados por Kelly en 2006.13) La especie A. cajennense es reportada como vector de R. rickettsii especialmente en América del Sur.14 Las garrapatas del perro Rh. sanguineus y D. nitens, que son encontradas frecuentemente en équidos, también han sido reportadas como vectores de R. rickettsii.15 En Argentina se reportó la presencia de R. massiliae en Rh. sanguineus.16) y en un estudio realizado en Brasil se aisló R. bellii en garrapatas A. ovale.17 En las últimas décadas las técnicas moleculares han permitido caracterizar genéticamente nuevas especies de rickettsia, que de acuerdo con los expertos pueden ser consideradas con el estatus de Candidatus y son contempladas como potenciales nuevas especies de Rickettsia.18
El número de microorganismos con el criterio de “Candidatus” está en aumento en todos los continentes, la asociación de una bacteria con la patogenicidad en humanos puede ser encontrada, incluso 65 años después como ocurrió con R parkeri, por lo tanto, todos los Candidatus de Rickettsia deben ser considerados como potenciales patógenos para el hombre.19
En Centroamérica fue aislada Candidatus R. nicoyana a partir de garrapatas colectadas de murciélagos al igual que Candidatus Rickettsia wissemanii” y Rickettsia peacockii. Pertenecientes al grupo de las fiebres manchadas.20 De igual forma en 2012 Miranda et al reportaron la presencia de una nueva especie de rickettsia del GFM, la nueva especie se denominó Candidatus Rickettsia sp. cepa colombianensi y fue detectada en Amblyomma dissimile, una garrapata comúnmente encontrada en reptiles.21 Aunque faltan estudios para caracterizar mejor a Candidatus R. colombianensi, se considera un potencial patógeno, por el hecho de pertenecer al Grupo de las fiebres manchadas, grupo caracterizado porque la mayoría de sus especies son patógenas para el hombre. Otra razón para considerar potencialmente patógena a Candidatus Rickettsia sp., cepa colombianensi es que tiene una alta identidad genética 99% con Rickettsia tamurae, cuyo ADN fue aislado del área de inflamación en el sitio de mordedura de garrapata en un paciente japonés.22
Quintero et al. en 2017 encontraron a Candidatus R.colombianensien garrapatas que estaban infestando humanos, hallazgo importante porque en estudios anteriores solo se había reportado su hallazgo en garrapatas que infestan iguanas (Iguana iguana) y una ninfa Amblyommacolectada en la rata espinosa (Proechimys semiespinosus).23
A diferencia de los países donde hay estaciones y el ciclo de las garrapatas depende de esto, en Colombia, la ausencia de estaciones hace que la actividad de las garrapatas sea constante y mayor en la época de sequía que va de diciembre a febrero aumentando la oportunidad de encuentro entre el artrópodo y el hombre.14
La principal limitación de nuestra investigación fue el bajo número de garrapatas colectadas, debido al cambio climático que se presentó en la etapa de estudio en que hubo un periodo prolongado de lluvias, meses en los cuales no se pudieron colectar garrapatas. Además, no se pudo hacer colecta de los artrópodos de otros municipios del departamento debido a las grandes distancias entre uno y otro.
Conclusiones
En conclusión, se demostró la circulación de Rickettsia durante el periodo de estudio en los municipios de Villavicencio y Puerto López y gracias a los análisis moleculares basados en los genes parciales gltA, ompA y ompB se logró identificar por primera vez en el departamento del Meta a Candidatus Rickettsia colombianensi a partir de garrapatas del género Amblyomma capturadas de pradera. Los resultados encontrados deben servir de alerta a las autoridades de salud acerca de un grupo de enfermedades que están olvidadas en nuestro medio.















