Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
MediSur
versión On-line ISSN 1727-897X
MediSur v.7 n.4 Cienfuegos jul.-ago. 2009
Artículos Originales
Revista Científica de las Ciencias Médicas en Cienfuegos
Modelación molecular de cuatro penicilinas: bencilpenicilina, fenoximetilpenicilina, ampicilina y amoxicilina
Elso Manuel Cruz Cruz,1 Eslhey María Sánchez Domínguez,2
Resumen
Fundamento: Las penicilinas se diferencian estructuralmente entre sí por las cadenas laterales. Al mismo tiempo, poseen diferencias en su acción farmacológica, que pueden estar asociadas a modificaciones inducidas en el anillo betalactámico y/o efectos localizados en las propias cadenas. Esclarecerlo, sería un aporte en la búsqueda de las características estructurales y electrónicas más adecuadas para el diseño de mejores penicilinas. Objetivo: Comparar las propiedades estructurales, densidades de cargas atómicas y los orbitales de frontera de cuatro penicilinas (bencilpenicilina, fenoximetilpenicilina, ampicilina y amoxicilina) mediante la modelación de la Teoría del Funcional de la Densidad. Método: Se optimizaron las estructuras moleculares con cálculos semiempíricos PM3. Las propiedades moleculares se calcularon según la Teoría del Funcional de la Densidad, a un nivel B3LYP/6-31G (d). Se analizaron las densidades de cargas atómicas y los orbitales de frontera. Se evaluó el efecto de los sustituyentes sobre las propiedades del anillo betalactámico. Resultados: Los parámetros estructurales del anillo betalactámico no variaron como consecuencia de las modificaciones en la cadena lateral unida al carbono 6. El anillo presentó una marcada tendencia a la planaridad. Tampoco se observaron variaciones en la densidad de carga positiva del carbono carbonílico. Conclusiones: La estructura y propiedades electrónicas del anillo betalactámico, base estructural de estos antibióticos, no tienen modificaciones significativas entre las penicilinas modeladas.
Palabras clave: Penicilinas;estructura molecular;modelos moleculares;
INTRODUCCIÓN
La era antibiótica en la medicina se inicia en 1928 con el descubrimiento de la penicilina, que 10 años más tarde se logra desarrollar como agente terapéutico y cuyo empleo comienza a generalizarse en 1950. En la misma década del 50 se crea la primera penicilina semisintética, la fenoximetilpenicilina. Le siguieron la meticilina, carbenicilina y otras, hasta completar un largo listado. A pesar de la variedad existente, la penicilina encabezó la lista durante mucho tiempo. En 1984 se inicia el uso del ácido clavulánico asociado a las penicilinas, comienza así el empleo de los inhibidores de betalactamasas. (1)
Estructuralmente, las penicilinas están formadas por la fusión de los anillos betalactámico y tiazolidínico, originando el ácido 6-aminopenicilánico, con una cadena lateral en la posición 6 del anillo. Variando esta cadena se modifican propiedades y surge la diversidad de miembros de la familia. Son antibióticos potentes, de amplio espectro bactericida y baja toxicidad. Dentro del grupo se pueden destacar la bencilpenicilina (penicilina G) y la fenoximetilpenicilina (penicilina V), consideradas como representantes clásicos de esta familia de betalactamas, efectivas frente a gérmenes grampositivos. También resultan relevantes la ampicilina y la amoxicilina, caracterizadas por su amplio espectro de acción y eficacia al ser combinadas con inhibidores de las betalactamasas. (1-3)
Las penicilinas actúan inhibiendo las etapas finales de la síntesis del péptido glucan, sustancia que le confiere la forma, rigidez y estabilidad a la membrana celular de muchas bacterias de importancia médica. (1-5) Con el paso del tiempo, las bacterias han reaccionado desarrollando mecanismos de resistencia que neutralizan la acción de los antibióticos, las penicilinas no han escapado a esta respuesta. El principal mecanismo de resistencia, para esta familia de antibióticos, consiste en la ruptura del anillo betalactámico por la acción de enzimas betalactamasas de las bacterias. Otro mecanismo importante es la síntesis de nuevas proteínas ligadoras de penicilinas (PBPs, por las siglas en inglés de Penicillin Binding Proteins), con menor afinidad por estas últimas. (5-8)
La búsqueda de nuevas fórmulas más potentes y generales que superen la resistencia bacteriana continúa. (9) Las penicilinas siguen estando en el centro de atención de los investigadores; sus propiedades antimicrobianas, características estructurales, mecanismo de acción, posibilidades de síntesis, entre otras, justifican este interés. El desarrollo alcanzado en los procesos de síntesis enzimática ha permitido la creación de una amplia colección de nuevas penicilinas, candidatas a convertirse en fármacos. (10)
En el presente trabajo se realiza la modelación molecular de cuatro penicilinas comerciales: bencilpenicilina, fenoximetilpenicilina, ampicilina y amoxicilina, con el objetivo de comparar sus propiedades estructurales, densidades de cargas atómicas y los orbitales de frontera. Además de la mencionada importancia de estos fármacos, su elección responde a las características de las cadenas laterales en el carbono 6, que permite medir el efecto de los sustituyentes en esa posición.
MÉTODOS
Se estudiaron 4 miembros de la familia de las penicilinas: bencilpenicilina, ampicilina, amoxicilina y fenoximetilpenicilina. (Figura 1). La modelación molecular se realizó utilizando computadoras personales IBM compatibles. Las estructuras tridimensionales se obtuvieron mediante la optimización de las geometrías moleculares a un nivel semiempírico PM3, (11) empleando el paquete de programas MOPAC Versión 7.01.3 para Linux. (12) Una vez obtenidas las geometrías de equilibrio se calcularon las densidades de cargas naturales y los orbitales moleculares, mediante el análisis de los Orbitales Naturales de Enlace, según la Teoría del Funcional de la Densidad (DFT, por las siglas en inglés de Density Functional Theory) a un nivel B3LYP/6-31G (d) // HF/PM3. (13) Se utilizó la teoría de los orbitales de frontera de Fukui para analizar la reactividad. (14) Los cálculos se realizaron con el paquete de programas Gaussian98 Versión A.7 para Linux. (15) El análisis gráfico se realizó con el programa MOLDEN Versión 3.6. (16)
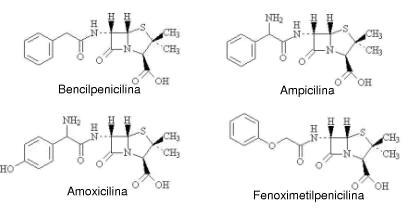
Figura 1. Estructura molecular de las penicilinas estudiadas
RESULTADOS
Los valores de las variables geométricas más relevantes del anillo betalactámico no varían como consecuencia de los cambios en la cadena lateral unida al carbono 6. (Tabla 1). En general, las diferencias no superan los 0,0004 nm y 0,3 grados para las distancias y ángulos de enlace, respectivamente. Solo la distancia de enlace N1 - C5 varía un poco más, unos 0,008 nm en ampicilina y amoxicilina, respecto a un modelo de penicilinas en el cual se sustituyó la cadena lateral por un grupo -CH3. Al comparar la fenoximetilpenicilina con el modelo, la planaridad del anillo se modifica 1,8 grados en el caso extremo.
Anexo 1. Datos estructurales más significativos.
Figura 2. Representación de las estructuras tridimensionales obtenidas.
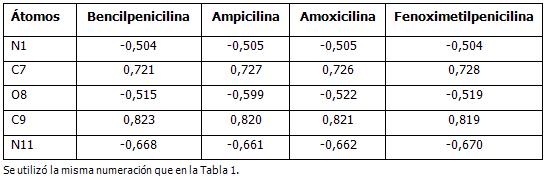
En los átomos del anillo betalactámico, las mayores densidades de cargas negativas recayeron sobre los heteroátomos, oxígeno (O8) y nitrógeno (N1). El carbono C7 presentó una marcada densidad de carga positiva. Fuera del anillo betalactámico, resaltó la densidad de carga negativa sobre el nitrógeno de la cadena lateral que define a cada penicilina (N11) y la carga positiva del carbono C9 del grupo carboxilo. (Tabla 2).
Las densidades de cargas en los átomos que conforman la base estructural de las penicilinas, el ácido 6-aminopenicilánico, no recibieron el efecto de los diferentes sustituyentes en el carbono C6 y mostraron valores acordes a lo esperado según su entorno electrónico.
Tabla 1. Estructura del anillo betalactámico en las penicilinas.
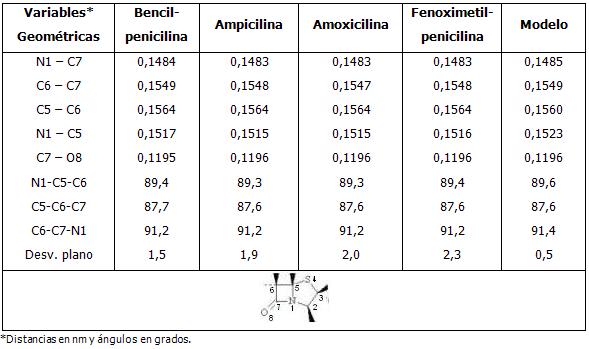
Tabla 2. Densidades de cargas atómicas más importantes.

Se utilizó la misma numeración que en la Tabla 1.
Las diferencias energéticas LUMO - HOMO no variaron significativamente en la muestra de penicilinas estudiadas. Sus valores fueron menores a 0,2 eV, propios de moléculas susceptibles a las transiciones electrónicas y que en consecuencia son muy reactivas. Esto ocurrió con todas las penicilinas modeladas. La forma de los orbitales se mostró muy compleja y existió una elevada deslocalización sobre diferentes átomos, características que limitan su empleo en el análisis de la reactividad.
DISCUSIÓN
Determinar la estructura tridimensional de una biomolécula constituye una premisa básica para poder explicar la función que desempeña. En este sentido, ha aumentado el empleo de las técnicas de modelación molecular, fundamentadas en métodos de la mecánica molecular y/o de la química cuántica. (11, 13, 17, 18) Una vez establecida la estructura molecular, es posible calcular las propiedades electrónicas y relacionarlas con la actividad biológica que manifieste el compuesto. Este es el fundamento de los estudios donde se establece la relación cuantitativa entre la estructura y la actividad (QSAR, por las siglas en inglés de Quantitative Structure-Activity Relationships), muy empleados en las modernas técnicas de diseño de fármacos asistido por computadoras. (19-26)
En la literatura científica aparecen reportes de trabajos donde se abordan problemáticas de interés médico a través de la modelación molecular. (24-32) Es común el empleo de los métodos de la mecánica molecular para calcular los parámetros estructurales en las biomoléculas, debido al elevado número de átomos que las forman. Sin embargo, los métodos de la química cuántica (semiempíricos, DFT y ab initio[1]) son más precisos, aunque también más costosos computacionalmente. (11, 13, 33-36) Anteriormente, otros autores han modelado compuestos betalactámicos determinando su estructura por métodos de la mecánica molecular. (30-32) Las penicilinas objeto de este estudio tienen un menor número de átomos, lo que permitió elevar la calidad del método a un nivel semiempírico PM3. (37-40) Partiendo de estas estructuras se calcularon las propiedades electrónicas según cálculos DFT, de probada calidad en la evaluación de conceptos químicos como: electronegatividad, dureza y blandura, reactividad y otos. (13, 33) Ya con anterioridad los cálculos DFT han sido empleados exitosamente en sistemas de interés biomédico. (20, 28, 41, 42)
Por otra parte, las investigaciones sobre las bases moleculares de la acción antibacteriana y de los mecanismos de resistencia desarrollados por las bacterias, han tenido un fructífero avance. (43-55)
Los antibióticos betalactámicos realizan su acción a nivel de la pared celular. Mediante una inhibición competitiva se unen a las proteínas ligadoras de penicilinas (PBPs), imposibilitando que estas ejerzan su acción catalítica en la unión y entrecruzamiento de las cadenas de péptido glucan. El mecanismo comienza por el ataque del oxígeno del grupo -OH de la serina, presente en las PBPs, al carbono carbonílico del anillo betalactámico (C7 en este trabajo). Se forma un complejo acil-enzima que se estabiliza mediante un enlace covalente, con la simultánea ruptura del anillo betalactámico. El complejo formado tiene una mayor estabilidad y se hidroliza muy lentamente, entorpeciendo la liberación de la enzima. La estructura del complejo acil-enzima define la potencia en la actividad antimicrobiana. Así, los antibióticos betalactámicos que forman complejos muy estables, con menores distancias de enlace, tienen una mayor acción contra esa bacteria. Las conformaciones espaciales y las características electrostáticas del antibiótico resultan determinantes en este mecanismo. Análogamente, la resistencia bacteriana por acción de las lactamasas sigue un mecanismo de reacción muy similar. En este caso el complejo se forma entre el antibiótico y la enzima betalactamasa. Existen evidencias experimentales de complejos acil-enzima que apoyan los mecanismos descritos. (43, 46-55)
En la reacción antibacteriana de los betalactámicos, el complejo acil-enzima se forma por un ataque nucleofílico al carbono carbonílico (C7) del anillo. Una mayor carga positiva en este átomo favorecerá el ataque. La planaridad del anillo permite un mejor acercamiento del -OH de la serina por un plano perpendicular, forma en la cual probablemente ocurra. Además, enlaces más fáciles de romper entre el nitrógeno del anillo (N1) y el carbono carbonílico (C7), facilitarían la formación del complejo. Atendiendo a estas premisas se analizaron los valores de estas propiedades en las modelaciones realizadas. Los resultados demuestran que no existen diferencias significativas entre las cuatro penicilinas. La estructura del anillo betalactámico no sufre modificaciones por efecto de los sustituyentes en el carbono 6. La similitud del anillo con el modelo de penicilinas, donde solo existe un grupo -CH3 en la cadena, lo confirman. El anillo betalactámico es el fragmento básico en la estructura del antibiótico, al transformarse y permitir materializar su acción. Sin embargo, otras partes de la estructura son determinantes para que el complejo acil-enzima llegue a formarse. Para alcanzar este complejo, el reconocimiento molecular del sustrato por la enzima debe permitir el mayor acercamiento entre ambos.
La ampicilina solo se diferencia estructuralmente de la bencilpenicilina por la presencia de un grupo amino (-NH2) unido al carbono contiguo al grupo carbonilo de la cadena lateral. El grupo amino crea una nueva región con notable polaridad. Otra marcada diferencia en la polaridad surge en la amoxicilina por la adición de un grupo -OH en posición para del anillo bencénico (Anexo 1). La presencia de grupos que crean polaridad y que poseen hidrógenos muy reactivos puede facilitar la interacción con grupos en las cadenas laterales de las PBPs, lo que aumenta las probabilidades de formación del complejo acil-enzima. Lo anterior coincide con el mayor espectro de acción de estas penicilinas. No ocurre así con la fenoximetilpenicilina, en la cual se sustituye el radical bencilo de la bencilpenicilina por un fenoximetil. A pesar de la presencia del oxígeno en este radical, su ubicación en el interior del esqueleto principal de la cadena no incrementa las regiones con polaridad en la molécula. En el caso de esta última penicilina, las propiedades analizadas no permiten explicar su menor fortaleza y espectros de acción.
En resumen, la modelación de las penicilinas es factible combinando cálculos semiempíricos para determinar la estructura y DFT para las propiedades electrónicas. Según los resultados obtenidos, las propiedades estructurales y las densidades de cargas atómicas en el anillo betalactámico del ácido 6-aminopenicilánico, así como las diferencias energéticas en los orbitales de frontera, no varían significativamente entre las penicilinas estudiadas. Es necesario considerar otros elementos para poder determinar las causas en las diferencias farmacológicas existentes entre ellas. A pesar de ello, para lograr un incremento de los sitios que potencialmente interactúen con las PBPs, es recomendable que en la estructura de las penicilinas existan grupos polares en posiciones externas de las cadenas laterales, lo que puede facilitar el proceso de formación del complejo acil-enzima.
[1] Locución latina que significa desde el principio.
Summary
Molecular modelling of four penicillins: bencylpenicillin, phenoxymethylpenicillin, ampicillin and amoxicillin
Background:Penicillins differ structurally among themselves by the lateral chains. At the same time, they have differences in their pharmacological action that can be associated to modifications included in the betalactamic ring and/or effects localized in the same chains. Making it clear would be a contribution to the search of the most adequate structural and electronic characteristics to design better penicillins. Objective:To compare structural properties, density of the atomic charges and the frontier orbitals of four penicillins: bencylpenicillin, phenoxymethylpenicillin, ampicillin and amoxicillin. Method:The molecular structures were optimized with PM3 semiempirical calculi. The molecular properties were calculated according to the Density Functional Theory, at a B3LYP/6-31G(d) level. The density of the atomic charges and the frontier orbitals were analyzed. The effect of the substituents on the properties of the betalactamic ring was evaluated. Results: The structural parameters of the betalactamic ring do not change as consequence of the modifications in the lateral chain linked to carbon 6. The ring has a marked tendency to planarity. There are no variations in the density of the positive charge of the carbonylic carbon. Conclusions: The structures and electronic properties of the betalactamic ring, structural basis of these antibiotics, do not have significant modifications among modelled penicillins.
Key words: Penicillins;molecular structure;models, molecular;
Referencias bibliográficas
1. Lozano Valdés D, Larrondo Muguercia H, Herrera Torres ML, Rivero Arias E, Zamora Marín R, Araújo Praderes LJ. Penicilinas. Acta Médica. 1998; 8(1):28-39.
2. Cué Brugueras M, Morejón García M. Antibacterianos de acción sistémica. Parte I. Antibióticos betalactámicos. Rev Cubana de Med Gen Integr. 1998; 14(4):347-61.
3. Larrondo Muguercia H. Amoxicilina/sulbactam. Alternativa terapéutica en las infecciones respiratorias extrahospitalarias. Acta Médica. 2000; 9(1-2):96-100.
4. Singh GS. ß-lactams in the new millennium. Mini-Reviews in Medicinal Chemistry. 2004; 4(1):69-92.
5. Zapun A, Contreras Martel C, Vernet T. Penicillin-binding proteins and ß-lactam resistance. FEMS Microbiol Rev. 2008; 32:361-85.
6. Gniadkowski M. Evolution of extended-spectrum ß-lactamases by mutation. Clin Microbiol Infect. 2008; 14 Suppl 1:S11-32.
7. Marín, M, Gudiol, F. Antibióticos betalactámicos. Enferm Infecc Microbiol Clin. 2003; 21(1):42-55.
8. Coates A, Hu Y, Bax R, Page C. The future challenges facing the development of new antimicrobial drugs. Nature Reviews Drug Discovery. 2002; 1:895-910.
9. Demain AL, Sánchez S. Microbial drug discovery: 80 years of progress. The Journal of Antibiotics. 2009; 62(1):5-16.
10. Kurochkina VB, Satarova DE, Nys PS. Combinatorial enzymology. Synthesis of novel betalactam libraries. Khimiya. 2000; 41 Suppl 6:S139-43.
11. Levine IN. Semiempirical and Molecular Mechanics treatments of molecules. In: Levine IN. Quantum Chemistry. 5a ed. New Jersey: Prentice-Hall; 2000. p. 626-92.
12. MOPAC [Molecular Orbital Package] v 7.01.3. Debrecen: Institute of Nuclear Research, Hungary; 2000. Avaidable from: http://esca.atomki.hu/mopac7/LINUX.
13. Levine IN. Ab initio and Density Functional treatments of molecules. In: Levine IN. Quantum Chemistry. 5a ed. New Jersey: Prentice-Hall; 2000. p. 480-625.
14. Fukui K, Fujimoto H. Frontier orbitals and reaction paths. In: Selected Papers of Kenichi Fukui. Japón: Fukui K, Fujimoto H (ed); 1997. p. 103-67.
15. Gaussian98 [Programa para computadora] Revision A. 7. Pittsburgh PA: Gaussian Inc.; 1998. Avaidable from: http//www.gaussian.com.
16. MOLDEN [Programa para computadora] v 3.6. University of Nijmegen: CAOS/CAMM Center. The Netherlands; 2000. Avaidable from: http://www.caos.kun.nl/schaf/molden/molden.html.
17. Gasteiger J. Chemoinformatics: a new field with a long tradition. Anal Bioanal Chem. 2006; 384:57-64.
18. Boeyens JCA, Comba P. Molecular mechanics: theoretical basis, rules, scope and limits. Coordination Chemistry Reviews. 2001; 212:3-10.
19. Torrens F. Structural, chemical topological, electrotopological and electronic structure hypotheses. Combinatorial Chemistry & High Throughput Screening. 2003; 6:801-9.
20. Dudek AZ, Arodzb T, Gálvez J. Computational methods in developing quantitative structure-activity relationships (QSAR): A review. Combinatorial Chemistry & High Throughput Screening. 2006; 9:213-28.
21. Veselovsky AV, Ivanov AS. Strategy of computer-aided drug design. Current Drug Targets - Infectious Disorders. 2003; 3:33-40.
22. Gasteiger J. Physicochemical effects in the representation of molecular structures for drug designing. Medicinal Chemistry. 2003; 3:789-96.
23. Raevsky OA. Physicochemical descriptors in property-based drug design. Medicinal Chemistry. 2004; 4:1041-52.
24. Hall LH, Hall LM. QSAR modeling based on structure-information for properties of interest in human health. SAR and QSAR in Environmental Research. 2005; 16(1-2):13-41.
25. Van Drie JH. Computer-aided drug design: the next 20 years. J Comput Aided Mol Des. 2007; 21:591-601.
26. Doweyko AM. QSAR: dead or alive?. J Comput Aided Mol Des. 2008; 22:81-9.
27. Zolotoy AB, Plouvier BP, Beatch GB, Hayes ES, Wall RA, Walker MJ. Physicochemical determinants for drug induced blockade of herg potassium channels: effect of charge and charge shielding. Curr Med Chem. 2003; 1:225-41.
28. Juan Wu W, Can Chen J, Qian L, Cheng Zheng K. QSAR and molecular design of benzo[b]acronycine derivatives as antitumor agents. Journal of Theoretical and Computational Chemistry. 2007; 6(2):223-31.
29. Anderson RD, Milletti MC. Structural and electronic characteristics of a series of glycosidase inhibitors. Drug Design & Discovery. 2007; 4:587-95.
30. Fenollar-Ferrer C, Frau J, Vilanova B, Donoso J, Muñoz F. Molecular modelling studies on Henry-Michaelis complexes of a class C ß- lactam compounds. J Mol Struct (THEOCHEM). 2002; 578:19-28.
31. Vilanova B, Coll M, Frau J, Muñoz F, Donoso J. Kinetic and molecular-modelling study of the interaction between Staphylococcus aureus PC1 enzyme and imipenem. Helv Chim Acta. 2001; 84:3366-79.
32. Vilanova B, Donoso J, Frau J, Muñoz F. Kinetic and molecular modelling studies of reactions of a class-A ß - lactamase with compounds bearing a methoxy group on the ß -lactam ring. Helv Chim Acta. 1999; 82:1274-88.
33. Levine IN. Comparison of methods. In: Levine IN. Quantum Chemistry. 5a ed. New Jersey: Prentice-Hall; 2000. p. 693-709.
34. Tubert-Brohman I, Guimaraes CRW, Repasky MP, Jorgensen WL. Extension of the PDDG/PM3 and PDDG/MNDO semiempirical molecular orbital methods to the halogens. J Comput Chem. 2004; 25:138-50.
35. Stewart JJ. Comparison of the accuracy of semiempirical and some DFT methods for predicting heats of formation. J Mol Model. 2004; 10:6-12.
36. Casadesus R, Moreno M, Gonzalez-Lafont A, Lluch JM, Repasky MP. Testing electronic structure methods for describing intermolecular H - H interactions in supramolecular chemistry. J Comput Chem. 2004; 25:99-105.
37. Verma RP, Hansch C. Camptothecins: A SAR/QSAR study. Chemical Reviews. 2009; 109:213-35.
38. Gasteiger J. Chemoinformatics - a textbook. Weinheim: Wiley-VCH; 2003.
39. Gasteiger J. Modeling chemical reactions for drug design. J Comput Aided Mol Des. 2007; 21:33-52.
40. Dwyer DS. Electronic properties of amino acid side chains: quantum mechanics calculation of substituent effects. BMC Chemical Biology [Serie en Internet]. 2005[ cited 7 Jul 2009]; 5: [aprox. 23p]. Avaidable from: http://www.biomedcentral.com/1472-6769/5/2.
41. McCormack AL, Somogyi A, Dongre AR, Wysocki VH. Fragmentation of protonated peptides: surface-induced dissociation in conjunction with a quantum mechanical approach. Anal Chem. 1993; 65:2859-72.
42. Anderson RD, Milletti MC. Structural and electronic characteristics of a series of glycosidase inhibitors. Drug Design & Discovery. 2007; 4:587-95.
43. Smith CA, Caccamo M, Kantardjieffc KA, Vakulenko S. Structure of GES-1 at atomic resolution: insights into the evolution of carbapenamase activity in the class A extended-spectrum ß -lactamases. Acta Cryst. 2007; 63:982-92.
44. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nature Reviews Microbiology. 2007; 5:175-86.
45. Smith PA, Romesberg FE. Combating bacteria and drug resistance by inhibiting mechanisms of persistence and adaptation. Nature Chemical Biology. 2007; 3(9):549-56.
46. Macheboeuf P, Fischer DS, Brown Jr T, Zervosen A, Luxen A, Joris B. Structural and mechanistic basis of penicillin-binding protein inhibition by lactivicins. Nature Chemical Biology. 2007; 3(9):565-9.
47. Fuda C, Hesek D, Lee M, Heilmayer W, Novak R, Vakulenko SB, Mobashery S. Mechanistic basis for the action of new cephalosporin antibiotics effective against methicillin and vancomycin resistant Staphylococcus aureus. Journal of Biological Chemistry. 2006; 281(15):10035-41.
48. Hujer AM,Kania M, Germen T, Anderson VE, Buynak JD, Ge X, Caspers P, Page MGP, Rice LB, Bonomo RA. Structure-activity relationships of different lactam antibiotics against a soluble form of Enterococcus faecium PBP5, a type II bacterial transpeptidase. Antimicrobial Agents and Chemotherapy. 2005; 49(2):612-8.
49. Sauvagea E, Kerffa F, Fonzéa E, Hermana R, Schootb B, Marquetteb JP. The 2.4-Å crystal structure of the penicillin-resistant penicillin-binding protein PBP5fm from Enterococcus faecium in complex with benzylpenicillin. Cell Mol Life Sci. 2002; 59:1223-32.
50. Sougakoff W, LHermite G, Pernot L, Naas T, Guillet V, Nordmann P, et al. Structure of the imipenem-hydrolyzing class A ß -lactamase SME-1 from Serratia marcescens. Acta Cryst. 2002; 58:267-74.
51. Lim D, Strynadka NCJ. Structural basis for the ß -lactam resistance of PBP2a from methicillin-resistant Staphylococcus aureus. Nature Structural Biology. 2002; 9(11):870-6.
52. Lim D, Ung Park H, De Castro L, Gyun Kang S, Sook Lee H, et al. Crystal structure and kinetic analysis of ß - lactamase inhibitor protein-II in complex with TEM-1 ß -lactamase. Nature Structural Biology. 2001; 8(10):848-52.
53. Walsh C. Molecular mechanisms that confer antibacterial drug resistance. Nature. 2000; 406:775-81.
54. Paetzel M, Danel F, De Castro L, Mosimann SC, Page MGP, Strynadka NCJ. Crystal structure of the class D ß - lactamase OXA-10. Nature Structural Biology. 2000; 7(10):918-25.
55. Chittock RS, Ward S, Wilkinson AS, Caspers P, Mensch B, Page MGP, et al. Hydrogen bonding and protein perturbation in ß-lactam acyl-enzymes of Streptococcus pneumoniae penicillin-binding protein PBP2x. Biochem J. 1999; 338:153-9.
Recibido: 28 de abril de 2009. Aprobado: 03 de septiembre de 2009.
Modelación molecular de cuatro penicilinas. Facultad de Ciencias Medicas Cienfuegos. Calle 51A y Avenida 5 de Septiembre. Cienfuegos, Cuba. CP 55100. Email: mikhail@infomed.sld.cu
1 Doctor en Ciencias Químicas. Profesor Asistente. Facultad de Ciencias Médicas ?Dr. Zoilo E. Marinello Vidaurreta?. Licenciada en Ciencias Farmacéuticas. Profesor Asistente. Facultad de Ciencias Médicas ?Dr. Zoilo E. Marinello Vidaurreta?.
2













