Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Cultivos Tropicales
versión impresa ISSN 0258-5936
cultrop vol.32 no.2 La Habana abr.-jun. 2011
La selección asistida por marcadores (MAS, “Marker-assisted selection”) en el mejoramiento genético del tomate (Solanum lycopersicum L.).
Marker-assisted selection (MAS, "Marker-assisted selection") in the genetic improvement of tomato (Solanum lycopersicum L.).
Dr.C. Marta Alvarez Gil
Instituto Nacional de Ciencias agrícolas (INCA)
RESUMEN
La mejora convencional de plantas se basa en la selección por el fenotipo de los individuos de interés por alguna característica distintiva, entre los individuos de progenies segregantes resultado de la hibridación. La obtención de nuevos cultivares por esta vía se toma no menos de
Palabras clave: Selección asistida por marcadores, mejora genética, tomate, marcadores moleculares, mapas de ligamiento.
ABSTRACT
Conventional plant breeding is primarily based on phenotypic selection of superior individuals among segregating progenies resulting from hybridization. It is often time consuming as breeding a new cultivar takes between eight and twelve years and even then, the release of improved cultivar is not guaranteed. Hence, breeders are extremely interested in new technologies that could make this procedure more efficient. Marker-assisted selection, often simply referred to as Marker-Assisted Selection (MAS) offers such a possibility by adopting a wide range of novel approaches to improving the selection strategies in crop breeding. Molecular markers are powerful research tools that make it possible to determine the genetic makeup of plants; for that it is possible the selection by plants genotype. In this paper, we refer the significant advancements towards application of molecular marker technology for crop improvement in the last decade, in tomato. Finally, we discuss reasons why the greater adoption of MAS in the future is inevitable, although the extent of its use will depend on available resources, and may be delayed in less-developed countries.
Key words: Marker-assisted-selection, MAS, tomato, plant breeding, molecular markers, linkage maps.
INTRODUCCIÓN
El tomate (Solanum lycopersicum L.) (Peralta y Spooner, 2005; (http://www.sgn.cornell.edu/about/solanum nomenclature.pl), es la especie que menor variabilidad genética posee dentro del género Solanum. Según Miller y Tanksley (1990) posee sólo el 5% de la variabilidad genética total de las especies de la subsección Lycopersicon, cuya amplia variabilidad genética radica en el genoma de las especies silvestres, las que han sido la fuente de la mayoría de los genes introducidos (“introgresados”) en el tomate, fundamentalmente, aquellos que determinan resistencia a enfermedades, calidad de los frutos y otros caracteres de importancia.
Es por ello que, la introducción de genes desde las especies silvestres hacia el tomate ha sido y seguirá siendo la base genética fundamental para el logro de cultivares e híbridos con nuevas características (Foolad 2007). Los primeros, fueron mediante métodos convencionales, lo cual requirió un gran esfuerzo por parte de los mejoradores. En los últimos años, gracias al desarrollo científico y tecnológico, se ha logrado la implementación de nuevas herramientas biotecnológicas que ofrecen amplias posibilidades para que los mejoradores se puedan trazar objetivos que fueron inalcanzables, hasta hace unos años. Entre estas, la de los marcadores genéticos estrechamente ligados a los genes de interés, que les permite la introducción de genes al tomate con mayor efectividad (Díez y Nuez, 2008).
Los marcadores genéticos se han empleado en diferentes fases de la mejora de las plantas: el pre-mejoramiento (conservación y uso de los recursos genéticos, conocimiento y uso del sistema reproductivo, caracterización y pureza de cultivares para emplearlos como progenitores); el mejoramiento (selección de progenitores y monitoreo en los híbridos, selección asistida para caracteres cualitativos y cuantitativos, introgresión de genes desde especies silvestres) y el post mejoramiento (caracterización y pureza de cultivares en los lotes de semilla, desarrollo e identificación de Organismos Genéticamente Modificados, entre otros). Estos se han usado, también con éxito, en otras líneas de investigación en plantas, tales como la filogenia, el diagnóstico de patógenos, y la agroindustria (Nuez et al., 2000; Borém, 2009).
Aún cuando algunos tipos de marcadores (morfológicos y bioquímicos) existían desde hace décadas, estos se empleaban con discreción, y no fue hasta el advenimiento de la tecnología de marcadores de Ácido desoxirribonucleico (ADN, “Deoxyribonucleic acid”), en la década de los años 80, que se abrió el camino hacia el uso práctico y rutinario de una nueva generación de marcadores, los marcadores moleculares de ADN. Gracias a los avances logrados en la automatización de la tecnología de ADN y proteinas, se ha abierto una nueva etapa en el conocimiento de las funciones génicas (genómica), lo que ha permitido el diseño de marcadores génicos a partir de las secuencias de los propios genes de interés, y con éstos, la mejora genética adquirirá un nuevo enfoque (Varshney et al., 2007).
Estos marcadores de ADN, ligados a genes de interés, pueden ser empleados para detectar la presencia de variaciones alélicas en éstos, con una eficiencia y precisión mejoradas. En los últimos años, se ha logrado saturar los mapas de ligamiento del tomate con dichos marcadores, y han sido asimilados por las compañías de semillas e Instituciones públicas para asistir a la selección en la mejora genética de esta especie (Foolad, 2005), mediante lo que se ha dado en llamar
La Selección asistida por marcadores está ganando una considerable importancia en el incremento de la eficiencia de la mejora de las plantas, al permitir transferir de forma más precisa, que los métodos clásicos de selección, regiones genómicas de interés (“foreground selection”) y acelerar la recuperación del genoma del parental recurrente (“background selection”).
Actualmente, algunos laboratorios trabajan en el estudio de las bases genéticas de los caracteres de importancia agronómica en las especies del género Solanum, incluidos los caracteres complejos, como el rendimiento, la calidad de los frutos y la tolerancia a estreses y a algunas enfermedades complejas. El genoma del tomate está siendo secuenciado por un consorcio internacional de 10 países (Mueller et al., 2005), que ha derivado múltiples bases de datos sobre genes, expresiones génicas y metabolitos, entre otros, que pueden ser localizados en
En este artículo, se expondrán las potencialidades de la selección asistida por marcadores moleculares en el tomate, por ser una aplicación directa a los programas de mejora de esta especie, dirigidos a la obtención de nuevos cultivares e híbridos. Se hará referencia también al surgimiento y aplicación de estos, la disponibilidad actual de mapas de ligamiento y marcadores de ADN en el tomate, mapeados y estrechamente ligados a caracteres de interés, así como los cambios en la concepción y estrategias en la mejora actual y futura de este cultivo, condicionados por los avances tecnológicos de los últimos años.
Surgimiento del mejoramiento molecular
La asimilación de las innovaciones logradas por la biología y la genética en la mejora de las plantas data desde sus inicios, en que algunos mejoradotes de plantas reconocieron de inmediato la importancia de la genética Mendeliana, aunque no fue hasta comienzos del siglo 20, cuando la genética cuantitativa reconcilió los principios mendelianos con la variación continua, que se reconoció de forma pública una integración completa de los principios de la genética y la mejora de las plantas (Paul and Kimmelman, 1988).
Los avances continuados que ha tenido la biología vegetal, el análisis e inducción de variaciones genéticas, la citogenética, la genética cuantitativa, la biología molecular, la biotecnología y, mas recientemente, la genómica, han incrementado los conocimientos científicos y la aplicación de nuevas tecnologías al proceso de mejora de plantas (Baenziger et al., 2006; Jauhar, 2006; Varshney et al., 2006) y han establecido nuevas herramientas para la creación, análisis y manipulación de la variación genética y el desarrollo de cultivares mejorados (Sharma et al., 2002; Varshney et al., 2006; Collard y Mackill, 2008), siendo una realidad actual el mejoramiento molecular como una práctica cotidiana y estandarizada para algunos cultivos.
Tal es así, que a principios de 1980 aparecieron los primeros reportes de plantas transgénicas usando Agrobacterium (Bevan et al., 1983; Fraley et al., 1983; Herrera-Estrella et al., 1983) y en 1996, poco mas de 10 años, ya se comercializaban los primeros cultivares transgénicos (Koziel et al., 1993; Delannay et al., 1995). También en esa década, comenzaron a desarrollarse mapas genéticos de alta resolución y a explotarse el ligamiento genético entre marcadores y caracteres de importancia para los cultivos (Paterson et al., 1988).
En los 90s, aparecieron ya las primeras aplicaciones de
La incorporación de genes múltiples afectando el mismo carácter es un gran reto para los programas de mejoramiento de plantas, ya que para muchos cultivos se requieren cultivares o híbridos que posean una combinación de diversas resistencias a estreses bióticos, tolerancia a estreses abióticos, caracteres agronómicos y de calidad, que permitan estabilidad en los rendimientos y la aceptación por los productores.
Si bien en este sentido se han reportado cultivares o híbridos obtenidos mediante
Según Barone et al. (2009), la genómica está definiendo la revolución tecnológica del mejoramiento asistido. En el futuro, ya no se enfocará
También, será posible un nuevo enfoque y aplicación de los resultados obtenidos por la genómica, gracias al avance en el conocimiento sobre las estructuras y funciones de los genes en diferentes especies de plantas. Estas nuevas herramientas tecnológicas permitirán una mejor integración de la mejora convencional, la genética y la genómica, lo que hará transitar
Marcadores moleculares en plantas, con referencia al tomate
Durante muchos años, la mejora genética se basó en la selección por el fenotipo, es decir, la selección directa por el carácter de interés, cuya eficiencia dependía de la heredabilidad del carácter, la influencia del ambiente, el tipo de herencia (monogénica o multigénica) y de la dominancia parcial o total del carácter en estudio.
Muchas de las complicaciones de la selección por el fenotipo se han podido solucionar, gracias a la selección indirecta con marcadores genéticos ligados al gen de interés. En este artículo, se hará referencia solo a este tipo de marcadores indirectos, ya que los marcadores que permiten una selección directa, y que son aquellos derivados del polimorfismo dentro del propio gen de interés, están aun siendo objeto de investigación o no están accesibles para utilizarse en la práctica rutinaria de los programas de mejora vegetal.
Un marcador genético es, según definió Griffiths et al. (1993), una variante alélica que se utiliza para marcar una estructura biológica o proceso a lo largo de un experimento genético. Para Nuez et al. (2000), es cualquier diferencia fenotípica controlada genéticamente y utilizada en el análisis genético. Por tanto, un marcador genético es un locus marcador que tiene que ser polimórfico y emplearse con una doble finalidad, para marcar o señalar el locus que controla la diferencia fenotípica y/o para marcar otro locus próximo (ligado) que controla el carácter de interés. Los atributos ideales de un marcador genético para estos autores son: (a) polimórfico (multialélico); (b) codominante; (c) no epistático, es decir, se puede leer el genotipo a partir del fenotipo, independientemente del genotipo de otros loci; (d) neutro, las sustituciones alélicas no tienen otros efectos fenotípicos y (e) insensible al ambiente, es decir, el genotipo se infiere a partir del fenotipo, independientemente del medio.
Los primeros marcadores utilizados en el análisis genético fueron morfológicos, que fueron muy usados para analizar la segregación de los cruzamientos y los primeros en ser mapeados. Estos marcadores, sin embargo, tuvieron un uso limitado, principalmente, porque su expresión podía estar influenciada por el ambiente, por factores genéticos (por ejemplo, epistasis o genes modificadores), lo que altera el fenotipo de la planta e interfiere en el mejoramiento (Staub et al., 1996).
Un segundo grupo de marcadores, los bioquímicos, comprenden proteínas de reserva de la semilla e isoenzimas, considerados estos últimos, como la primera generación de marcadores moleculares; a pesar de su naturaleza codominante y poca influencia ambiental en su expresión, su número es limitado, por lo que detectan bajos niveles de variación, condición necesaria para un mapa denso (Pérez de
Dentro del tercer grupo de marcadores se encuentran aquellos que detectan polimorfismo a nivel del ADN. Diversas son las técnicas que se utilizan para obtener estos marcadores moleculares basados en el ADN, tales como:
1- Marcadores basados en la hibridación: Polimorfismo de la longitud de los fragmentos de restricción (RFLP, “Restriction Fragment Lenght Polymorphism”; Botstein et al. 1980).
Los RFLPs, a pesar de tener los atributos ideales para un marcador y haber sido ampliamente empleados para generar mapas de alta densidad en tomate (Bernatzky and Tanksley 1986; Tanksley et al. 1992) y otros cultivos, tienen el inconveniente de que su análisis en el laboratorio es costoso y, frecuentemente, se necesitan isótopos radiactivos para su resolución, y aún cuando en los últimos años los costos se han reducido progresivamente y nuevas técnicas limpias de fluorescencia han sustituido a los isótopos radiactivos, no es accesible para el análisis automatizado del “genotipado” de las poblaciones, ni para el análisis rutinario de selección en poblaciones segregantes dentro de un programa de mejora.
2- Marcadores basados en
Los RAPDs, muy fáciles de analizar (después de la extracción del ADN, solo se necesitan de
Los marcadores AFLPs, basados en la combinación de dos técnicas, la digestión con enzimas de restricción, propia de los RFLPs, y
Los microsatélites o SSRs, son secuencias cortas de ADN que se encuentran distribuidas amplia y uniformemente en el genoma de la mayoría de los eucariontes, no son transcritas a ARN (“Ribonucleic acid”) y se les atribuye varias funciones, entre ellas, la de regulación génica. Además de polimórficos, son codominantres y altamente reproducibles. Han sido usados en plantas en la construcción de mapas genéticos (Chen y Foolad 1999), identificación y pureza varietal, estudios de diversidad, análisis de loci de caracteres cuantitativos (QTLs), entre otras aplicaciones.
3- Marcadores basados en secuencias: El Polimorfismo de nucleótidos simples (SNP, “Single Nucleotide Polimorphism”; Landegren et al. 1998), son el producto de sustituciones nucleotídicas que ocurren en el genoma con muy alta frecuencia, muy útiles en la confección de mapas, pero su desarrollo depende de secuenciaciones automatizadas, por lo que son caros, lo que limita su uso en proyectos a gran escala.
Para evitar los inconvenientes y aprovechar las ventajas de algunos de los marcadores altamente polimórficos (RFLPs, RAPDs y AFLPs) anteriormente mencionados, se han generado marcadores basados en una Región amplificada y caracterizada secuencialmente (SCAR, “Sequence Characterised Amplified Region”; Paran y Michelmore, 1993) o Secuencia polimórfica amplificada y digerida (CAPS, “Cleaved Amplified Polymorphic Sequence”; Konieczny y Ausubel, 1993).
Estos marcadores, basados en
Por otra parte, la identificación de marcadores SSRs y SNPs ha sido grandemente mejorada a partir del acceso a bases de datos de secuencias. También, el libre acceso a grandes colecciones de secuencias expresadas (EST, “Expressed Sequence Tag”; Adams et al., 1991), que han sido derivadas de cerca de 23 librerías de clones de ADN (cADN; Moore et al. 2002; van der Hoeven et al. 2002). Su uso en tomate está ampliamente descrito por van der Hoeven et al. (2002); Fei et al. (2004) y Labate y Baldo (2005); y están accesibles en el sitio web: http://ted.bti.cornell.edu/).
Asimismo, se han creado los COS (“Conserved Ortholog Set”, Fulton et al., 2002), obtenidos a partir de tamizar grandes bases de datos de marcadores EST y contrastarlas con las secuencias genómicas de Arabidopsis, identificándose una colección de 1025 marcadores COS, que son copias pequeñas o simples coincidentes en ambos genomas, muy útiles para el mapeo comparativo entre géneros, pero de bajo polimorfismo por ser secuencias conservadas.
Los tipos de marcadores de ADN que se han usado, mayoritariamente, en el mapeo genético de plantas superiores, incluidas las Solanáceas, han sido los RFLPs, RAPDs, AFLPs y SSRs, entre otros. El tomate, ha sido una de las especies cultivadas para la que una gran cantidad de marcadores se ha identificado y mapeado, entre los que se destacan los RFLPs, ESTs, SSRs, CAPSs, RAPDs, SCARs y AFLPs (ver tabla I).
La cantidad de marcadores identificados cambia muy rápidamente, y están accesibles para Solanáceas, en el sitio http://soldb.cit.cornell.edu/. Gracias al esfuerzo que se ha realizado en el Proyecto internacional de secuenciación del tomate (Mueller et al. 2005) se ha incrementado sustancialmente el desarrollo de los marcadores moleculares y su efectividad en los últimos años. Los cebadores para muchos de los marcadores en tomate se encuentran accesibles en los siguientes sitios: http://www.sgn.cornell.edu (Mueller et al. 2005)
http://www.kazusa.or.jp/jsol/microtom/index.html (Tsugane et al. 2005) http://www.tomatomap.net, http://www.ncbi.nlm.nih.gov (Wheeler et al. 2006) http://www.dpw.wau.nl/pv/CAPStomato/ (Bai et al. 2004).
No obstante, un problema en el desarrollo de los marcadores en tomate ha estado presente para la mayoría de los marcadores, incluidos los RFLPs y los marcadores basados en
Una revisión completa sobre las aplicaciones de los marcadores moleculares en la mejora de plantas puede ser encontrada en Nuez et al. (2000), pra una revisión sobre marcadores moleculares en tomate, ver a Foolad and Sharma (2005).
Marcadores ligados a un gen de interés en el tomate
Los primeros mapas de ligamiento que se confeccionaron para el tomate se basaron en caracteres morfológicos visualmente distinguibles. Labatte et al (2007), citaron a Hedrick and Booth, 1907, por la publicación del primer mapa de ligamiento mendeliano en caracteres morfológicos. En 1952, Rick desarrolló uno de los mapas genéticos más completos en tomate, utilizando caracteres morfológicos (Rick, 1978). En cambio, Butler (1968) reconoció como el primer mapa de ligamiento clásico de tomate el reportado en 1968, con 153 marcadores morfológicos y fisiológicos, ubicados sobre los 12 grupos de ligamiento de la especie.
Posteriormente, se le adicionaron las isoenzimas, publicándose en 1980 el primer mapa de ligamiento completo con 19 marcadores isoenzimáticos mapeados (Tanksley and Rick 1980), convirtiéndose este en el último mapa de ligamiento clásico publicado, con cerca de 400 genes morfológicos, fisiológicos, isoenzimáticos y de resistencia a enfermedades (Stevens and Rick 1986; Tanksley 1993) (ver tabla I).
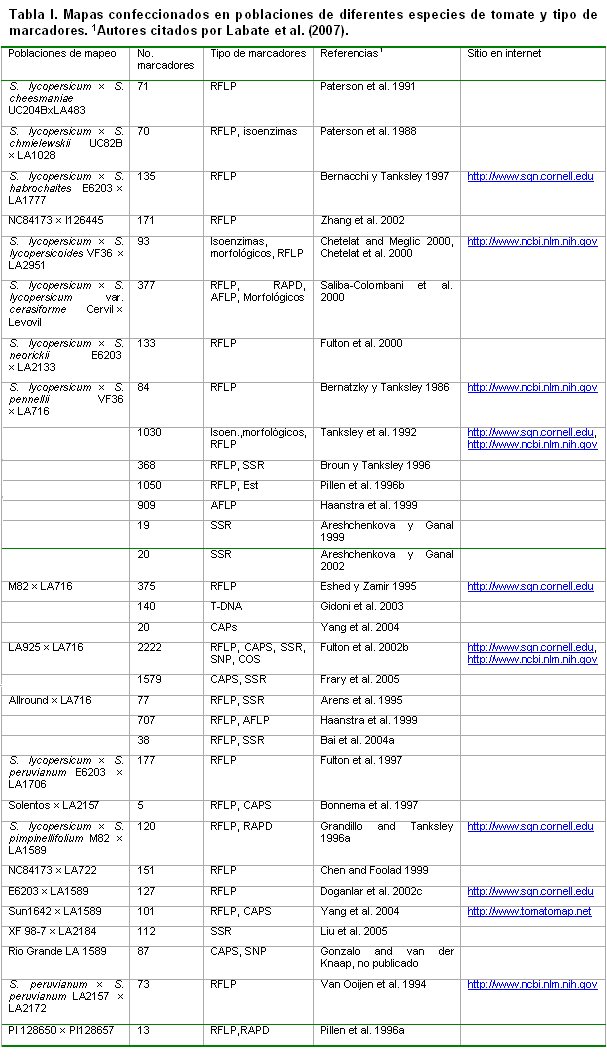
Aunque en 1986 fue publicado el primer mapa de ligamiento molecular de tomate, conteniendo 18 marcadores isoenzimáticos y 94 marcadores de ADN (Bernatzky and Tanksley 1986), no fue hasta 1992 que se publicó el primer mapa de ligamiento de alta densidad, conteniendo 1030 marcadores (Tanksley et al. 1992), basado en 67 plantas F2 de un cruce entre L. esculentum cv. VF36-Tm2a y L. pennellii LA716, que incluye marcadores morfológicos, isoenzimáticos y de ADN. En 1996, fue publicada otra versión mas saturada de este mapa (Pillen et al. 1996) y ya en 2007, se publicó un mapa de ligamiento de alta densidad con 2,222 marcadores moleculares mapeados (http://www.sgn.cornell.edu/cview/map.pl?map_id=9), con el cual fue posible que cualquier gen de interés ubicado en la población segregante de L. esculentum y ?L. pennellii esté próximo, al menos, a un marcador molecular (Foolad, 2007).
La mayor parte de los mapas de ligamiento del tomate referidos anteriormente han sido construidos basándose en marcadores RFLPs, RAPDs, AFLPs y SSRs. Varsnhey et al. (2007) los denominaron Marcadores de ADN aleatorios (RDMs, “Random DNA Markers”), debido a que estos, no necesariamente, fueron desarrollados a partir de sitios polimórficos en el propio gen; en cambio, denominaron Marcadores moleculares génicos (GMMs, “Genic Molecular Markers”) a una nueva generación de marcadores que ha surgido, en los últimos años, y que se desarrollan a partir de las secuencias previamente codificadas de genes completos, o parte de estos.
Actualmente, los grandes números de secuencias de genes expresados, ESTs, junto a los Análogos de Genes de Resistencia (RGAs) en el tomate, podrían ser empleados, según Sharma et al. (2008), en el desarrollo de marcadores moleculares génicos y en la saturación de los mapas, o ser usados como candidatos para identificar genes funcionales. Un enfoque tratado por Druka et al. (2010) es tratar de mapear loci de caracteres cuantitativos por su expresión, mediante el análisis y clasificación de series de mRNAs (“messenger Ribonucleic Acid”).
A pesar de los mapas creados, a partir de cruces entre especies, a partir de los cruces de S. lycopersicum con especies silvestres: S. cheesmaniae, S. chmielewskii; S. habrochaites, S. lycopersicoides, S. lycopersicum var. cerasiforme, S. neorickii, S. pennellii, S. peruvianum y S. pimpinellifolium (ver Tabla I y Sol genomics network, http://www.sgn.cornell.edu, muchos caracteres de importancia agrícola no segregaban en la misma población, y muchos marcadores de un mapa no eran polimórficos en otras poblaciones de tomate, principalmente, en aquellas derivadas de cruces entre variedades cultivadas o entre éstas y especies muy relacionadas, como S. pimpinellifolium L. y S. cheesmaniae (L. Riley) Fosberg, lo cual representa una limitante para la mejora.
La posibilidad de crear mapas intraespecíficos, a partir de cruces entre cultivares de tomate cultivado fue posible, recientemente, gracias a la explosión de información que se ha hecho accesible. Shirasawa et al. (2010) lograron construirlo mediante el desarrollo de marcadores SNPs, a partir de secuenciar clones de cADN (ESTs), obtenidos dentro del tomate cultivado. Estos SNPs se consideran la más abundante fuente de variación genómica, y ese elevado polimorfismo que detectan, los han convertido en una buena elección para la construcción de mapas de ligamiento en el tomate cultivado.
Esa estrategia de desarrollar mapas basándose en marcadores moleculares génicos, obtenidos a partir de secuencias informativas y de expresión, serán mucho más útiles para la identificación y uso de genes, incluidos los QTLs, de gran significación para
Para una amplia y detallada metodología acerca de la construcción de mapas genéticos en especies diploides y poliploides, consultar Cornides et al. (2002)
El surgimiento de diversos marcadores moleculares y los mapas de ligamiento saturados hizo posible encontrar asociaciones entre marcadores y fenotipos. Los mejoradotes pueden usar una asociación conocida entre marcadores moleculares y un carácter de interés para seleccionar por la presencia del marcador molecular, en lugar de por el fenotipo del gen de interés. Este proceso es conocido como Selección Asistida por Marcadores (MAS, Bai y Lindhout, 2007).
Si bien se avanzó de forma espectacular en los estudios de mapeo, diversidad genética, identificación de cultivares, entre otros, a partir de los marcadores de ADN, en primer lugar, los RFLPs que, a nuestro juicio, revolucionaron los estudios genéticos en plantas y, posteriormente, los AFLPs y RAPDs, que jugaron un extraordinario papel en la diseminación del empleo de los marcadores a numerosos laboratorios, estos no fueron empleados de forma práctica en los programas de selección, por su costo, no tener expresión de codominancia o, simplemente, no ser estables.
Estas características, bajo costo, expresión de codominancia y estar ligados de forma estable al gen de interés, son indispensables para la selección asistida, al menos, para la mejora de los cultivos que se reproducen por autogamia, como el tomate, ya que hay que practicar la selección en varias generaciones segregantes, a numerosas plantas, hasta obtener los nuevos cultivares. Es un hecho que los marcadores de isoenzimas cumplen con estos tres requisitos y, de hecho, fue uno de los primeros ejemplos prácticos del empleo de
Sin embargo, producto de la baja distribución de las isoenzimas en el genoma, no fue posible obtener muchos marcadores ligados a caracteres de interés, por lo que su empleo se limitó a los escasos caracteres en que se encontraron marcadores ligados a estos. Como ejemplos, Cubero, (2003) hizo referencia al marcador de fosfatasa ácida (Aps-11), ligado al gen Mi; o al de androesterilidad Ms ligado con Prx-2, una peroxidasa; los que han sido empleados en la mejora práctica por mucho tiempo.
No fue una práctica rutinaria
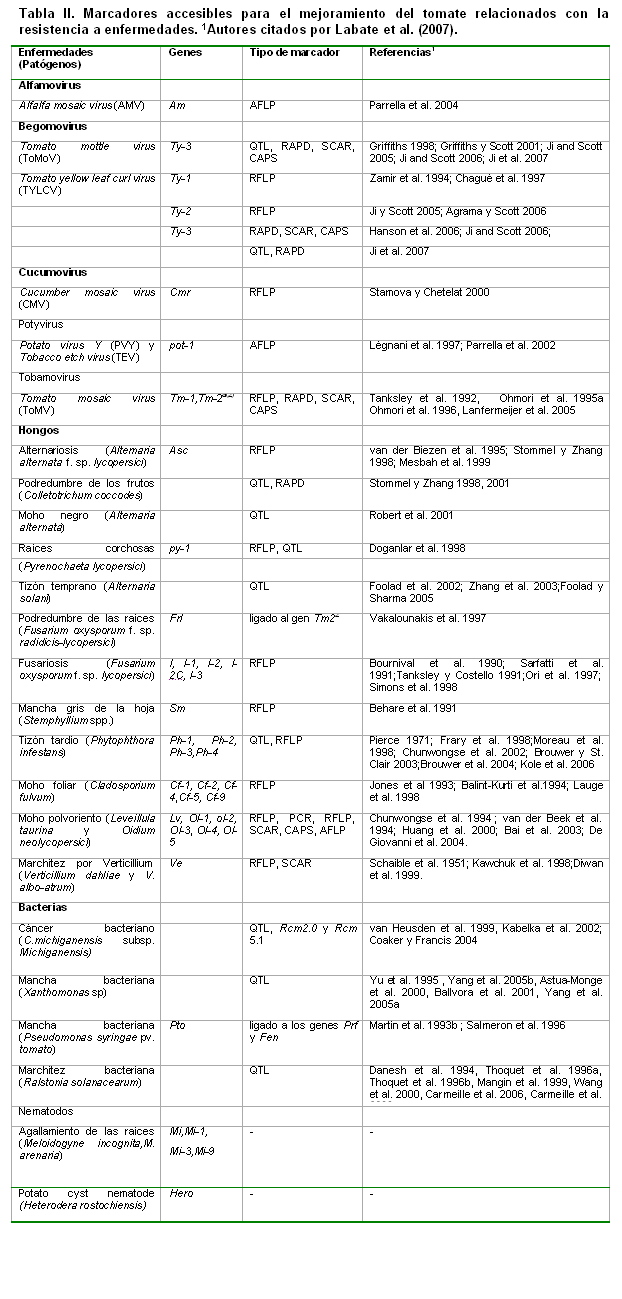
Muchos marcadores moleculares ligados a genes de interés en el mejoramiento del tomate han sido asociados, principalmente, a genes de resistencia a enfermedades, la mayoría, genes mayores (ver Tabla II).
Arens et al. (2010), compararon el empleo de marcadores moleculares y los ensayos biológicos en la selección de varios genes de resistencia en el tomate y llegaron a la conclusión de que, aunque hubo una respuesta correlacionada entre los resultados por ambos métodos, los marcadores mostraron superioridad en cuanto a poder distinguir las plantas heterozigóticas de las homocigóticas. Además de que los ensayos biológicos para evaluar la respuesta de las plantas a las enfermedades estudiadas, no siempre fueron confiables, debido a la influencia del ambiente y las dificultades propias de este tipo de evaluación.
Hongos
Alternariosis (Alternaria alternata f. sp. lycopersici) Asc RFLP van der Biezen et al. 1995; Stommel y Zhang 1998; Mesbah et al. 1999
Podredumbre de los frutos (Colletotrichum coccodes) QTL, RAPD Stommel y Zhang 1998, 2001
Moho negro (Alternaria alternata) QTL Robert et al. 2001
Raíces corchosas (Pyrenochaeta lycopersici) py-1 RFLP, QTL Doganlar et al. 1998
Tizón temprano (Alternaria solani) QTL Foolad et al. 2002; Zhang et al. 2003;Foolad y Sharma 2005
Podredumbre de las raíces (Fusarium oxysporum f. sp. radidicis-lycopersici) Frl ligado al gen Tm22 Vakalounakis et al. 1997
Fusariosis (Fusarium oxysporum f. sp. lycopersici) I, I-1, I-2, I-
Mancha gris de la hoja (Stemphyllium spp.) Sm RFLP Behare et al. 1991
Tizón tardío (Phytophthora infestans) Ph-1, Ph-2, Ph-3,Ph-4 QTL, RFLP Pierce 1971; Frary et al. 1998;Moreau et al. 1998; Chunwongse et al. 2002; Brouwer y St. Clair 2003;Brouwer et al. 2004; Kole et al. 2006
Moho foliar (Cladosporium fulvum) Cf-1, Cf-2, Cf-4,Cf-5, Cf-9 RFLP Jones et al 1993; Balint-Kurti et al.1994; Lauge et al. 1998
Moho polvoriento (Leveillula taurina y Oidium neolycopersici) Lv, Ol-1, ol-2, Ol-3, Ol-4, Ol-5 RFLP, PCR, RFLP, SCAR, CAPS, AFLP Chunwongse et al. 1994 ; van der Beek et al. 1994; Huang et al. 2000; Bai et al. 2003; De Giovanni et al. 2004.
Marchitez por Verticillium (Verticillium dahliae y V. albo-atrum) Ve RFLP, SCAR Schaible et al. 1951; Kawchuk et al. 1998;Diwan et al. 1999.
Bacterias
Cáncer bacteriano (C.michiganensis subsp. Michiganensis) QTL, Rcm2.0 y Rcm 5.1
van Heusden et al. 1999, Kabelka et al. 2002; Coaker y Francis 2004
Mancha bacteriana (Xanthomonas sp) QTL Yu et al. 1995 , Yang et al. 2005b, Astua-Monge et al. 2000, Ballvora et al. 2001, Yang et al. 2005a
Mancha bacteriana (Pseudomonas syringae pv. tomato) Pto ligado a los genes Prf y Fen Martin et al. 1993b ; Salmeron et al. 1996
Marchitez bacteriana (Ralstonia solanacearum) QTL Danesh et al. 1994, Thoquet et al. 1996a, Thoquet et al. 1996b, Mangin et al. 1999, Wang et al. 2000, Carmeille et al. 2006, Carmeille et al. 2006
Nematodos
Agallamiento de las raíces (Meloidogyne incognita,M. arenaria) Mi,Mi-1,
Mi-3,Mi-9
- -
Potato cyst nematode (Heterodera rostochiensis) Hero - -
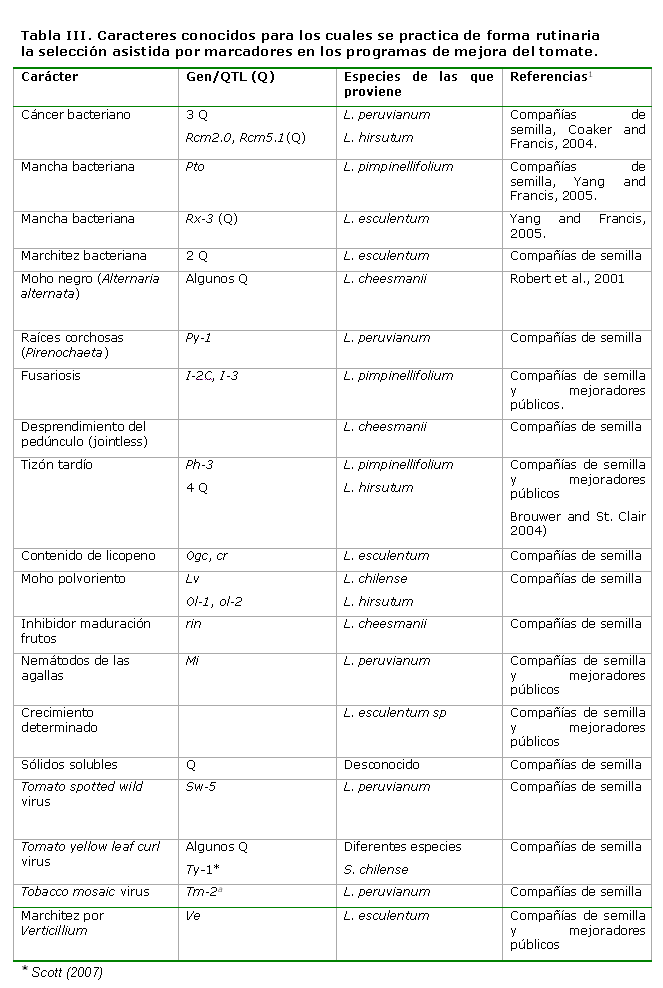
Muchos de estos marcadores están siendo utilizados en la selección asistida de forma rutinaria por las compañías de semilla e investigadores de centros públicos (ver Tabla III) para obtener líneas que se serán empleadas en la obtención de híbridos o cultivares, la mayoría, para caracteres de herencia simple y algunos QTLs.
El empleo de estos marcadores en el proceso rutinario de un programa de mejora, no solamente acelera el proceso de transferencia génica, sino también permite combinar (“piramiding”) diferentes genes deseables provenientes de diversas fuentes, con el objetivo de elevar el nivel de expresión del carácter de interés, desechar los genes que no interesan, sobre todo, cuando el donante es una especie silvestre y recuperar con más rapidez y efectividad el genoma del parental recurrente.
Numerosos esfuerzos se están haciendo para el mapeo de caracteres complejos a partir de la localización de QTLs. Es asombroso la cantidad de QTLs que ya han sido mapeados en los últimos años. Una revisión completa de marcadores QTLs ligados a la calidad de los frutos, la resistencia/tolerancia a los estreses de frío, sequía y salinidad, la floración, los frutos y el rendimiento, así como otras características de interés, está publicada (Foolad y Sharma 2005; Foolad 2007).
Sin embargo, la mayoría de éstos no están accesibles para ser utilizados de forma rutinaria en los programas de mejora, o como planteara Collard y Mackil (2008), las razones para que exista un número limitado de reportes publicados sobre el empleo de los QTLs, es que cuando se registra el producto ‘final’ de la mejora, que son los cultivares o híbridos, no se expone detalladamente el procedimiento del uso de los marcadores de ADN durante el proceso de obtención, sobre todo, por la competencia con otras compañías de semilla.
El alto costo de
En la actualidad, existe una gran fortaleza técnica que ha permitido avanzar en el ‘genotipado y ‘fenotipado’ de los principales cultivos de importancia agrícola, entre los que se encuentra el tomate. Sin embargo, el éxito de MAS a escala particular de los programas de mejora dependen, como se ha podido apreciar, de muchos factores críticos, como el número de genes de interés a ser transferidos, la distancia entre éstos y los marcadores que lo flanquean, el número de genotipos a seleccionar en cada generación, el tipo de cultivo y las facilidades de laboratorio para el trabajo con los marcadores.
Barone et al. (2009) hizo referencia a la necesidad de una mayor explotación de la plataforma bioinformática existente y desarrollar métodos que permitan integran las grandes bases de datos de los sistemas biológicos.
Otra dificultad que afrontan los mejoradores es que, a pesar de lo logrado en la saturación de mapas de alta densidad en muchas especies, solamente unos pocos loci de genes importantes han sido reportados con la documentación suficiente para reproducir la localización de éstos o, simplemente, faltan los trabajos de ligamiento que deben existir entre el marcador informado y su aplicación.
En resumen, la explotación de las potencialidades de
En Cuba, se ha empleado por algunos mejoradores de tomate los marcadores como apoyo a la mejora genética del tomate, en la selección de progenitores por su resistencia al ‘Virus del encrespamiento amarillo de la hoja de tomate’ (Tomato Yellow Leaf Curl Virus, TYLCV) (Piñón, 2009) y para la selección asistida por el marcador Aps 11, ligado al gen Mi, que confiere resistencia a algunas de las especies de nemátodos del género Meloidogyne en la obtención de cultivares resistentes (Alvarez et al. 2006).
A pesar de que no toda la información está disponible, con la que existe, y el hecho de que cada día se abaratan más los costos para asistir la selección de los genotipos, más que de los fenotipos; se puede lograr el incremento de la eficiencia de los programas de mejora en centros públicos no competitivos. Para ello, se requiere aunar esfuerzos a nivel local y regional; la formación de redes, beneficiaría, con mucho, la asimilación de este tipo de tecnologías. Compartir información, instalaciones, personal, e inclusive, materiales genéticos, sería una buena oportunidad para los países en desarrollo que no cuentan con toda la infraestructura necesaria o el personal capacitado.
Glosario de las abreviaturas más empleadas en el texto
ADN (Ácido desoxirribonucleico; DNA, “Deoxyribonucleic acid”)
AFLP (Polimorfismo de la longitud de los fragmentos amplificados, “Amplified Fragment Length Polymorphism”)
CAPS (Secuencia polimórfica amplificada y digerida, “Cleaved Amplified Polymorphic Sequence”)
EST (Etiquetas de secuencias expresadas, “Expressed Sequence Tag”)
GMMs (“Genic Molecular Markers”)
MAS (Selección asistida por marcadores, “Marker-Assisted Selection”)
PCR (Reacción en cadena de la polimerasa, “Polymerase Chain Reaction”)
QTL (locus de caracter cuantitativo, “Quantitative Trait Locus“)
RAPD (Polimorfismo del ADN amplificado al azar, “Random Amplified Polymorphic DNA”)
RDMs (“Random DNA Markers”)
RFLP (Polimorfismo de la longitud de los fragmentos de restricción, “Restriction Fragment Length Polymorphism”)
SCAR (Región amplificada y caracterizada secuencialmente, “Sequence Characterized Amplified Region”)
SGN (Red Genómica SOL, “SOL Genomics Network”)
SNP (Polimorfismo de nuceótidos simples, “Single Nucleotide Polymorphism”)
SSR (Microsatélites, “Simple Sequence Repeats”)
STR (Microsatélites, “Short Tandem Repeats“)
STS (“Sequence-Tagged Sites”)
REFERENCIAS
1. Adams, M.D.; Kelley, J.M.; Gocayne, J.D.; Dubnick, M.; Polymeropoulos, M.H.; Xiao, H.; Cerril, C.R.; Wu, A.; Olde, B.; Moreno, R.F.; Kerlavage, A.R.; McCombie, W.R. y Venter, J.C. Complementary DNA sequencing: Expressed sequence tags and human genome project. Science, 1981, vol. 252,p. 1651-1656.
2. Alvarez, M.; Lara, R. M.; Rodríguez, J.; Fernández-Muñoz, R. y Cuartero, J. Incorporación del gen Mi a variedades de tomate mediante el marcador Aps-11. Cultivos Tropicales, 2006, 27 (3), p. 69-73.
3. Arens, P.; Mansilla, C.; Deinum, D.; Cavellini, L.; Moretti, A.; Rolland, S.; Van der Schoot, H.; Calvache, D.; Ponz, F.; Collonnier, C.; Mathis, R.; Smilde, D.; Caranta, C. y Vosman, B. Development and evaluation of robust molecular markers linked to disease resistance in tomato for distinctness, uniformity and stability testing. Theor Appl Genet., 2010, vol.120(3) p. 655–664.
4. Baenziger, P.S.; Russell, W.K.; Graef, G.L. y Campbell, B.T. Improving lives: 50 years of crop breeding, genetics, and cytology (C-1). Crop Sci, 2006, vol. 46, p. 2230–2244.
5. Bai, Y. y Lindhout, P. Domestication and Breeding of Tomatoes: What have We Gained and What Can We Gain in the Future? Annals of Botany, 2007, vol. 100: 1085–1094.
6. Bai, Y.; Feng, X.; van der Hulst, R. y Lindhout, P. A set of simple PCR markers converted from sequence specific RFLP markers on tomato chromosomes 9 to 12. Mol Breed, 2004, vol. 13, p.281–287.
7. Barone, A.; Di Matteo, A.; Carputo, D. y Frusciante, L. High-Throughput Genomics Enhances Tomato Breeding Efficiency. Curr Genomics, 2009, vol. 10(1), p.1–9.
8. Bernatzky, R. y Tanksley, S.D. Towards a saturated linkage map in tomato based on isozymes and random cDNA sequences. Genetics, 1986, vol. 112, p. 887-898.
9. Bevan, M.W.; Flavell, R.B. y Chilton, M.D. A chimaeric antibiotic resistance gene as a selectable marker for plant c ell transformation. Nature , 1983, vol. 304, p. 184–187.
10. Borém, A. y Caixeta, E.T. Marcadores moleculares. Ed. Folha de Vicosa, eds. A. Borém y E.T. Caixeta. 532 p., 2009.
11. Botstein, D.; White, R.L.; Skolnick, M. y Davis, R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphism. Am J Hum Genet, 1980, vol. 32: 314-331.
12. Butler, L. Linkage summary. Tomato Genet Coop, 1968, Rep. 18, p. 4-6.
13. Chen, G.Q. y Foolad, M.R. A molecular linkage map of tomato based on a cross between Lycopersicon esculentum and L. pimpinellifolium and its comparison with other molecular maps of tomato. Genome, 1999, vol. 42, p. 94–103.
14. Child, A. A Synopsis of Solanum Subgenus Potatoe (G. Don) D’Arcy Tuberarium (Dun.) Bitter (s.l.). Feddes Repert.,1990, vol.101, p. 209-235.
15. Collard, B.C.Y. y Mackill, D.J. Marker assisted selection: an approach for precision plant breeding in the twenty-first century. Phil. Trans. R. Soc. B, 2008, vol. 363, p. 557–572.
16. Cornides, M.T. et al. MARCADORES MOLECULARES, nuevos horizontes en la genética y la selección de las plantas. Editorial Félix Varela, 2002, 367 p.
17. Cubero, J. I. Introducción a la Mejora genética vegetal. Ediciones Mundi-Prensa, 2da. Edición revisada y ampliada, 2003, 567 p.