Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.48 n.3 Ciudad de la Habana sep.-dic. 1996
Instituto de Medicina Tropical "Pedro Kourí"
Aplicación de la técnica de hibridación en colonias para la identificación de Vibrio cholerae 01 toxigénico
RESUMEN
Por medio de la reacción en cadena de la polimerasa (RPC), se obtuvo una sonda para el gen que codifica la subunidad B de la toxina colérica (CTxB) que portaba una cepa de referencia de Vibrio cholerae 01. La comprobación del producto amplificado se realizó por la técnica de hibridación en colonias. El producto amplificado hibridó con el gen que codifica para la subunidad B de la toxina colérica aislada de Perú y Ecuador, representante de la presente epidemia en América Latina y no lo hizo con las cepas filogenéticamente relacionadas.
Palabras clave: REACCIÓN EN CADENA POR POLIMERASA/métodos; VIBRIO CHOLERAE/aislamiento y purificación; TOXINA DEL CÓLERA/genética; HIBRIDACIÓN DE ÁCIDO NUCLEICO/genética.
INTRODUCCIÓN
El cólera permanece como una enfermedad endémica en muchas partes del mundo. Clásicamente, el mecanismo de patogenicidad de Vibrio cholerae 01 está mediado por la interacción de una enterotoxina (toxina colérica), cuya acción sobre la mucosa del intestino es responsable de la diarrea característica de la enfermedad.1
Esta toxina puede ser detectada por métodos biológicos in vitro e in vivo, inmunológicos y genéticos. En la literatura se reporta una amplia y diversa aplicación de los métodos de Biología Molecular para la identificación de la toxina colérica.2,3
En este trabajo se describe la detección del gen que codifica para la subunidad B (CtxB) de la toxina termolábil mediante hibridación en colonias.
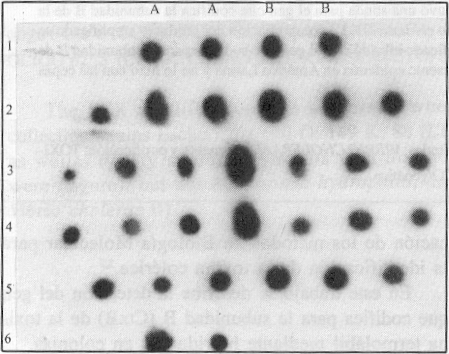
La hibridación fue llevada a cabo con diferentes cepas de colección: Vibrio cholerae 01 biotipo El Tor (2:2637.91 LANARE), Vibrio cholerae 01 biotipo El Tor (1:84.91 LANARE), Vibrio cholerae 01 biotipo El Tor (1:88012 LANARE), Vibrio cholerae 01 biotipo El Tor (3.2588.91 LANARE), Vibrio cholerae 01 biotipo El Tor (1.631.91LANARE) Perú; así como con 32 cepas aisladas de pacientes con cólera (10 cepas de Ecuador, 20 de Perú y 2 de la India). Como controles negativos se utilizaron las cepas: Escherichia coli 0:140 K:88 (LT+); Vibrio cholerae no-01, Plesiomonas shigelloides, Aeromonas hydrophila ATCC 7966 (no representadas estas 3 últimas en la figura).
FIGURA. Hibridación en colonias usando la sonda ctxB Vibrio cholerae 01 (569 B). Línea 1. A (E. coli 0:149 k:88). B (Vibrio cholerae 01 569 B biotipo clásico). Línea 2. Vibrio cholerae 01 biotipo El Tor (cepa de colección). Línea 3. Vibrio cholerae 01 biotipo El Tor (cepa de colección). Línea 4. Vibrio cholerae 01 biotipo El Tor aislado de pacientes con cólera en Perú). Línea 5. Vibrio cholerae 01 biotipo El Tor (aislado de pacientes con cólera en Ecuador).Línea 6. Vibrio cholerae 01 biotipo El Tor (aislado de pacientes con cólera en la India).
El método in situ estuvo basado en la hibridación en colonias sobre el papel Whatman 541, y todas las pruebas fueron llevadas a cabo con una alta astrigencia (formamida 50 % con lavados repetitivos a 65 EC).4
La sonda específica para la detección del gen CTxB usada en la hibridación en colonias fue obtenida del producto amplificado del gen que codifica para CTxB mediante la técnica de RPC, usando como cepa de referencia Vibrio cholerae 01 569B biotipo clásico.5 Este producto fue marcado con (alfa p32) dATP por la técnica de Random Primer con la utilización del un kit comercial Megaprimer, se obtuvo una actividad específica de 5 x 108 cpm/ìg de ADN.4
Como se observa en la figura, la sonda especifica hibridó con la cepa de referencia Vibrio cholerae 01 569B (control positivo), así como las otras cepas de Vibrio cholerae 01 aisladas de pacientes con cólera. Resultados negativos fueron obtenidos con las cepas
Escherichia coli 0:149 K:88 (LT+), Aeromonas hydrophila y Plesiomonas shigelloides.
Nuestros resultados están acorde con los publicados por otros autores que han aplicado la técnica de hibridación en colonias para la detección del gen que codifica para la subunidad A de la toxina colérica y no con cepas filogenéticamente relacionadas.6,7
Los autores concluyen que con la introducción de esta técnica en el Laboratorio Nacional de Referencia de Enfermedad Diarreica Aguda del Instituto de Medicina Tropical "Pedro Kourí" se cuenta con un método de buena sensibilidad y especificidad, que permite el estudio de un gran número de cepas en un simple experimento.
SUMMARY
By means of the polymerase chain reaction (PCR) it was obtained a probe for the gen that codifies the subunit B of cholerae toxin (CTxB), which carried a Vibrio cholerae 01 reference strain. The checking of the amplified product was performed by using the hybridization techniques in colonies. This product hybridized with the gen that codifies for the subunit B of cholerae toxin isolated from Peru and Ecuador, representing the present epidemics in Latin America, but it did not so with the phylogenetically related strains.
Key words: POLYMERASE CHAIN REACTION/methods; VIBRIO CHOLERAE/isolation and purification; CHOLERA TOXIN/genetics; NUCLEIC ACID HYBRIDIZATION/genetics.
REFERENCIAS BIBLIOGRÁFICAS
- Glass RI, Black RE. The epidemiology of cholera. En: Barua D, Greenough III WB, eds. Cholera. New York: Plenum Medical Book 1992.
- Karper BJ, Morris GJ, Levine MM. Cholerae. Clin Microbiol Rev1995;8:48-86.
- Almeida RJ, Hickman-Brenner FW, Sowers EG, Phr ND, Farmer III JJ, Wachsmuth IK. Comparison of a latex agglutination assay and a enzyme-linked immunosorbent assay for detecting cholera toxin. J Clin Microbiol 1990;28:128-30.
- Maniatis T, Fritsh EF, Sambrok J. Molecular cloning: a laboratory manual. New York: Cold Spring Harbor Laboratory, 1982.
- Bravo L, Monté R, Ramírez M, Maestre J, Llop A, Barro M, et al. Detection of toxigenic Vibrio cholerae 01 using polimerase chain reaction. Mem Inst Oswaldo Cruz 1992;87:443-4.
- Miyagi K, Matsumoto Y, Hayashi K, Takarada Y, Shibata SY, Yoh M, et al. Cholera diagnosed in clinical laboratory by DNA hybridization. Lancet 1992;339:988-9.
- Bera TK, Ghosh SK, Das J. Development and testing of cholera enterotoxins gene probe for detection of toxigenic Vibrio cholerae 01. Kansenshogaku-Zasshi 1990;64:1330-6.
Recibido: 12 de enero de 1996. Aprobado: 22 de julio de 1996.
Lic. Laura Bravo Fariñas. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana. Cuba.
1 Licenciada en Ciencias Biológicas. Investigadora Auxiliar. Instituto "Pedro Kourí" (IPK).
2 Especialista de II Grado de Medicina Tropical en Microbiología. Investigador Auxiliar. IPK.
3 Especialista de I Grado en Microbiología. Investigador Agregado. IPK.
4 Especialista de I Grado en Microbiología. Investigador Auxiliar. IPK.
5 Licenciada en Ciencias Biológicas. Investigadora Agregada. IPK.
6 Doctor en Ciencias Biológicas. Centro de Ingeniería Genética y Biotecnología (CIGB).