Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.57 n.3 Ciudad de la Habana sep.-dic. 2005
Instituto de Medicina Tropical Pedro Kouri
Estudio de la estabilidad genética del sistema de expresión de la proteína E del virus dengue 4 en la levadura metilotrófica Pichia pastoris
Lic. Rosa Ramírez Bartutis,1 Lic. Mayra Muné Jiménez,2 Ing. Manuel Mansur,3 Lic. Yudira Soto Brito4 y Lic. Tamara Pimentel5
Resumen
Se realizó el análisis de la estabilidad plasmídica de la levadura metilotrófica Pichia pastoris que expresa la proteína recombinante E del virus dengue 4. Para ello se estimó el número de generaciones desde el proceso de crecimiento en placa petri hasta la propagación en zaranda, así como el proceso de fermentación. Además se determinó en las colonias seleccionadas el patrón de integración por Dot-Blot y secuencia nucleotídica del gen E del virus dengue 4. Este estudio permitió comprobar la conservación e integridad de la secuencia aminoacídica de la proteína E, a pesar de encontrarse cambios genéticos producidos por mecanismos moleculares de la levadura; por otra parte, formó parte del control y chequeo realizado al banco de células primario de levadura que contiene el gen de interés, actualmente utilizado en el proceso de expresión de la proteína E del virus dengue 4.
Palabras clave: Levadura Pichia pastoris, banco de células primario, ADN recombinante, virus del dengue 4.
La idea de convertir células vivas en fábricas microscópicas para la producción de alimentos, enzimas y productos farmacéuticos fue planteada a inicios de la década de los ochenta por muchos investigadores y ya en los años 90 emergieron con gran fuerza sistemas de expresión de genes recombinantes en bacteria, levaduras y células mamíferas como métodos de producción para importantes productos.
La calidad de un banco de células usado para inocular un cultivo a gran escala es uno de los parámetros más importantes a tener en cuenta en el proceso de producción, porque este afecta directamente la calidad del producto final.1 Para asegurar esto es necesario monitorear el estado del sistema de expresión del DNA recombinante, así como demostrar que el cultivo se mantiene puro y las células estables en el proceso de producción final.
El análisis de la estabilidad genética de las células de un banco es imprescindible para determinar que la secuencia que codifica para el producto final es correcta y se mantiene en las células hasta el final del proceso.2 Mutaciones al nivel de la secuencia genética producidas por las células pueden ser incorporadas y mantenidas, dando como resultado final una proteína cuyas características pueden ser adversas en su utilización posterior (International Conference on Harmonization: Final Guideline on Quality of Biotechnological Products: Analysis of the Expression Construct in Cells Used for Production of rDNA-Derived Protein Products. ICH Secretariat, Geneva, Switzerland, February 1996).
En el presente trabajo se realizó la comprobación de la estabilidad y calidad genética de un banco de células de la levadura metilotrófica Pichia pastoris, como sistema de expresión para la proteína E del virus dengue 4, a partir del cual se desarrollaron estrategias para la obtención de un candidato vacunal.3 Esta proteína se obtiene de forma recombinante por un proceso de fermentación de la cepa MP36 mutante en el gen HIS3, a la cual se le incorporó por recombinación homóloga el gen que codifica para la proteína E del virus dengue 4 contenido en el vector de integración pFAO.
La integridad del marcador de selección, la detección del gen de la envoltura del virus dengue 4, así como su secuenciación y comparación con la secuencia original son los objetivos propuestos a desarrollar para determinar la influencia de un elevado número de generaciones en dicho sistema de expresión.
Métodos
Microorganismos
Se empleó la cepa MP36 (his3) proveniente de la levadura Pichia pastoris.
Medios de cultivo
YPD constituido por 10g/L de extracto de levadura, 20g/L de peptona bacteriológica y 20g/L de glucosa se utilizó para crecer la cepa MP36 en erlenmeyers de 250mL.
Medio salino compuesto por 22 g/L (NH4)2SO4, 28,6 g/L KH2PO4, 7,5 g/L MgSO4.7H2O, 0,4 g/L CaCl2.2H2O, glicerol 30 mL/L, 1 mL elementos trazas 1 000x, 2,5 mL vitaminas 400x.
Solución de vitaminas con 0,8 g/L pantotenato de calcio, 8,0 g/L myo-inositol, 0,8 g/L tiamina Cl-HCl, 0,8 g/L clorhidrato de piridoxal, 0,2 g/L nicotinamida, 0,8 g/L D(+) Biotina, 15 g/L K2HPO4.
Solución de elementos trazas que contenía 65 g/L FeSO4.7H2O, 6 g/L CuSO4.5H2O, 20 g/L ZnSO4.7 H2O, 3,0 g/L MnSO4, 0,415 g/L KI, 0,1 g/L H3BO4, 1 g/L Na2MoO4.2 H2O, 10 mL/L H2SO4.
Cultivo celular
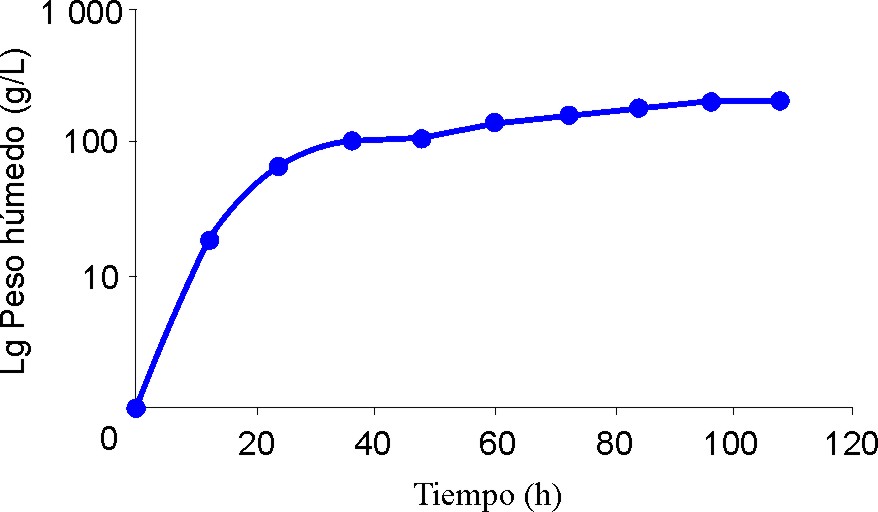
La determinación del número de generaciones se realizó creciendo a 28 °C y 10x g (Jouan GR 4.11 centrifuge) la levadura recombinante (clon 61) procedente del BCP en erlenmeyers de 250 mL con 50 mL de medio salino durante la etapa de propagación, la cual fue de 24 h. La relación de inóculo empleada fue de 1:200. La expresión se realizó en un fermentador Marubishi de 5 L con una etapa inicial de crecimiento con glicerol como fuente de carbono, seguida de la inducción con metanol a un flujo constante de 0,008 mL/h. El crecimiento óptimo en condiciones de fermentación de la levadura Pichia pastoris fue seguido por medición de peso húmedo durante un período de 120 h como se demuestra en la figura 1. La medición celular se realizó por medición de la densidad óptica (DO) a 600 nm durante el crecimiento en zaranda y por peso húmedo durante el proceso de fermentación, centrifugando muestras de 15 mL a 2000x g (Jouan GR 4.11 centrifuge) durante 15 min. La concentración de unidades formadoras de colonias se estimó por siembra en placas de YPD de muestras diluidas de los cultivos de incubación a 28 °C durante 72 h.
Fig. 1. Curva de crecimiento de la levadura metilotrófica Pichia pastoris durante el proceso de fermentación. Las determinaciones de peso húmedo se realizaron cada 12 h.
Determinación del número de generaciones
El número de generaciones (NG) se determinó teniendo en cuenta la expresión
NG=log2 nf/ni = (ln nf/ni)/ ln2
Donde n= DO= pH
n-número de generaciones
Determinación del patrón de integración
El ADN genómico de la levadura MP36 fue obtenido a partir de un cultivo de 200 mL de YPD y purificado utilizando el Kit WizardÒ Genomic ADN Purification System de Promega, que fue utilizado como control negativo en el análisis del patrón de integración del gen de la envoltura de dengue 4. Los ADN de 36 clones recombinantes fueron purificados por el mismo kit antes mencionado. La detección del gen de la envoltura se realizó por el método de hibridación Dot-Blot, utilizando como sonda el propio gen de la envoltura E del virus dengue 4 obtenido previamente por PCR (polymerase chain reaction, siglas en inglés de reacción en cadena de la polimerasa). El chequeo de los distintos ADN se realizó mediante una electroforesis en gel de agarosa horizontales a una concentración de 0,8 %, utilizando como buffer de corrida TBE (Tris-borato 40 mM, EDTA 2 mM pH 8,0)
La secuenciación del gen obtenido a partir de PCR de una colonia se realizó de forma semiautomática utilizando oligonucleótidos sintetizados en el Centro de Ingeniería Genética y Biotecnología (CIGB).
5 GGGAATTCT ATGCGATGCTTAGGAGTAGGA 3
5 GGGAATTCTTAAAACATCTTGCCAATGGAACT 3
Resultados
Los valores de los parámetros chequeados en cada una de las etapas durante el proceso de fermentación en cuanto a número de generaciones son mostrados en la tabla.
Tabla. Parámetros analizados durante el proceso de crecimiento en placas petri y de la propagación en zaranda durante 120 h
| Placas Petri | Erlenmeyer 250 mL | |
| Volumen inicial (L) | - | 0,25 |
| Volumen final (L) | - | 0,25 |
| Concentración biomasa inicial (g/L) | - | - |
| Concentración biomasa final (g/L) | - | 28 |
| Biomasa inicial (kg) | - | - |
| Biomasa final (kg) | - | 0,012 |
| Número de células inicial | 1 | - |
| Número de células final | - | 1,2 x 1013 |
| Número de generaciones | 66,9 | |
El número total de generaciones alcanzado en condiciones de proceso fue de 66,9, correspondiendo este valor al crecimiento en placas y al proceso de crecimiento en zaranda.
Para comprobar la calidad y estabilidad genética del banco desarrollado se procedió al estudio con un número de generaciones superior al obtenido, aproximadamente el doble de lo alcanzado.
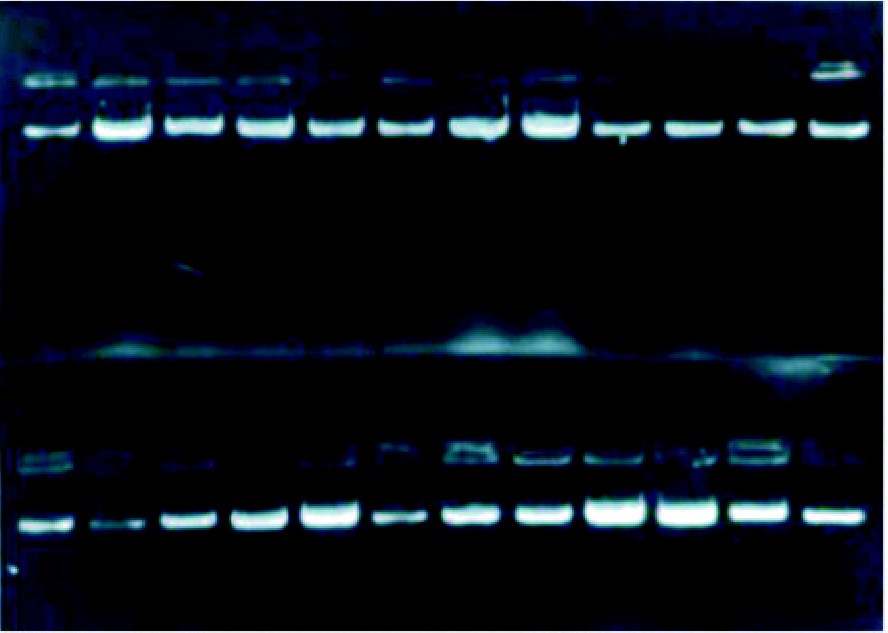
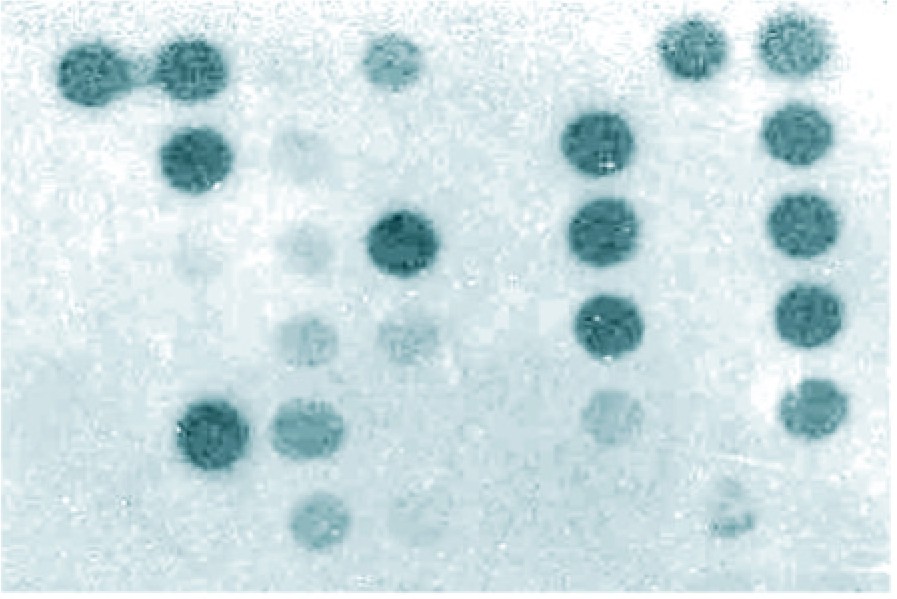
Una vez extraído el ADN de las clones recombinantes (fig. 2) se procedió a comprobar el patrón de integración por hibridación del gen E del virus dengue 4, utilizando la técnica de Dot-Blot en colonias seleccionadas después de un cultivo de alto número de generaciones. De las 36 colonias testadas 13 resultaron positivas (fig. 3) demostrando la presencia del gen E del virus dengue 4 en el genoma de la levadura.
Fig. 2. Extracción del ADN de las colonias.
Fig. 3. Dot-Blot de las colonias. Como control positivo se utilizó el gen de la envoltura de dengue 4 (posición superior izquierda), como control negativo la cepa MP36 sin transformar (posición inferior izquierda).
El análisis de la secuencia arrojó 3 cambios aminoacídicos (posiciones 261, 282, 575 de la secuencia nucleotídica) al compararla con la secuencia original antes obtenida.
ATGCGATGCGTAGGAGTAGGAAACAGAGACTTTGTGGAAGGAGTCTCAGGTGGAGCATGG 60
GTCGACCTAGTGCTAGAACATGGAGGATGCGTCACAACCATGGCCCAAGGAAAACCAACC 120
TTGGATTTTGAACTGACTAAGACAACAGCCAAGGAAGTGGCTCTGTTAAGAACCTATTGC 180
ATTGAAGCCTCAATATCAAACATAACTACGGCAACAAGATGTCCAACGCAAGGAGAGCCT 240
TATCTGAAAGAGGAACAGGACCAACAGTACATTTGCCGGAGAGATGTGGTAGACAGAGGG 300
TGGGGCAATGGCTGTGGCTTGTTTGGAAAAGGAGGAGTTGTGACATGTGCGAAGTTTTCA 360
TGTTCGGGGAAGATAACAGGCAATTTGGTCCGAATTGAGAACCTTGAATACACAGTGGTT 420
GTAACAGTCCACAATGGAGACACCCATGCAGTAGGAAATGACACATCCAATCATGGAGTT 480
ACAGCCATGATAACTCCTAGGTCACCATCGGTGGAAGTCAAATTGCCGGACTATGGAGAA 540
CTAACACTCGATTGTGAACCAGGTCTGGAATTGTACTTTAATGAGATGATTCTGATGAAA 600
ATGAAAAAGAAAACATGGCTCGTGCATAAGCAATGGTTTTTGAATCTGCCTCTTCCATGG 660
ACAGCAGGAGCAGACACATCAGAGGTTCACTGGAATTACAAAGAGAGAATGGTGACATTT 720
AAGGTTCCTCATGCCAAGAGACAGGATGTGACAGTGCTGGAATCTCAGGAAGGAGCCATG 780
CATTCTGCCCTCGCTGGAGCCACAGAAGTGGACTCCGGTGACGGAAATCACATGTTTGCA 840
GGACATCTTAAGTGCAAAGTCCGTATGGAGAAATTGAGAATCAAGGGAATGTCATACACG 900
ATGTGTTCAGGAAAGTTTTCAATTGACAAAGAGATGGCAGAAACACAGCATGGGACAACA 960
GTGGTGAAAGTCAAGTATGAAGGTGCCGGAGCTCCGTGTAAAGTCCCCATAGAGATAAGA 1020
GATGTAAACAAGGAAAAAGTGGTTGGGCGTATCATCTCATCCACCCCTTTGGCTGAGAAT 1080
ACCAACAGTGTAACCAACATAGAATTAGAACGCCCTTTGGACAGCTACATAGTGATAGGT 1140
GTTGGAAACAGCGCATTAACACTCCATTGGTTCAGGAAAGGGAGTTCCATTGGCAAGATG 1200
TTT 1203
Discusión
Las 13 colonias positivas por Dot-Blot corroboraron la presencia del gen E del virus dengue en el genoma de la levadura debido a su integración en este. Por otra parte los cambios ocurridos en la secuencia nucleotídica del gen de la envoltura pueden deberse a procesos moleculares de la propia levadura o a errores introducidos por la enzima Taq polimerasa durante el proceso de amplificación de la secuencia. Estos cambios no afectaron la integridad de la proteína, la cual se ha utilizado en posteriores estudios de inmunogenicidad del virus dengue.
Los experimentos desarrollados demostraron que a pesar de los cambios presentes en el gen de la envoltura luego del proceso de transformación en la levadura, se conserva la información aminoacídica y la integridad de la proteína, lo que permite la utilización del sistema de expresión de la levadura metilotrófica Pichia pastoris para la producción de la proteína recombinante E del virus dengue 4 y como candidato vacunal, requerimiento necesario y exigido por el Centro de Evaluación Biológica de la FDA (Center for Biologics Evaluation and Research, Guidance for Industry: for the submission of chemistry, manufacturing, and controls information for therapeutic recombinant DNA-derived product or monoclonal antibody product for in vivo use food and drug administration, Rockeville, MD, August 1996).
Agradecimientos
A la colaboración para la realización de este trabajo de las licenciadas Maida Candelario y Zeila Santana del laboratorio de Biología Molecular perteneciente a la Subdirección de Control de la Calidad del Centro de Ingeniería Genética y Biotecnología.
Study of the genetical stability of the system of expression of protein E of dengue virus 4 in methyltrophic yeast Pichia pastoris
Summary
The plasmidic stability of methyltrophic yeast Pichia pastoris expressing the recombinant protein E of dengue virus 4 was analyzed. To this end, the number of generations from the growth process in petri plaque to the propagation in zaranda was estimated, as well as the fermentation process. Besides, in the selected colonies the integration pattern was determined by Dot-Blot and neuclotide sequence of the gene E of dengue virus 4. This study allowed to prove the conservation and integrity of the aminoacid sequence of protein E, despite the genetic changes produced by molecular yeast mechanisms. On the other hand, it was also part of the control and checking of the primary bank of yeast cells that contains the gene of interest used at present in the process of expression of protein E of dengue virus 4.
Key words: Yeast Pichia pastoris, primary cell bank, recombinant DNA, dengue virus 4.
Referencias bibliográficas
2. Kittle JD, Pimentel BJ. Testing the Genetic Stability of Rrecombinant DNA Cell Bank. Pharm Technol 1998:38-44.
3. Muné M. Carboxyl terminally truncated dengue 4 virus envelope glycoprotein expressed in Pichia pastoris induced neutralizing antibodies and resistance to dengue 4 virus challenge in mice. Arch Virol 2003;148(11):2267-73.
Recibido: 19 de abril de 2005. Aprobado: 4 de octubre de 2005.
Lic. Rosa Ramírez Bartutis. Instituto de Medicina Tropical Pedro Kourí, AP 601, CP 11300, Ciudad de La Habana, Cuba. Correo electrónico: rosa@ipk.sld.cu
1 Máster en Biología Molecular y Biotecnología. Licenciada en Bioquímica. Aspirante a Investigadora. Instituto de Medicina Tropical Pedro Kourí (IPK).
2 Máster en Virología. Licenciada en Microbiología. Investigadora Auxiliar. IPK.
3 Máster en Procesos Biotecnológicos. Ingeniero Químico. Centro de Ingeniería Genética y Biotecnología.
4 Master en Virología. Licenciada en Microbiología. Investigadora Agregada. IPK.
5 Licenciada en Ciencias Farmacéuticas. IPK.