Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.60 n.2 Ciudad de la Habana Mayo-ago. 2008
ARTÍCULOS ORIGINALES
Detección y genotipificación de Helicobacter pylori sobre la base de los genes ADNr 16S
y el gen asociado a citotoxina (cagA) y posible asociación con enfermedades gastrointestinales
Detection and genotyping of Helicobacter pylori based on DNAr 16S genes and citotoxin-associated gen cagA and possible association with gastrointestinal diseases
Angie Patricia Molina Delgado,I Carlos Alberto Jaramillo Henao,II María del Pilar Delgado Perafán,III Mabel Elena Bohórquez LozanoIV
y Adolfo Ámezquita TorresV
I Especialista en Microbiología. Aspirante a Máster en Ciencias Biológicas. Asistente Graduada. Laboratorio de Diagnóstico Molecular y Bioinformática, Universidad de los Andes. Bogotá. Colombia.
II Especialista en Microbiología. Máster en Ciencias. Profesor Titular. Laboratorio de Diagnóstico Molecular y Bioinformática, Universidad de los Andes. Bogotá. Colombia.
III Especialista en Bacteriología. Máster en Ciencias. Profesora Asociada. Laboratorio de Diagnóstico Molecular y Bioinformática, Universidad de los Andes. Bogotá. Colombia.
IV Especialista en Patología. Profesora Asociada. Citogenética, Filogenia y Evolución de Poblaciones. Universidad del Tolima. Ibagué. Colombia.
V Doctor en Ciencias Biológicas. Profesor Asociado. Grupo de Ecofisiología del Comportamiento y Herpetología, Universidad de los Andes. Bogotá. Colombia
RESUMEN
Introducción: Helicobacter pylori es un microorganismo colonizador de la mucosa gástrica, reconocido como agente etiológico de enfermedades gástricas severas. No se conoce con exactitud la prevalencia en Colombia,
Objetivos: este estudio busca brindar información acerca de la frecuencia de la infección, en pacientes residentes en el Tolima, además de tipificar las cepas mediante la amplificación del gen asociado a citotoxina cagA.
Métodos: biopsias gástricas procedentes de 60 pacientes con diagnóstico de dispepsia fueron procesadas inicialmente mediante un análisis histopatológico, usando la coloración de Giemsa. La detección del microorganismo mediante reacción en cadena de la polimerasa se llevó a cabo usando como blanco una región del gen ADNr 16S especie-específico. La región 5' del gen cagA, fue el blanco de amplificación para la tipificación del microorganismo. El análisis estadístico incluyó el cálculo de la sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo. La concordancia entre las pruebas diagnósticas fue determinada con el cálculo del índice Kappa de Cohen.
Resultados: De los 60 pacientes incluidos, 62 % resultó positivo para la detección molecular del microorganismo, coherente con los datos reportados para la población; y de estas cepas, 43 % se genotipificó como cagA+. Factores como la sensibilidad (71 %), especificidad (58 %) y la concordancia de las pruebas a través del Índice Kappa de Cohen (0,271), permitieron la comparación de estos resultados con los obtenidos para la histología, prueba de oro para el diagnóstico.
Conclusiones: los resultados presentados permiten ilustrar una posible correlación entre la presencia del gen cagA y el desarrollo de gastritis atrófica en la población estudiada.
Palabras clave: Helicobacter pylori, factores de virulencia, cagA, ADNr 16S, detección, tipificación, reacción en cadena de la polimerasa, enfermedades gastrointestinales.
ABSTRACT
Background: Helicobacter pylori is a microorganism that colonizes the gastric mucosa and is recognized as the etiologic agent of severe gastric diseases. Its prevalence in Colombia is not well known.
Objective: this study tried to bring information about the frequency of infection in patients living in Tolima province, and also to type the strains through amplification of the cytotoxin-associated gene cagA.
Methods: Gastric biopsies from 60 patients diagnosed with dyspepsia were processed using histological and pathological analysis, using Giemsa staining. Helicobacter pylori was detected by PCR using species-specific 16S DNAr gene. For typing, the 5' region of cagA gene was chosen as amplification target. The statistical analysis included sensitivity, specificity, positive and negative predictive values. The agreement among diagnosis tests was estimated by Cohen's Kappa Index.
Results: Out of the 60 patients included in the study, 62 % was positive to the molecular detection of the microorganism, these results were similar to previous reports for that population. Of these strains, 43 % were genotyped as cagA+. Factors such as sensitivity (71 %), specificity (58 %) and test agreement using Cohen's Kappa Index (0.271) allowed the comparison of these results and those of the histological test, known as the gold standard for the diagnosis.
Conclusions: The results showed a possible correlation between the presence of the cagA gene and the development of atrophic gastritis in the studied population.
Key words: Helicobacter pylori, virulence factors, cagA, DNAr 16S, detection, typing, Polymerase Chain Reaction, gastrointestinal diseases.
INTRODUCCIÓN
Helicobacter pylori es un microorganismo microaerófilo que coloniza e infecta la mucosa gástrica humana. La infección está distribuida mundialmente y su prevalencia varía según la región geográfica y de acuerdo con el nivel socioeconómico de la población.1 En Colombia aunque no se han establecido datos certeros sobre la infección con este patógeno, se ha descrito que causa más de 90 % de las úlceras duodenales y más de 80 % de las gástricas dentro de la población trabajadora.2
Se ha postulado que algunos genes de H. pylori, expresados diferencialmente entre cepas, pueden ser usados como marcadores de virulencia y su presencia se ha asociado con un aumento en el riesgo de desarrollar enfermedades gastroduodenales severas como gastritis crónica, úlcera péptica, cáncer gástrico y linfoma tipo MALT.3 Uno de estos es el gen asociado a citotoxina cagA, marcador de la isla de patogenicidad cag, cuya presencia se ha visto asociada a un aumento en el riesgo de desarrollar úlcera péptica, gastritis atrófica y carcinoma, por causa de que la proteína interrumpe algunas de las vías de transducción de señales presentes en las células epiteliales e induce la secreción de citoquinas proinflamatorias que van a facilitar la colonización y transmisión persistente del microorganismo.4
Teniendo en cuenta que la infección por H. pylori es muy frecuente en la población colombiana y que la presencia de ciertos genotipos podría estar asociada con la generación y desarrollo de enfermedades gastroduodenales, el diagnóstico y genotipificación del microorganismo se establecieron como la base para la realización de este estudio. De igual forma se buscó determinar si existen posibles asociaciones entre la presencia del gen de virulencia cagA y una de las enfermedades descritas o más, en la población colombiana analizada.
MÉTODOS
Pacientes: la población de estudio estuvo constituida por 60 pacientes a quienes se les había indicado la realización de una endoscopia como parte del estudio de un cuadro de dispepsia. Cumpliendo con los requerimientos éticos, la inclusión de pacientes en el estudio se dio tras la consecución del consentimiento informado escrito. Fueron tomadas 2 muestras de biopsia gástrica, una de la región antral y otra de una porción del estómago a criterio del médico.
Análisis histológico: este incluyó coloración de Giemsa para la visualización de bacilos tipo H. pylori en el moco foveolar y el diagnóstico patológico de las lesiones de la mucosa gástrica, siguiendo los parámetros de la clasificación Sydney modificada.5
Extracción de ADN: se realizó la extracción de ADN a partir de un fragmento de tejido del antro gástrico, usando el kit Aqua Pure Genomic DNA® de Bio-Rad, de acuerdo con las indicaciones del fabricante.
Análisis molecular mediante la reacción en cadena de la polimerasa (PCR: polymerase chain reaction)
Detección del microorganismo: la detección de H. pylori se realizó mediante la amplificación de un fragmento del gen ADNr 16S, utilizando los iniciadores ACT-1 y ACT-2 (tabla 1) y siguiendo el protocolo descrito previamente.6 Cada reacción se realizó en un volumen final de 25 µL, que contenía 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 2,5 mM de MgCl2; 0,2 mM de cada dNTP; 1 pmol/µL de cada iniciador; 1,25 U de Taq polimerasa (Invitrogen TM) y 135 ng de ADN. El programa de la PCR consistió en un paso de desnaturalización a 94 °C por 4 min, seguido por 38 ciclos de 94 °C por 1 min, 52 °C por 1 min y 72 °C por 1 min; finalmente una elongación a 72 °C por 10 min. Para las muestras que resultaron negativas para la detección del microorganismo, se realizó un control de la reacción, amplificando el gen de la b-globina humana.7
Genotipificación de H. pylori: la región 5' del gen cagA, fue el blanco de amplificación para la tipificación del microorganismo. La reacción se efectuó en un volumen final de 25 µL que contenía 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 2,0 mM de MgCl2; 0,2 mM de cada dNTP; 1 pmol/µL de cada iniciador (cagAR-cagAF) (tabla 1); 0,625 U de Taq Polimerasa (Invitrogen TM) y 72 ng de ADN extraído. El programa de la PCR utilizado fue desnaturalización inicial a 94 ºC por 4 min, 40 ciclos de 94 ºC por 30 s, 50 ºC por 45 s y 72 ºC por 45 s, seguido de una elongación a 72 ºC por 5 min.8
Visualización y análisis de los productos de amplificación: los productos de amplificación fueron separados mediante electroforesis en geles de agarosa 1,5 y 2 % (para el gen ADNr 16S; y cagA y b-globina humana, respectivamente), a 100 voltios durante 60 min. La detección y visualización de los fragmentos de ADN se realizó con bromuro de etidio (0,4 %) bajo luz UV, en un sistema de documentación de geles ChemiDoc System XRS® de Bio- Rad.
Análisis estadísticos: el análisis incluyó el cálculo de la sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo. La concordancia entre las pruebas diagnósticas fue determinada mediante el cálculo del índice Kappa de Cohen. Los resultados fueron procesados con el paquete estadístico SPSS 14.0TM para Windows. Se realizó un análisis de regresión multinomial para evidenciar la posible correlación entre la presencia del gen cagA y el desarrollo de enfermedad gástrica. Se consideraron significativos valores de p menores que 0,05.
RESULTADOS
La población de estudio estuvo conformada por 60 pacientes que a criterio del médico fueron sometidos a una endoscopia de vías digestivas altas, al presentar un cuadro de dispepsia. Estos fueron categorizados en 4 grupos de acuerdo con los resultados del examen histopatológico aplicado:5 Gastritis no atrófica (75 %), gastritis atrófica multifocal (7 %), metaplasia intestinal y displasia (7 %) y carcinoma (2 %) (tablas 2 y 3).
De las muestras analizadas, 37 (62 %) resultaron positivas para la amplificación del gen especie-específico ADNr 16S (tabla 2). Se calculó la sensibilidad (71 %), especificidad (58 %), valor predictivo positivo (78 %) y valor predictivo negativo (48 %) de la PCR, con el fin de comparar esta técnica de detección contra la prueba de oro (análisis histológico). De igual forma, se calculó el índice Kappa de Cohen y se obtuvo un resultado de 0,271 y un error estándar (EE) de 0,127.
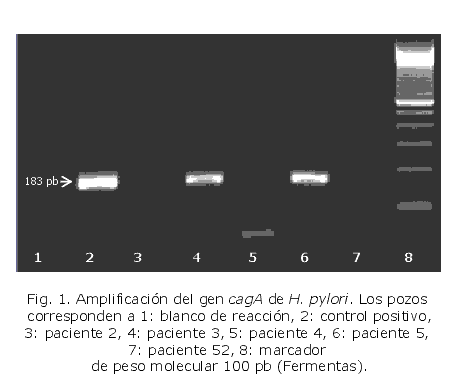
En cuanto a la tipificación, 16 de las 37 muestras analizadas (43 %), fueron positivas para el gen cagA (fig. 1 y tabla 2). Sin embargo, al analizar las muestras negativas para la amplificación del gen ADNr 16S (12 pacientes) se encontró que 2 de estas resultaron positivas para cagA (pacientes 6 y 43), las cuales reportan presencia de bacilos tipo H. pylori mediante el análisis histológico.
Teniendo en cuenta el cuadro clínico de los pacientes y el genotipo del microorganismo sobre la base del gen asociado a citotoxina cagA, se procedió a realizar una regresión multinomial, con el fin de determinar una posible asociación entre la presencia de cepas de H. pylori cagA+ y alguna de las enfermedades incluidas. Se obtuvo que la presencia del gen cagA podría estar asociada con el desarrollo de gastritis atrófica multifocal (estadístico de Wald = 2,77, g.l.= 1, p= 0,096, n= 53 pacientes), relación que está soportada gráficamente (fig. 2).
DISCUSIÓN
Por causa de que se ha reportado una prevalencia de H. pylori de 69 % en la población colombiana,9 toma importancia la identificación de marcadores de virulencia de la bacteria, que permitan la identificación de pacientes con una probabilidad de desarrollar enfermedades gástricas severas. Este estudio se centró en la detección y genotipificación de H. pylori, a partir de la extracción de ADN de tejido gástrico obtenido de pacientes que fueron remitidos a la unidad de endoscopia debido al cuadro clínico que presentaban, teniendo como blanco de amplificación los genes ADNr 16S y cagA.
La amplificación del gen especie-específico ADNr 16S reveló, que 62 % de los pacientes presentan el microorganismo, resultado que es congruente con lo reportado para Ibagué (60 %), ciudad que está incluida dentro de la población en estudio.9
Al comparar la detección molecular con la histológica (tablas 2 y 3) se observan discrepancias en cuanto a que, en 12 de los pacientes se evidencia el bacilo tipo H. pylori en la zona gástrica analizada usando la tinción histoquímica, mientras que la amplificación arrojó un resultado negativo. Este hecho podría explicarse teniendo en cuenta que las muestras extraídas para los análisis histológico y molecular no necesariamente provenían de la misma región del estómago, porque para el primero, la región era escogida a criterio del médico y de acuerdo con la localización de la afección, y para la detección molecular, del antro, que es la zona colonizada de forma preferencial por el microorganismo, debido a la ausencia de células secretoras de ácido.10
El análisis histológico incluyó tinción histoquímica con colorante Giemsa, el cual indica la presencia de bacilos, sin ser esta tinción realmente específica para H. pylori. Se ha reportado la presencia de 2 especies de Helicobacter en la cavidad gástrica humana: H. pylori y H. heilmannii, las cuales presentan características similares bajo tinción de Gram (espirilos gramnegativos), lo cual complica su distinción en el análisis microscópico. Al igual que H. pylori, H. heilmannii se ha asociado con la generación de gastritis en humanos; sin embargo, su frecuencia de infección es mucho menor que la de H. pylori.11,12
Diversos autores consideran la histología como prueba de oro para la detección de H. pylori en la mucosa gástrica,13,14 porque presenta valores de sensibilidad y especificidad altos en comparación a otras técnicas de diagnóstico (88-95 % y 90-95 %, respectivamente).15 La PCR como técnica alternativa para la detección del microorganismo fue evaluada, mediante la medición de la sensibilidad, especificidad, valor predictivo positivo (VPP) y valor predictivo negativo (VPN).
Al comparar los resultados de sensibilidad y especificidad de ambas técnicas (71 y 58 % para PCR; 88 y 90 % para histología, respectivamente) se observa, que la capacidad de la PCR para identificar a los individuos que no presentan el microorganismo, es baja. Esto se comprueba al calcular el VPN, el cual al ser bajo (48 %) indica que la probabilidad de que un resultado negativo por PCR indique la ausencia del patógeno no es confiable.
Por otro lado, se analizó la concordancia de las técnicas diagnósticas a través del cálculo del Índice Kappa de Cohen, el cual arrojó un valor de 0,271 con un error estándar de 0,127, que indica una concordancia aceptable y significativa,16 y que por consiguiente valida la PCR para la detección del microorganismo.
Diversos estudios han reportado que aproximadamente cerca de 60 a 70 % de los aislados de H. pylori provenientes de países de oeste son cagA+,17 datos que son congruentes con lo reportado para aislados colombianos.8 La caracterización de H. pylori sobre la base del gen cagA realizada en este estudio (43 %) mostró una frecuencia más baja a la reportada para Colombia (60-70 %).
Una vez genotipificadas las muestras, se procedió a determinar si la presencia del gen blanco podía estar relacionada con el desarrollo de alguna de las enfermedades incluidas en el estudio5 (gastritis no atrófica, gastritis atrófica multifocal, metaplasia intestinal y displasia, o carcinoma), porque se ha descrito que en las poblaciones de países de oeste, personas que portan cepas cagA+ presentan mayor riesgo de desarrollar gastritis atrófica, adenocarcinoma gástrico y úlcera péptica (especialmente duodenal).4
Al realizar un análisis de regresión multinomial para comprobar el efecto de la presencia del factor de virulencia cagA sobre la presentación de diversas enfermedades, se evidenció que el único efecto que alcanza un nivel de significancia marginal es el de la presencia del gen sobre la frecuencia de presentación de gastritis atrófica.
Aunque la cantidad de datos incluidos no fue suficiente para que el efecto no fuera marginal, en la figura 2 se muestra claramente el patrón entre la relación de la presencia del gen y la frecuencia de presentación; esto evidencia que el comportamiento para gastritis no atrófica y metaplasia intestinal y displasia (enfermedades 1 y 3) es similar y su presentación independiente de la presencia del gen. En contraposición, al observar la segunda enfermedad (correspondiente a gastritis atrófica multifocal) se evidencia claramente que el hecho de portar cepas cagA+ aumenta la frecuencia de presentación de la afección (fig. 2).
Para el caso de cáncer gástrico, que es otra de las enfermedades asociadas con la presencia del gen, no se puede inferir nada debido al bajo número de pacientes que presentaban la afección.
AGRADECIMIENTOS
Al Fondo de Investigaciones de la Facultad de Ciencias. Universidad de los Andes. Bogotá, Colombia.
A los participantes del proyecto: Universidad del Tolima, y miembros del Laboratorio de Diagnóstico Molecular y Bioinformática. Universidad de los Andes. Facultad de Ciencias. Bogotá, Colombia.
REFERENCIAS BIBLIOGRÁFICAS
1. Suerbaum S, Michetti P. Helicobacter pylori Infection. N Engl J Med. 2002;347(15):1175-86.
2. Bravo LE, Cortés A, Carrascal E, Correa P, Ordoñez N. Seroprevalencia de anticuerpos anti Helicobacter pylori en donantes de sangre de regiones colombianas con diferencias en la mortalidad por cáncer gástrico. Colomb Med. 2000;31:122-30.
3. Alm RA, Bina J, Andrews BM, Doig P, Hancock RE, Trust TJ. Comparative genomics of Helicobacter pylori: analysis of the outer membrane protein families. Infect Immun. 2000;68(7):4155-68.
4. Blaser MJ, Berg DE. Helicobacter pylori genetic diversity and risk of human disease. J Clin Invest. 2001;107(7):767-73.
5. Dixon MF, Genta RM, Yardley JH, Correa P. Classification and grading of gastritis. The updated Sydney System. International Workshop on the Histopathology of Gastritis, Houston 1994. Am J Surg Pathol. 1996;20(10):1161-81.
6. Thoreson ACE, Borre MB, Andersen LP, Elsborg L, Holck S, Conway P, et al. Development of a PCR-based technique for detection of Helicobacter pylori. FEMS Immunol Med Microbiol. 1995;10:325-34.
7. Saiki R, Scharf S, Faloona F, Mullis K, Horn G, Erlich H, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 1985;230(4732):1350-4.
8. Quiroga AJ, Cittelly DM, Bravo MM. Frecuencia de los genotipos babA2, oipA y cagE de Helicobacter pylori en pacientes colombianos con enfermedades gastroduodenales. Biomedica. 2005;25:325-34.
9. Bravo LE, Cortés A, Carrascal E, Jaramillo R, García LS, Bravo PE, et al. Helicobacter pylori: patología y prevalencia en biopsias gástricas en Colombia. Colomb Med. 2003;34:124-31.
10. Lundin A, Bjorkholm B, Kupershmidt I, Unemo M, Nilsson P, Andersson DI, et al. Slow genetic divergence of Helicobacter pylori strains during long-term colonization. Infect Immun. 2005;73(8):4818-22.
11. Joo M, Kwak JE, Chang SH, Kim H, Chi JG, Kim K, et al. Helicobacter heilmannii-associated Gastritis: Clinicopathologic Findings and Comparison with Helicobacter pylori-associated Gastritis. J Korean Med Sci. 2007;22:63-9.
12. Toyokawa T, Yokota K, Mizuno M, Fujinami Y, Takeneka R, Okada H, et al. Characterization of elongated Helicobater pylori isolated from a patient with gastric-mucosa associated lymphoid-tissue lymphoma. J Med Microbiol. 2004;53:207-12.
13. McNulty C and Wyatt J. Helicobacter pylori. J Clin Pathol. 1999;52:338-44.
14. Wilcox M, Dent T, Hunter J, Gray J, Brown D, Wight D, et al. Accuracy of serology for the diagnosis of Helicobacter pylori infection-a comparison of eight kits. J Clin Pathol. 1996;49(5):373-6.
15. Logan R,Walker M. ABC of the upper gastrointestinal tract. Epidemiology and diagnosis of Helicobacter pylori infection. BMJ. 2001;323:920-2.
16. Landis JR, Koch GG. An Application of Hierarchical Kappa-type Statistics in the Assessment of Majority Agreement among Multiple Observers. Biometrics 1977;33(2):363-74.
17. Chang Y, Wang L, Lee M, Cheng C, Wu C and Shiau M. Genotypic Characterization of Helicobacter pylori cagA and vacA from Biopsy Specimens of Patients with Gastroduodenal Diseases. Mt Sinai J Med. 2006;73(3):622-6.
Recibido: 17 de noviembre de 2007.
Aprobado: 21 de marzo de 2008.
Dra. Angie Patricia Molina Delgado. Universidad de los Andes. Bogotá, Colombia. Carrera 1ª N° 18A-10. Departamento de Ciencias Biológicas. Bloque J (Laboratorio J203). PBX +57(1) 339 4949 Ext. 3761-2773 Correos electrónicos: angi-mol@uniandes.edu.co; cjaramil@uniandes.edu.co; mdelgado@uniandes.edu.co; meboloza@gmail.com; aamezqui@uniandes.edu.co
Laboratorio de Diagnóstico Molecular y Bioinformática, Universidad de los Andes. Bogotá. Colombia.
Citogenética, Filogenia y Evolución de Poblaciones. Universidad del Tolima. Ibagué, Colombia
Grupo de Ecofisiología del Comportamiento y Herpetología. Universidad de los Andes. Bogotá, Colombia














