Introducción
El término Neurociencias es recogido en el diccionario como: “Ciencia que se ocupa del sistema nervioso o de cada uno de sus diversos aspectos y funciones especializadas”. Su empleo actual corresponde a la necesidad de integrar las contribuciones de las diversas áreas de la investigación científica y de las ciencias clínicas para la comprensión del funcionamiento del sistema nervioso. (Ferreira et al., 2019; Fothergill et al., 2019)
Los experimentos de neuroimagen dan como resultados datos complicados que se pueden organizar de distintas formas. Dichos datos son útiles para diagnóstico y estudio de padecimientos, por lo que su precisión y organización es importante, pues de ello depende la salud del paciente e incluso el desarrollo de nuevos terapias o medicamentos. Hasta el momento no hay consenso sobre cómo organizar y compartir los datos obtenidos en los experimentos de neuroimagen. Incluso dos investigadores que trabajan en el mismo laboratorio pueden optar por organizar sus datos de forma diferente. La falta de consenso (o de un estándar) conduce a malentendidos, a la pérdida de tiempo al reorganizar datos o la repetición de análisis por resultar incongruentes. Con la Brain Imaging Data Structure (BIDS), se describe una forma simple y fácil de adoptar para organizar los datos de neuroimagen y de comportamiento. (Norgaard et al., 2022) Si bien no existe un acuerdo oficial sobre que formato o estándar usar, BIDS es en la actualidad un estándar en auge y asciende la cantidad de grupos investigativos que lo adopta. Para establecer intercambios científicos fructuosos, se emplea BIDS y es incluso requisito en algunas revistas y plataformas como LORIS y OpenNeuro, las cuales aceptan y exportan datasets BIDS. (Silva et al., 2021)
Sin embargo, BIDS tuvo una creación muy reciente, en 2016, con respecto a otros formatos como DICOM, en 1993, por lo que la mayoría de los conjuntos de datos fueron generados en formatos que precedían a la creación de BIDS. Es donde los convertidores ganan su espacio generando datasets válidos para BIDS a partir de datasets que estaban en otros formatos: como DICOM, MINC, entre otros. (Contrera et al., 2022)
Se han desarrollado múltiples convertidores, siendo el dcm2niix (Gorgolewski et al., 2016) la base de muchos de ellos, por ser una herramienta para convertir datos de neuroimágenes al formato NIftTI. (Larobina & Murino, 2014) Cada convertidor requiere una configuración específica y es muy diversa en cada uno de ellos añadiendo un tiempo de preprocesamiento de la estructura de directorios y nombramiento de los mismos para poder luego pasar a la configuración de estos para un dataset en particular y después de configurados ejecutar la conversión, que en ocasiones se debe correr el convertidor en múltiples ejecuciones, todo esto agrega tiempo y dificultad al proceso de estandarización de bases de datos.
Bases de datos que no siguen estándares como BIDS dificultan el proceso de intercambio científico y el avance en estudios que están en la frontera del conocimiento. Además, el hecho de no poder realizar conversiones a gran escala fácilmente, dificulta el trabajar con grandes volúmenes de datos, almacenados en otros formatos como DICOM (Mustra et al., 2008) o MINC (Vincent et al., 2016), para neurocientíficos que no dominan las herramientas de conversión, puesto que la tarea es prácticamente imposible realizando la conversión por cada sujeto en cada sesión de los estudios de cualquier dataset. El objetivo del presente trabajo fue presentar una herramienta para la estandarización y estructuración de neurodatos de MRI y EEG, que permita solucionar las dificultades internacionales de propuestas previas
Métodos o Metodología Computacional
Se realizó una investigación de sistemas de conversión de la literatura, en el periodo comprendido a los años 2017 al 2022.
Para la ejecución de la presente investigación se sigue una estrategia explicativa y se emplearon los métodos: Análisis y Síntesis: análisis sobre los métodos de conversión de datasets existentes, determinando los elementos que los componen, las funciones que cumplen y su importancia. Determinar que parámetros se deben tener en cuenta para la selección del método a utilizar. Mediante el método comparativo: establecer la comparación entre los diferentes convertidores y poder establecer cuáles son los que van a utilizar para el desarrollo de la Herramienta de Conversión Multimodal.
La estructura para datos de imagen cerebral es un estándar para organizar, anotar, y describir los datos recogidos mediante pruebas de neuroimagen. Está basado en una estructura definida de archivos y carpetas, junto con documentación de los metadatos correspondientes en ficheros JSON con vocabulario controlado. (Pernet et al., 2019) BIDS aborda principalmente la heterogeneidad de organización de datos siguiendo los principios FAIR de búsqueda, accesibilidad, interoperabilidad y reutilización. (Martín-Forero et al., 2022)
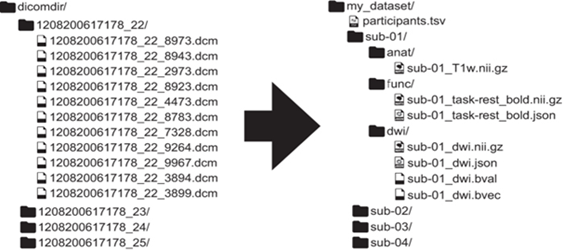
Dicho estándar describe una forma de organizar los datos y especificar metadatos (ver Fig. 1) para un subconjunto de experimentos de neuroimagen. Sigue una terminología simple pero cuidadosamente definida. Los nombres de archivo son formados con una serie de pares clave-valor y termina con un tipo de archivo, donde las claves y los tipos de archivo están predefinidos y los valores son elegidos por el usuario. Algunos aspectos del estándar son obligatorios, por ejemplo, cada conjunto de datos necesita tener al menos un directorio de materias. (Martín-Forero et al., 2022)
Algunos aspectos están regulados, pero son opcionales, por ejemplo, T1, no es necesario incluir escaneos ponderados, pero cuando estén disponibles, deben guardarse en un patrón de nombre de archivo particular especificado en el estándar. El estándar proporciona diccionarios de datos y estrictas convenciones de nomenclatura para datos de resonancia magnética estructural (T1w, T2w, etc.), de difusión y funcionales, así como datos de comportamiento y fisiológicos que lo acompañan. Además, definiciones claras de los términos utilizados en TSV y los archivos JSON se proporcionan junto con enlaces a DICOM.
Convertidores de datos de neurociencias
Dcm2bids: es un proyecto centrado en la comunidad. Su objetivo es ser una herramienta amigable y fácil de usar para convertir sus DICOMs. (Bedetti et al., 2021) Requiere dcm2niix previamente instalado y luego se puede usar el gestor de paquetes de Python (pip) para instalarlo. Es un convertidor que se ejecuta en la terminal de comandos. Tiene en su haber varias herramientas que ayudan al flujo de trabajo de la conversión al estándar BIDS como dcm2bids_scaffold para estructurar directorios y dcm2bids_helper para brindar ayuda e información sobre los comandos del convertidor y sus banderas. La conversión requiere además de un archivo de configuración. El archivo de configuración requiere que se cree una configuración por cada adquisición, permite que apliquemos una configuración a varios archivos por expresiones regulares en una llave denominada criteria.
Bidskit: intenta realizar un seguimiento de la Especificación BIDS lo más fielmente posible. Requiere de una instalación previa de dcm2niix y luego se puede usar el gestor de paquetes de Python (pip) para instalarlo. Bidskit se caracteriza por sus comandos muy intuitivos y poco complicados para el usuario final. Para llevar a cabo una conversión se debe correr bidskit en dos ocasiones la primera es para estructurar adecuadamente el árbol de directorios (en caso de ser preciso) y la segunda donde se ejecuta la conversión al estándar BIDS. (Lopez-Novoa et al., 2019)
BIDScoin: es una aplicación de Python de código abierto y fácil de usar que convierte ("coins") datasets de neuroimagen (sin procesar) a nivel de fuente en conjuntos de datos nifti / json / tsv que se organizan de acuerdo con el estándar BIDS. En lugar de depender de una lógica programática compleja para la identificación del tipo de datos de origen, BIDScoin utiliza un enfoque de mapeo para descubrir los diferentes tipos de datos de origen en su repositorio y convertirlos en tipos de datos de BIDS. (Zwiers et al., 2022)
Requiere tener instalado dcm2niix y el gestor de paquetes de Python (pip) se instalaría sin problemas.
BIDScoin requiere que el repositorio de datos de origen esté organizado de acuerdo con una estructura de sujeto/[sesión]/datos (la subcarpeta de la sesión es opcional). La carpeta de datos se puede estructurar de varias maneras:
Organización ‘seriesfolder’.
Organización ‘DICOMDIR’.
Organización plana de DICOM.
Organización PAR/REC.
Bidscoin ofrece dos herramientas para el preprocesamiento dicomsort y rawmapper. Dicomsort es capaz de convertir cualquiera de las 4 organizaciones antes mencionadas a la que es requerida por BIDScoin. rawmapper puede mostrarle una descripción general (map) de todos los valores de los atributos DICOM de interés en su conjunto de datos y, opcionalmente, se utiliza para cambiar el nombre de las subcarpetas de datos de origen.
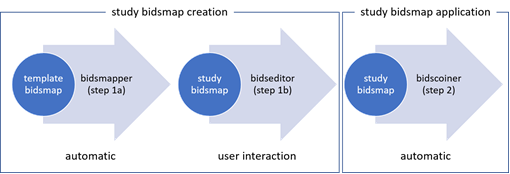
Luego de la preparación de los directorios de los datasets, viene el trabajo con el editor donde el usuario asegura los valores que son extraídos automáticamente por el convertidor, valores que utiliza un archivo de configuración que le servirá luego para ejecutar la conversión automática del dataset. Este proceso es mejor descrito en la documentación oficial del convertidor. (Kim et al., 2023)
Heudiconv: es un convertidor DICOM flexible para organizar datos de imágenes cerebrales en diseños de directorios estructurados. Permite diseños de directorios flexibles y esquemas de nombres a través de implementaciones heurísticas personalizables. Solo convierte los DICOM necesarios, no todo en un directorio. Puede mantener enlaces a archivos DICOM en el diseño del participante. Se integra con DataLad para colocar los datos convertidos y originales bajo el control de versiones de git/git-annex, mientras anota automáticamente los archivos con información confidencial (p. ej., anatómicos no desfigurados, etc.).( Tapera et al., 2021)
Similar a otros convertidores, heudiconv necesita ejecutar una vez antes de realizar la conversión. Durante esta conversión crea carpetas donde se almacenarán los datos convertidos y otras que le serán útil para la conversión, como el archivo de heurística, por defecto heuristic.py pero, puede tener otro nombre, incluso, asumir varios y cada uno se ajusta a un dataset específico.
El archivo de configuración de heudiconv es intuitivo siempre que los datos tengan correctos sus metadatos, es suficiente crear llaves en diccionario para cada modalidad de adquisición empleadas en el dataset. Heudiconv tomará todos los archivos de esa modalidad y les dará tratamiento de acuerdo a su modalidad y arrojando un dataset convertido con solo conocer las modalidades o secuencias empleadas en el dataset.
Resultados y discusión
La herramienta la conversión de base de datos de neurociencias al estándar BIDS para la plataforma BrainSSys. El propósito de su creación es responder a la necesidad de reducir el tiempo y la dificultad que conlleva el proceso de estandarización de bases de datos multimodales al estándar BIDS.
Tiene como objetivos específicos:
Permitir la conversión de un solo sujeto.
Permitir la conversión de varios sujetos dentro de un mismo estudio.
Permitir la integración de nuevos convertidores.
Se desarrolla la solución como una API (Application Programming Interface) REST (Representational State Transfer) proveedora de servicios, de modo que expone las funcionalidades de conversión al resto de servicios de plataforma mediante, y permitiendo además ser desplegada como software independiente de cualquier plataforma.
Todo se implementa bajo una arquitectura de plugins, con el objetivo de incluir nuevos convertidores como plugins para el sistema núcleo, permitiendo abarcar mayor cantidad de formatos y tipos de entrada, y de esta manera dar una solución más completa que las existentes. Esto permitiría que la herramienta no solo trabajase con formato DICOM como entrada sino, con formatos como MINC, Analyze y PAR/REC, por mencionar algunos. Constituyendo una solución distinta a las existentes en la literatura, enfocadas en un solo tipo de entrada.
Para ello se hace uso de FastAPI v0.79.0 como framework de desarrollo y Python como lenguaje de programación, uvicorn v0.18.2 como servidor de desarrollo. Siendo un conjunto de tecnologías ligeras para la solución, actualizadas y en constante mantenimiento.
Para desarrollar la herramienta siguiendo la arquitectura mencionada, se utilizó una implementación propia haciendo uso del patrón de diseño factory method y la librería de Python importlib para la importación dinámica de módulos o, en este caso, plugins. También se emplea los protocolos de la librería typing para establecer lo que sería un objeto Convesor, dicho objeto tiene un método para ejecutar la conversión y otro para retornar que parámetros acepta el método de conversión.
Para retornar los parámetros que acepta el método convert se hace uso de la clase BaseModel de pydantic para definir los parámetros que requiere la llamada o request a ese método de conversión en FastAPI, parámetros que son diferentes con cada convertidor, por lo que cada convertidor define su propio schema con el uso de la clase BaseModel.
Se hace empleo del factory method para la instanciación dinámica de convertidores, permitiendo instanciar un convertidor solo teniendo su nombre. La fábrica de convertidores define tres métodos:
register para el registro de nuevas funciones de creación con un nombre asociado a ellas,
unregister para eliminar una función de creación dado su nombre y,
create para crear instancias de un convertidor previamente registrado.
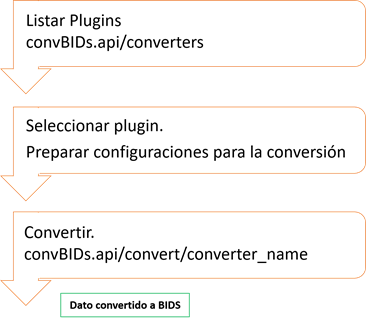
El proceso de conversión de datasets al estándar BIDS se compone de diferentes pasos. Estos varían en dependencia de cada convertidor, cada plugin maneja la lógica de conversión de manera distinta dentro de su método de conversión. Para el uso de la API se exponen cuatro endpoints:
Un flujo de trabajo común debería seguir el esquema de la figura 3. Dejando los endpoints de activar o desactivar plugins para la administración de la plataforma, aquellos usuarios con roles de administración y desarrollo.
Para la utilización del software es necesario contar como mínimo con 2 GB de RAM, procesador: Intel(R) Dual Core CPU @ 2.50 GHz y espacio en disco disponible de 4GB.
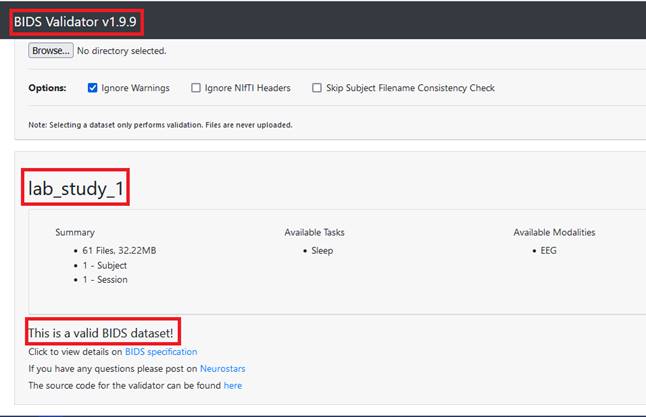
Como prueba de aceptación fundamental se acude al empleo de la herramienta online BIDS Validator, que su función principal es la de evaluar un conjunto de datos y determinar si es un dataset que cumple con las normativas de BIDS. Estas pruebas permiten validar si la conversión se ejecutó exitosamente. La figura 4 muestra los parámetros empleados para el método de conversión de la API desarrollada y la figura 5, los resultados de la evaluación de los resultados de la operación sobre un conjunto de datos de la base de datos internacional. (Kemp et al., 2000)
Estos resultados demuestran que se alcanzaron los objetivos que se requerían de la propuesta de solución. La API desarrollada permite convertir la información de uno o varios sujetos dentro de un mismo estudio e integrar nuevos convertidores como plugins, que ofrezcan otras vías de conversión; siendo el empleado para las pruebas un plugin integrado a la solución.
Conclusiones
El análisis de las dificultades existentes mostró las limitantes existentes para la estandarización de los datos de neurociencias y sus metadatos. Se analizaron los convertidores al estándar BIDS y se obtuvieron conversiones que permitieron identificar las potenciales fallas y, las funcionalidades que podían ser útiles para la propuesta de solución. Se implementó una arquitectura de plugins que permite la adición de nuevos plugins que modifiquen el comportamiento de la propuesta de solución. Se demostró que la herramienta ejecuta la conversión de datos de neurociencias al estándar BIDS, teniendo en cuenta los resultados en BIDS Validator.