Introducción
El aumento de la resistencia a los antimicrobianos constituye actualmente una grave amenaza para la salud. A escala mundial, se considera que 700,000 personas mueren cada año por infecciones resistentes a los antibióticos. Si no se hace nada, se prevé que causen anualmente millones de muertes; para 2050, la resistencia a los antimicrobianos y las infecciones vinculadas podría convertirse en una causa de muerte más común que el cáncer.1,2,3,4
En la actualidad las infecciones causadas por enterobacterias y pseudomonas aeruginosa multirresistentes productoras de betalactamasas de espectro extendido o de carbapenemasas, constituyen un peligroso problema de salud pues las opciones terapéuticas eficaces frente a estos patógenos son muy limitadas.
El crecimiento en el número de infecciones causadas por bacterias farmacorresistentes supera a la aparición de nuevos antibióticos, lo cual apunta hacia un futuro estremecedor.1,5,6,7,8
Un número creciente de enfermedades infecciosas graves está relacionado con la presencia de cepas bacterianas patógenas dentro de nuestro microbioma. Estas cepas se pueden diferenciar de las bacterias inofensivas al dirigirse a determinantes genéticos específicos que llevan en sus cromosomas o plásmidos.9,10,11
Recientemente, un grupo de investigadores del Instituto Tecnológico de Massachusetts (MIT, por sus siglas en inglés) con sede en Francia llamado Eligo Bioscience (antes conocido como Phagex) ha decidido lanzar al mercado un nuevo tipo de medicamento, los eligobióticos.12
La plataforma bioterapéutica de la empresa es capaz de realizar entregas de precisión al microbioma humano: la compleja y vasta comunidad de microbios presentes dentro y sobre el cuerpo.13
El microbioma se encuentra “en la interfaz de la salud y la enfermedadˮ, todavía no se dispone de una forma de intervenir selectivamente a nivel bacteriano.14,15
Los eligobióticos eliminarán las bacterias patógenas multirresistentes o, por ejemplo, la reconvertirán en productoras de medicamentos. Y todo ello gracias al uso de la técnica de edición genética conocida como CRISPR (Clustered Regularly Interspersed Short Palindromic Repeats), que utiliza virus bacteriófagos para infectar a las bacterias e insertarles circuitos de ADN sintético.
El sistema CRISPR así como su proteína asociada Cas (CRISPR- associated) se encuentran ampliamente distribuidos en el mundo microbiano, de hecho, están presentes en el 40 % de las especies bacterianas y en el 90 % de las arqueas reportadas.16,17,18
El sistema de repeticiones palindrómicas cortas agrupadas y regularmente espaciadas o CRISPR se refiere a un locus del cromosoma bacteriano donde residen unos genes que permite manipular el ácido desoxirribonucleico (ADN o DNA, por sus siglas en inglés) de cualquier organismo viviente sobre nuestro planeta.19,20
El nombre CRISPR identifica a una técnica o herramienta con la que se logran cambios específicos en los genomas y que ha funcionado exitosamente en diversos organismos, incluyendo recientemente el microbioma humano.21
La importancia biológica de este sistema radica en que se constituye como un sistema inmune adaptativo microbiano que responde a elementos de ADN invasores, como pueden ser plásmidos o virus.22,23,24
Recientemente se logró adaptar este sistema como una herramienta para la edición genética y su uso se ha extendido rápidamente debido a su simplicidad y gran precisión.
Se realizó una revisión actualizada sobre las causas y mecanismos de la resistencia a los antibióticos y la adaptación del sistema CRISPR/Cas para el desarrollo de innovadores antimicrobianos como parte esencial de una estrategia altamente específica en el tratamiento de infecciones producidas por bacterias resistentes.
Métodos
Se realizó una revisión documental, se empleó la bibliografía nacional e internacional especializada publicada en los últimos 5 años. Se utilizó el motor de búsqueda Google Académico y se consultaron artículos de libre acceso en las bases de datos Pubmed, SciELO, LILACS, CUMED y HINARI en el período comprendido entre marzo de 2020 hasta el mes de enero de 2021. Para la búsqueda se emplearon palabras como eligobióticos, resistencia a antibióticos, genes de resistencia a antibióticos, CRISPR/Cas, terapia génica, y sus equivalentes en inglés, según el descriptor de Ciencias de la Salud (DeCS). Fueron seleccionados 41 artículos (27 en idioma español, 14 en inglés) y (87,8 %) corresponden a los últimos 5 años.
Antimicrobianos
Los antimicrobianos han salvado innumerables vidas, pero lamentablemente tanto su uso apropiado como indiscriminado ha ido acompañado de la rápida aparición de cepas resistentes a múltiples fármacos.
Las bacterias pueden presentar resistencia a los antibióticos como resultado de mutaciones cromosomales e intercambio de material genético de otras bacterias o fagos (virus que utilizan bacterias para su desarrollo y reproducción), a través de mecanismos como25
Transformación: transferencia o incorporación por una bacteria de ADN libre extracelular procedente de la lisis de otras bacterias.
Transducción: transferencia de ADN cromosómico o plasmídico de una bacteria a otra mediante un bacteriófago (virus que infecta bacterias).
Transposición: movimiento de una sección de ADN (transposon) que puede contener genes para la resistencia a diferentes antibióticos y otros genes casetes unidos en equipo para expresión de un promotor en particular.
Conjugación: intercambio de material genético entre dos bacterias (donante y receptor), a través de una hebra sexual o contacto físico entre ambas.
La resistencia bacteriana puede ser natural o intrínseca y adquirida, y debe ser analizada desde varios puntos de vista (farmacocinético, farmacodinámico, poblacional, molecular y clínico).26
La resistencia natural o intrínseca es una propiedad específica de las bacterias, su aparición es anterior al uso de los antibióticos y tiene la característica de ser inherente a una especie en particular. La adquisición de material genético por las bacterias susceptibles a antimicrobianos de bacterias con resistencia ocurre a través de conjugación, transformación o transducción con transposones que a menudo facilitan la incorporación de genes de resistencia múltiple al genoma o plásmido.
La resistencia adquirida es un verdadero cambio en la composición genética de la bacteria y constituye un verdadero problema en la clínica. Existe un fenómeno conocido como tolerancia, considerado como un tipo de resistencia adquirida, aun cuando el microorganismo sigue sensible al medicamento. Los siguientes son los principales mecanismos de resistencia desarrollados por las bacterias: Bombas de eflujo o de excreción (BE) o expulsión del antibiótico del interior de la célula bacteriana (los genes y proteínas de las BE están presentes en todos los organismos, se localizan en el cromosoma o los plásmidos), modificación o inactivación del antibiótico mediante enzimas hidrolíticas, la modificación del sitio activo (modificación de PBP (penicilin-binding-protein) y modificación ribosomal), alteración o disminución de la permeabilidad de la membrana celular bacteriana, biofilmes o biopelículas y sobre-expresión del sitio blanco. Otros añaden la indiferencia al fármaco (las bacterias que se dividen no son sensibles al fármaco) y la persistencia (resistencia adaptativa o tolerancia fenotípica).
La tasa de crecimiento de la resistencia bacteriana supera a la creación de nuevos antibióticos, lo cual ha dado como resultado la necesidad imperante del desarrollo de terapias alternas a los antibióticos convencionales, ya sean dirigidos al patógeno o al huésped.1
Muchos antibióticos matan a las bacterias “buenasˮ de forma indiscriminada incluye aquellas que necesitamos para llevar a cabo múltiples funciones fisiológicas y más importantes aún, su uso contribuye a la aparición de infecciones farmacorresistentes y superbacterias.
El escenario que se vislumbra es tan aterrador que la Organización Mundial de la Salud lo ha calificado de peligroso y ha vaticinado que las infecciones bacterianas resistentes a los antibióticos serán una de las principales causas de muerte para 2050.
Actualmente el problema es más complejo, ya que el desarrollo de nuevos antibióticos ha descendido estrepitosamente desde la década de 1980, a pesar de iniciativas internacionales encargadas de promover la aparición de nuevos antimicrobianos. Existen impedimentos comerciales y científicos en el desarrollo de nuevos antibióticos. La industria farmacéutica ha desviado su interés hacia drogas de uso crónico, ya que el desarrollo de antimicrobianos supone un alto costo y poseen un tiempo de mercado inestable debido al desarrollo de resistencia en las bacterias.1,27
Las soluciones actuales como tratamiento adyuvante a la antibióticoterapia convencional incluyen vacunas, anticuerpos, la terapia con fagos, péptidos antimicrobianos, prebióticos, probióticos y simbióticos, que están diseñados para tener un impacto restaurador sobre las bacterias “buenasˮ, no obstante varían en efectividad.28,29,30,31,32,33
La compañía biofarmaceútica líder Eligo Bioscience está desarrollando una solución única: la primera plataforma bioterapéutica programable del mundo. La idea es tratar las enfermedades en su origen mientras se mejora la salud general del microbioma. La nueva técnica ha sido denominada “eligobióticosˮ.34
Los eligobióticos están más cerca de los nanobots biológicos que los medicamentos convencionales. Están hechos de ADN y proteínas y pueden ofrecer una carga útil terapéutica personalizada para tipos específicos de bacterias. Una vez que se han encontrado las bacterias objetivo, los eligobióticos pueden matarlas o convertirlas transitoriamente en productores de fármacos.35
En contraste con el enfoque de “todo o nada” de los antibióticos conocidos, la versatilidad de la plataforma conocida como Eligobiotics TM es capaz de entregar una carga útil en el organismo humano para matar selectivamente las bacterias patógenas y lograr el restablecimiento de un equilibrio saludable.
El equipo de investigadores de Eligo ha utilizado las herramientas de la biología sintética para crear una solución novedosa a un problema increíblemente complejo: cómo abordar, con extrema precisión, las causas fundamentales de las enfermedades asociadas al microbioma.
Eligo Bioscience ha encontrado una herramienta promisoria en la tecnología de edición del genoma CRISPR / Cas9 y se encuentra desarrollando ultraprecisos antibióticos que dejarán intacto el microbioma humano.
El sistema de repeticiones palindrómicas cortas agrupadas a intervalos regulares o CRISPR es parte de un sistema inmune adaptativo que los microorganismos desarrollaron para defenderse de la incorporación de material genético exógeno.
El sistema CRISPR-Cas9 es una herramienta de inmunidad adaptativa de las bacterias para defenderse del ataque de virus. Este sistema de inmunidad está mediado por una endonucleasa específica Cas9 que degrada al DNA invasor y posteriormente algunos fragmentos de la molécula degradada se almacenan para reconocer y eliminar secuencias similares en el futuro.36,37,38
Recientemente fue posible reprogramar este sistema para reconocer cualquier secuencia de DNA y realizar ediciones genéticas en una gran cantidad de organismos de manera altamente específica. Gracias a las distintas modificaciones de la Cas9, es posible usar el sistema CRISPR para aplicaciones limitadas solo por la imaginación.39
El sistema CRISPR/Cas ha sido adaptado para el desarrollo de una estrategia altamente específica en el tratamiento de infecciones producidas por bacterias resistentes a antimicrobianos.40
La empresa biofarmacéutica Eligo está en la búsqueda de la perfección de esta estrategia mediante el empaquetamiento de programas genéticos en vectores de administración altamente eficientes derivados de bacteriófagos. Estos «bioportadores» ya no contienen nada del ADN del fago original, no son replicativos, son estables a 4°C y pueden usarse para administrar cargas útiles genéticas en los microbiomas del intestino, la piel o los pulmones.
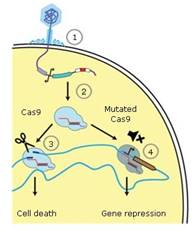
La nueva generación de bioterapéuticos elimina cepas bacterianas en función de su firma genética por los siguientes mecanismos (Fig).
un fagémido, plásmidos empaquetados dentro de una cápside vírica, a los que se les han incorporado los genes que codifican la nucleasa Cas y el sgRNA modificado para atacar a los genes de interés diseñado se adhiere a las bacterias e inyecta un circuito de ADN.
Cas9, tracRNA y crRNA se sintetizan y procesan inmediatamente y una vez que se ensambla el complejo Cas9 (nucleasa Cas9 + tracrRNA + sgRNA), escanea el genoma en busca de la secuencia objetivo en cuestión de minutos.
Si la secuencia objetivo se encuentra en el genoma bacteriano, Cas9 creará una ruptura de doble hebra en una secuencia genética de elección (factor de virulencia, gen de resistencia a antibióticos, etc.) que conducirá a una rápida muerte en la mayoría de las bacterias debido a la falta de sistemas de reparación del ADN.
Cuando se usa la versión mutada dCas9, la secuencia objetivo se puede silenciar.
Después del tratamiento algunas bacterias pudieran lograr sobrevivir, no contendrían los genes de resistencia a antimicrobianos contra los que estaba dirigida la nucleasa Cas.
Eligo en la actualidad desarrolla la primera plataforma bioterapéutica programable del mundo la cual utiliza las herramientas de la biología sintética para abordar los problemas más urgentes del mundo.
El método de Eligo emplea la Tecnología CRISPR / Cas9 que lo hace muy fácil para identificar y modificar hebras de ADN. Se inserta un par de “tijeras genómicasˮ en una cápside de bacteriófago que actúa como un vehículo de transporte. Dentro de las bacterias los eligobióticos buscan secuencias genéticas únicas y las cortan, destruyen los microbios eficazmente.
Su plataforma modular permite atacar la causa de las enfermedades mediante el desarrollo de una nueva generación de bioterapéuticos (los eligobióticos) que permiten la expresión de ARN terapéuticos, proteínas o moléculas pequeñas dentro de poblaciones microbianas objetivo. De esta forma, estos se pueden programar para diagnosticar, matar o controlar el metabolismo de las cepas bacterianas causales y, al mismo tiempo, preservar el resto del microbioma.
Los eligobióticos alcanzan intervenir con precisión a bacterias patógenas específicas que causan la infección, dejan ilesas a otras cepas inofensivas dentro del microbioma, asimismo, se puede programar para matar solo las bacterias resistentes a los antibióticos, dirigiéndose al ADN de estas.
Los patógenos bacterianos asociados con las epidemias de enfermedades humanas se ha propagado rápidamente; más pacientes mueren de infecciones por superbacterias cada año (y se prevé que las superbacterias serán más asesinas que el cáncer en 2050) y la línea de desarrollo de nuevos antibióticos es preocupantemente pobre. Una base de datos reciente enumera la existencia de más de 20,000 genes de resistencia potencial (genes r) de casi 400 tipos diferentes predichos a partir de las secuencias del genoma bacteriano disponibles.41
Durante el crecimiento los microorganismos se adaptan a su entorno. Si algo le impide crecer y propagarse, desarrollan nuevos mecanismos para resistir a los antimicrobianos cambiando su estructura genética. La reciente identificación de nuevos genes mediados por plásmidos que confieren resistencia a la colistina vaticina una crisis que se estima que causará 10 millones de muertes por año para 2050, resultará en una enorme morbilidad y eliminará más de 100 billones de USD de la economía mundial.42
Los eligobióticos han de exhibir perfiles de sensibilidad prometedores pues atacarán específicamente a las bacterias sin los efectos nocivos de los antibióticos. Igualmente permite la entrega de cargas útiles de ADN terapéutico sintético a poblaciones bacterianas del microbioma.
En el futuro se podrían utilizar diferentes cargas útiles para modular las respuestas inmunitarias, alterar el metabolismo de los fármacos e incluso crear una producción transitoria de fármacos.
El director ejecutivo de Eligo, el Dr. Xavier Duportet, expresó que: “…los antibióticos son armas de destrucción masiva: extremadamente poderosas pero imprecisas. Con los eligobióticos, podemos intervenir con precisión en el microbioma, dirigiéndonos a bacterias específicas para las intervenciones de nuestra elección. Al diseñar el microbioma en sí mismo con precisión de francotirador, podemos abordar la causa, no solo los síntomas, de las enfermedades asociadas a las bacterias”.12
Los eligobióticos y otras opciones como los péptidos antibiofilm o inhibidores de las bombas de eflujo no tienen productos disponibles en el mercado y siguen aún en fases de investigación.
Estos productos se encuentran en las primeras fases de desarrollo y todavía se tiene que demostrar su eficacia y seguridad. Según informes, en el mejor de los casos, los primeros productos estarán disponibles en los próximos 3 años.
Eligo ha confirmado que las últimas inversiones se destinarán a fortalecer la plataforma bioterapéutica y demostrar su valor en escenarios de la vida real. Los ensayos clínicos están en el horizonte y la compañía espera hacer crecer su equipo internacional de científicos, ingenieros y ejecutivos.
Eligo, es una empresa de biotecnología que es pionera en la ingeniería del microbioma y en el abordaje de las enfermedades asociadas al mismo. Un ejemplo lo constituye la aplicación de la tecnología Eligobiotics® de Eligo, para modificar con precisión la composición del microbioma de la piel para tratar o reducir el riesgo de desarrollar acné de moderado a severo.43
El acné es un trastorno inflamatorio crónico que afecta al 85 % de los adolescentes y adultos jóvenes de todo el mundo y tiene un impacto significativo en la salud física y mental a nivel mundial.
Eligo Bioscience líder mundial en terapia génica del microbioma está aprovecha los conocimientos recientes sobre el acné, muestran las bacterias de la piel que de otro modo serían beneficiosas, cuando expresan moléculas proinflamatorias, activan el sistema inmunológico y provocan la inflamación. El programa EB005 tiene como objetivo desarrollar eligobióticos de aplicación tópica para eliminar de manera precisa y selectiva estas cepas bacterianas proinflamatorias del microbioma, sin afectar al resto del microbioma de la piel.
Si se demuestra que es seguro y eficaz a lo largo de su desarrollo bajo la asociación con la empresa de atención médica global GSK (GlaxoSmithKline), este enfoque único tiene el potencial de cambiar el paradigma del tratamiento del acné al dirigirse específicamente a una de sus causas fundamentales.
Eligobiotics® es considerada una plataforma tecnológica patentada con la excelencia científica y las capacidades para llevar la innovación desde el laboratorio, a través del desarrollo clínico, y a los pacientes.
La Resistencia antimicrobiana se encuentra en un punto crítico, que la ubica dentro de los diez problemas de especial atención para la Organización Mundial de la Salud, organismo que lidera y ejecuta mancomunadamente los planes de acción para su combate con otros organizaciones e instituciones de salud, de alimentos y agricultura, los estados y las comunidades.
Hasta el momento, el uso de esta biotecnología para el tratamiento de infecciones bacterianas ha probado ser muy eficaz y tiene el potencial para el diseño de tratamientos personalizados, ya que puede utilizarse únicamente en las bacterias responsables de la infección o adaptarse para atacar genes de interés que codifican factores de resistencia a antimicrobianos, como las â-lactamasas de espectro extendido, o factores de virulencia, como los determinantes en la formación de biofilm.
Se concluye que Eligo en su investigación muestra que muchas enfermedades son inducidas por la microbiota humana, por tanto, conjeturemos lo que podríamos lograr si con terapia génica pudiéramos diseñar los microbiomas.
Sin dudas, un gran logro de las próximas tres décadas será el esclarecimiento de los enigmas de la microbiota y el microbioma humano, brinda así colosales medios terapéuticos que ineludiblemente modificarían la práctica médica para siempre.