Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Hematología, Inmunología y Hemoterapia
versión On-line ISSN 1561-2996
Rev Cubana Hematol Inmunol Hemoter v.24 n.1 Ciudad de la Habana ene.-abr. 2008
ARTÍCULOS ORIGINALES
Diagnóstico de linfomas cutáneos mediante detección de clonalidad por PCR-heterodúplex
Diagnosis of cutaneous lymphomas by clonicity detection through PCR-heteroduplex technique
DraC. Ana M. Amor VigilI; Dr. Rafael Matos BorgesII; DraC. Gisela Martínez AntuñaI
I Instituto de Hematología e Inmunología.
II Instituto Nacional de Oncología y Radiobiología.
RESUMEN
La detección de clonalidad en los síndromes linfoproliferativos mediante el estudio del reordenamiento de los genes de las inmuglobulinas y del receptor de células T, es utilizada para esclarecer si una proliferación o infiltrado de linfocitos es maligno o no. Este tipo de estudio es de particular utilidad en presencia de lesiones cutáneas cuyo origen linfoide o dermatológico resulta difícil de definir. Mediante la técnica de PCR-heterodúplex se estudiaron los genes de la cadena pesada de las inmunoglobulinas y de la cadena gamma del receptor de las células T, en 10 pacientes que presentaban manifestaciones dermatológicas atribuibles a algún tipo de linfoma cutáneo. Se observó reordenamiento clonal en 7 pacientes, lo cual permitió confirmar el diagnóstico de micosis fungoide y otros tipos de linfomas cutáneos. En 3 pacientes que no mostraron reordenamiento clonal, no fue posible confirmar por esta técnica un proceso linfoide de carácter maligno. Se demostró la utilidad del estudio cuando en presencia de una afección en la piel, es difícil diferenciar un proceso dermatológico de un síndrome linfoproliferativo con manifestaciones en piel.
Palabras clave: linfoma cutáneo, clonalidad, PCR-heterodúplex.
ABSTRACT
The clonicity detection in the lymphoproliferative syndromes by studying the rearrangement of the immunoglobulin genes and of the T-receptor cells is used to make clear if a proliferation or infiltrate of lymphocytes is malignant or not. This type of study is particularly useful in the presence of cutaneous lesions whose lymphoid or dermatological origin is difficult to define. By the PCR-heteroduplex technique, the genes of the immunoglobulin heavy chain and of the T-cell receptor chain were studied in 10 patients that presented dermatological manifestations attributable to some kind of cutaneous lymphoma. Clonal rearrangement was observed in 7 patients, which allowed to confirm the diagnosis of mycosis fungoides and other types of cutaneous lymphomas. It was not possible to confirm a lymphoid process of malignant character by this technique in 3 patients who did not show clonal rearrangement. The usefulness of the study was proved when in the presence of a skin affection, it was difficult to differentiate a dermatological process from a proliferative syndrome with cutaneous manifestations.
Key words: Cutaneous lymphoma, clonicity, PCR-heteroduplex technique.
INTRODUCIÓN
El estudio de clonalidad en los síndromes linfoproliferativos mediante el estudio del reordenamiento de los genes de las inmuglobulinas (Igs) y del receptor de células T (RCT), se utiliza para esclarecer si una proliferación o infiltrado de linfocitos es maligno o no. Sin embargo, es preciso destacar que la presencia de clonalidad no debe considerarse como el único criterio a tener en cuenta para establecer la malignidad de un proceso linfoproliferativo,1-3 y que la ausencia de esta no debe considerarse sinónimo de proceso benigno. Por ejemplo, algunos procesos inflamatorios cutáneos benignos como el liquen plano,4,5 la pitiriasis liquenoide6,7 y el liquen esclerótico atrófico,8 pueden mostrar reordenamiento clonal.
Además, ciertos linfomas surgen de células tan inmaduras que en ocasiones no han realizado aún el reordenamiento génico y por lo tanto, no es posible detectar la clonalidad mediante el estudio de este marcador.
En el caso específico de las afecciones de la piel, esta técnica es de gran utilidad para diferenciar los linfomas cutáneos de otras afecciones dermatológicas de origen benigno que en ocasiones presentan características similares.
El presente trabajo muestra el resultado del estudio de clonalidad del gen de la cadena pesada de las Igs (IgH) y del gen gamma del RCT (RCT ) por la técnica de PCR-heterodúplex en biopsias o aspirados de piel (BP o BAAF, respectivamente) de un número de pacientes cuyos signos clínicos en conjunto con el análisis histopatológico no permitían precisar el diagnóstico de un proceso linfoproliferativo maligno.
MÉTODOS
Se estudió un total de 10 pacientes adultos y niños de ambos sexos. En todos se sospechaba la presencia de un proceso linfoproliferativo de origen maligno con manifestaciones cutáneas, por lo que le fueron tomadas muestras de piel en los sitios afectados: 10 BP y 1 BAAF de piel. Las BP fueron sumergidas en solución salina fría inmediatamente después de disecadas.
Extracción y cuantificación de ADN
Para la extracción del ADN genómico, las BP fueron perfundidas con solución salina fría. La suspensión celular fue centrifugada a 3 000 rpm durante 10 min a 4 °C y vuelta a resuspender en 1 mL de solución de lisis que contenía tampón Tris-EDTA (10 mmol/L y 20 mmol/L, respectivamente) a pH 7,4; 2 L de SDS al 10 % y 2 L de proteinasa K (Merck) 10 mg/mL. Esta mezcla fue incubada a 55 °C durante 10 min y 1 h a 37 °C.
El ADN fue aislado mediante la técnica estándar de extracción con mezcla de solventes fenol-cloroformo-alcohol isoamílico (25:24:1) descrita por Sambouk y colaboradores,9 precipitado con etanol absoluto y disuelto en agua destilada estéril. Se calculó la concentración de ADN mediante la lectura de densidad óptica a 260 nm en un espectrofotómetro (UV-160A, Shimadzu).
Técnica de PCR o reacción en cadena de la polimerasa
En total se amplificaron 4 fragmentos de ADN: 2 correspondientes al gen IgH y 2 al gen RCT . Los oligonucleótidos o cebadores utilizados para el gen IgH fueron FR1 C y FR3, correspondientes a 2 sitios de la zona de segmentos variables y JHc para la zona de los segmentos de unión.10 Para el RCT se utilizaron V 1 y V 2 y J 1.3/2.3 respectivamente.11 Teniendo en cuenta que la mayoría de los síndromes linfoproliferativos cutáneos son de origen T, siempre se estudió el gen RCT en primer lugar y posteriormente, si este no mostraba reordenamiento clonal, se estudió el gen IgH.
Todas las PCRs se realizaron en iguales condiciones de reacción 7 en un termociclador (MiniCyclerTM, MJ Research, Inc.) y contenían 0,5 g de ADN genómico en presencia de: una solución tampón con 0,5 unidades de Taq ADN polimerasa (Promega Corp.), una mezcla 100 pM de nucleótidos y 12,5 pmoles de los cebadores correspondientes a cada fragmento, en un volumen total de 50 L. Se utilizaron 3 programas de amplificación, uno para cada fragmento del IgH y un tercero común para ambos fragmentos del RCT. En paralelo a las muestras se amplificó en cada caso un control positivo, un control negativo compuesto por un pool de ADN genómico de 5 personas sanas y un control de los reactivos. Para comprobar el resultado de la amplificación, el producto resultante de la PCR se analizó por electroforesis en gel de agarosa al 2 % con bromuro de etidio como marcador de fluorescencia.
Análisis por heterodúplex
Conocida la presencia de fragmentos de ADN, se provocó la formación de heterodúplex. Para esto el producto de la PCR fue desnaturalizado a 94 °C durante 5 min, incubado 1 h a 50 °C y finalmente enfriado en baño de hielo durante 5 min.12 Al finalizar la formación de heterodúplex, las muestras se analizaron por electroforesis en un gel de poliacrilamida (poliacrilamida-bisacrilamida, 29:1) no desnaturalizante, al 6 %, en tampón TAE (Tris-ácido acético-EDTA). El patrón de bandas fue visualizado a la luz UV con bromuro de etidio como marcador de fluorescencia.
Esta técnica facilita el análisis del resultado, ya que en caso de existir clonalidad se observan bandas intensas y definidas, mientras que en muestras policlonales se observa fluorescencia a través de todo el trayecto de la corrida electroforética sin definición de bandas, o bien no se observa fluorescencia si el material es muy escaso.
RESULTADOS
Los resultados del estudio del reordenamiento del gen IgH y del gen RCT mediante la técnica de PCR-heterodúplex se presentan en la tabla.
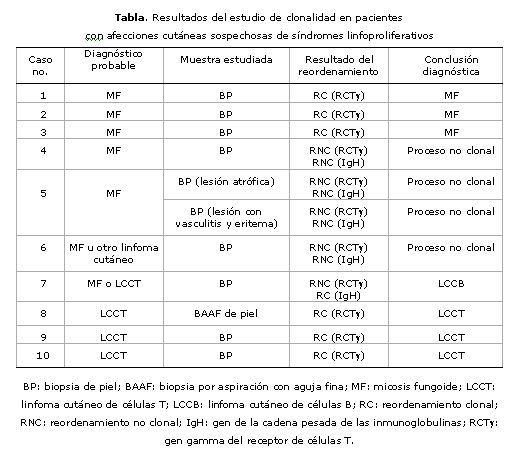
Los primeros 3 pacientes (casos 1-3) fueron enviados como posible MF y reordenaron de manera clonal para el gen RCT.
Otros 3 casos enviados también con diagnóstico de posible MF, no presentaron reordenamiento clonal para el RCT ni para el IgH. En uno de estos (caso 5) se estudiaron 2 BP correspondientes a lesiones con características diferentes, pero el estudio molecular, como se mencionó, no mostró reordenamiento clonal. En estos 3 casos, el estudio molecular del reordenamiento de los genes amplificados por PCR,no pudo demostrar la presencia de infiltración clonal de linfocitos.
Seguidamente, en un paciente enviado como posible MF o linfoma cutáneo de células T (LCCT), no se observó reordenamiento clonal del gen RCT, pero sí del IgH.
Los 3 últimos enfermos, uno de ellos estudiado en BAAF de piel y 2 en BP, que fueron enviados para estudio como posibles LCCT, mostraron reordenamiento clonal del gen RCT.
DISCUSIÓN
En ocasiones es difícil diagnosticar un linfoma cutáneo analizando solamente los signos clínicos del paciente y el resultado histológico de la biopsia.13 Desde el punto de vista clínico, existen varias dermatosis inflamatorias que simulan un linfoma, particularmente a la MF.1 Por otra parte, en el estudio histológico no siempre se observan características específicas de linfomas cutáneos, y el criterio de diagnóstico basado en ellas es, por lo tanto, impreciso. Además, otras características histológicas que se aprecian con frecuencia en los linfomas son también comunes a procesos dermatológicos y el diagnóstico de linfoma basado en ellas se hace inespecífico.14 Otro factor que introduce dudas es la variabilidad inter e intra observador, es decir, de los patólogos, con respecto a dictaminar de manera precisa la presencia de linfoma cutáneo.15
La determinación de clonalidad por PCR-heterodúplex con vistas a determinar si una población de linfocitos infiltrada en piel es maligna o no, se ha convertido en una herramienta importante para diferenciar procesos dermatológicos de síndromes linfoproliferativos cutáneos.1,13,16
En un grupo de pacientes atendidos en el Servicio de Dermatología del Instituto Nacional de Oncología y Radiobiología y que no respondían a tratamientos propios de procesos cutáneos, se sospechó la presencia de algún tipo de linfoma cutáneo y se les realizó el estudio de clonalidad por PCR-heterodúplex del gen IgH y del gen RCT.
Tres de estos pacientes fueron enviados como posible MF y mostraron reordenamiento clonal para el gen RCT, lo cual estuvo en concordancia con el análisis histológico que mostraba características propias de estos procesos, pero que no habían sido totalmente concluyentes. En los 3 casos se pudo llegar al diagnóstico de MF después de conocido el resultado molecular.
Otro paciente, enviado como posible MF o linfoma cutáneo de células T (LCCT), no mostró reordenamiento clonal del gen RCT , pero sí del IgH. El resultado del estudio de clonalidad permitió concluir que se trataba de un linfoma cutáneo de células B (LCCB). Este es un ejemplo de la utilidad del PCR-heterodúplex para definir la línea celular afectada en un proceso linfoproliferativo clonal, ya que, aunque inicialmente se pensaba que se trataba de un proceso linfoproliferativo de células T, el reordenamiento clonal del gen IgH permitió definir la naturaleza B del proceso.
En otros 3 pacientes que fueron enviados para estudio como posibles LCCT, se pudo confirmar que se trataba de este tipo de linfoma al observarse reordenamiento clonal del gen RCT.
Se conoce que en los linfomas cutáneos la línea celular afectada con mayor frecuencia es la T (60-70 %), mientras que los linfomas cutáneos de origen B aparecen entre el 30 y 40 %.17 En el presente estudio se encontró solo un proceso de origen B de un total de 7 que reordenaron, lo que representa el 14 % de linfoma tipo B, porcentaje inferior al señalado para este tipo de linfoma. Al respecto, debe tenerse en cuenta que se estudió un número muy pequeño de pacientes, y que por lo tanto, no es de esperar que la proporción B/T reportada se reproduzca en el grupo analizado.
Por último, en 3 de los procesos sospechosos de linfoma, no se encontró reordenamiento clonal en ninguno de los genes estudiados y no pudo confirmarse que estuvieran relacionados con algún tipo de linfoma. En cuanto a la obtención de un resultado negativo en este tipo de estudio, debe señalarse que esto no es sinónimo de proceso benigno, puesto que los genes aquí estudiados aunque son los que con más frecuencia aparecen reordenados, no son los únicos que lo hacen.17 Otro factor que conduce a un falso resultado negativo, son las mutaciones somáticas que en ocasiones ocurren en el sitio de unión de los cebadores. Cuando esto sucede, los cebadores no pueden unirse con el segmento de gen complementario para el cual fueron diseñados, y no es posible detectar la presencia de una población clonal de linfocitos aunque esta se encuentre presente.17 También debe tenerse en cuenta que ciertos precursores de linfoma como los linfomas linfoblásticos, pueden ser tan inmaduros que no desarrollen reordenamiento génico18 y que los linfomas de células T/NK no muestran regularmente reordenamiento del RCT,19,20 por consiguiente, en estos casos tampoco sería posible detectar clonalidad celular.
Un trabajo conjunto entre 22 laboratorios de Europa para estandarizar los protocolos de PCR en los estudios de clonalidad, ha diseñado nuevos cebadores y protocolos de trabajo, entre cuyos objetivos se encuentra el de minimizar la incidencia de falsos negativos.17 Los resultados obtenidos con su aplicación denotan un mayor porcentaje de resultados positivos y avalan su introducción como herramienta diagnóstica en el estudio de los síndromes linfoproliferativos.21-24
REFERENCIAS BIBLIOGRÁFICAS
1. Alessi E, Coggi A, Venegoni L, Merlo V, Gianotti R. The usefulness of clonality for the detection of cases clinically and/or histopatologically not recognized as cutaneous T-cell lymphoma. Dermatology 2005;153:368-71.
2. Sandberg Y, Heule F, Lam K, Lugtenburg P, Wolvers-Tettero ILM, van Dongen JM, et al. Molecular immunoglobulin/T-cell receptor clonality analysis in cutaneous lymphoproliferations. Experience with the BIOMED-2 standardized polymerase chain reaction protocol. Haematologica 2003;88:659-70.
3. Cerroni L, Arzberger E, Ardigo M, Pütz B, Kerl H. Monoclonality of intraepidermal T lymphocytes in early mycosis fungoides detected by molecular analysis after laser-beam-based microdissection. J Invest Dermatol 2000;114:1154-7.
4. Sander CA, Kind P, Flaig M. Genotypic analysis in cutaneous lymphoproliferative disease: A reliable test? J Cutan Pathol 1998;5:511-4.
5. Schiller PI, Flaig MJ, Puchta U, Kind P, Sander CA. Detection of clonal T cells in lichen planus. Arch Dermatol Res 2000;292:568-9.
6. Dereure O, Levi E, Kadin ME. T-cell clonality in pityriasis lichenoides et varioliformis acuta: A heteroduplex analysis of 20 cases. Arch Dermatol 2000;136:1483-6.
7. Shieh S, Mikkola DL, Wood GS. Differentiation and clonality of lesional lymphocytes in pityriasis lichenoide chronica. Arch Dermatol 2001;137:305-8.
8. Lukowsky A, Muche JM, Sterry W, Audring H. Detection of expanded T cell clones in skin biopsy sample of patients with lichen sclerosus et atrophicus by T-cell receptor gamma polymerase chain reaction assays. J Invest Dermatol 2000;115:254-9.
9. Sambouk J, Fritsch E, Maniatis T. Molecular cloning. A laboratory manual. Vol. III. New York: Cold Spring Harbor Laboratory Press; 1989; E. 3.
10. Aubin T, Davi F, Nguyen-Salomon F, Leboeuf D, Debert C, Taher M, et al. Description of a novel Fr1 IgH PCR strategy and its comparison with three other strategies for the detection of clonality in B cell malignancies. Leukemia 1995;9:471-9.
11. Pongers-Willemse MJ, Seriu T, Stolz F, d´Aniello E, Gameiro P, Pisa P, et al. Cebadores and protocols for standardized detection of minimal residual disease in acute lymphoblastic leukemia using inmunoglobulin and T cell receptor gene rearrangements and TAL1 deletions as PCR targets. Report of the BIOMED-1 Concerted Action: Investigation of minimal residual disease in acute leukemia. Leukemia 1999;13:110-8.
12. Bottaro M, Berti E, Biondi A, Migone N, Crosti L. Heteroduplex analysis of T-cell receptor gene rearrangements for diagnosis and monitoring of cutaneous T-cell lymphomas. Blood 1994;11:3271-8.
13. Bergman R. How useful are T-cell receptor gene rearrangement studies as an adjunt to the histopathologic diagnosis of mycosis fungoides. Am J Dermatopathol 1999;21:498-502.
14. Bachelez H, Bioul L, Flageul B, Baccard M, Moulonguet-Michau I, Verola O, et al. Detection of clonal T-cell receptor gene rearrangements with the use of the polymerase chain reaction in cutaneous lesions of mycosis fungoides and Sézary syndrome. Arch Dermatol 1995;131:1027-31.
15. Liebmann RD, Anderson B, McCarthy KP, Chow JWM. The polymerase chain reaction in the diagnosis of early mycosis fungoides. J Pathol 1997;182:282-7.
16. Lukowsky A, Marchwat M, Sterry W, Gellrich S. Evaluation of B-cell clonality in archival skin biopsy samples of cutaneous B-cell lymphoma by inmunoglobulin heavy chain gene polymerase chain reaction. Leuk Lymphoma 2006;47:487-93.
17. Van Dongen JJM, Langerak AW, Brüggemann M, Evans PAS, Hummel M, Lavender FL, et al. Design and standardization of PCR cebadores and protocols for detection of clonal inmunoglobulin and T-cell receptor gene recombinations in suspect lymphoproliferations: Report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia 2003;17:2257-317.
18. Sander CA, Flaig MJ. Morphologic spectrum of cutaneous B-cell lymphomas. Dermatol Clin 1999;17:593-9.
19. Jaffe ES, Chan JKC, Su IJ, Mori S, Feller AC, Ho FC. Report of the workshop on nasal and related extranodal angiocentric T/natural killer cell lymphomas. Definition, differential diagnosis, and epidemiology. Am J Surg Pathol 1996;20:103-11.
20. Weiss LM, Picker LJ, Grogan TM, Warnke RA, Sklar J. Absence of clonal beta and gamma T-cell receptor gene rearrangements in a subset of peripheral T-cell lymphomas. Am J Pathol 1988;130:436-42.
21. Droese J, Langerak AW, Groenen PJ, Bruggemann M, Neumann P, Wolvers-Tetero IL, et al. Validation of BIOMED-2 multiplex PCR tubes for detection of TCRB gene rearrangements in T-cell malignancies. Leukemia 2004;9:1531-8.
22. Langerak AW, Molina TJ, Lavender FL, Pearson D, Flohr T, Sambade C, et al. Polymerase chain reaction-based clonality testing in tissue samples with reactive lymphoproliferations: Usefulness and pitfalls. A report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia 2007;2:222-9.
23. Bruggemann M, White H, Gaulard P, Garcia-Sanz R, Gameiro P, Oeschger S, et al. Powerful strategy for polymerase chain reaction-based clonality assessment in T-cell malignancies Report of the BIOMED-2 Concerted Action BHM4 CT98-3936. Leukemia. 2007;2:215-21.
24. Van Krieken JH, Langerak AW, Macintyre EA, Kneba M, Hodges E, Sanz RG, et al. Improved reliability of lymphoma diagnostics via PCR-based clonality testing: report of the BIOMED-2 Concerted Action BHM4-CT98-3936. Leukemia. 2007;2:201-6.
Recibido: 15 de diciembre del 2007.
Aprobado: 3 de enero del 2008.
DraC. Ana M. Amor Vigil.
Instituto de Hematología e Inmunología. Apartado Postal 8070, Ciudad de La Habana, CP 10800, Cuba. Tel (537) 6438268, 6438695. Fax (537) 6442334. e-mail: ihidir@hemato.sld.cu














