Introducción
La enfermedad de Wilson (EW, MIM 277900) presenta un patrón de herencia autosómico recesivo. Constituye un problema de salud mundial. Se caracteriza por la acumulación de cobre, en diferentes órganos, tales como, hígado, cerebro, córnea, entre otros, como consecuencia del transporte defectuoso de cobre en la célula. Existen diversas manifestaciones clínicas asociadas a la EW y el diagnóstico clínico de esta enfermedad es complejo.1 Se caracteriza por daños en el hígado, que pueden ser muy variables, desde la elevación de los niveles séricos de transaminasas hasta hepatitis fulminante, cirrosis hepática. Los pacientes con EW también pueden presentar afectaciones en el cerebro, que se manifiestan como temblores, distonía; también trastornos psiquiátricos, tales como, depresión, problemas en el lenguaje, tendencias suicidas, entre otras.
La causa molecular que la provoca la EW son las mutaciones en el gen atp7b, el cual presenta 20 intrones y 21 exones que codifican para la proteína ATP7B, transportadora de cobre en el hepatocito. Se han identificado más de 130 polimorfismos que se distribuyen en todo el gen atp7b y en los intrones. Los exones más polimórficos son: 2, 3, 8 y 16.2
El polimorfismo p.L456V se localiza en el exón 3 del gen atp7b. Este se ha identificado en disímiles poblaciones, en países como China, Rusia, Egipto y Venezuela.3,4,5,6 Este polimorfismo de un solo nucleótido (SNP) se utiliza en la construcción de los haplotipos en familias donde haya al menos un paciente con diagnóstico clínico certero de la EW y posibilita la realización del diagnóstico molecular en los mismos.6
Para la determinación del espectro de polimorfismos en el gen atp7b, se requiere una apropiada tecnología de cribaje. Una de las técnicas más utilizadas para este propósito es el Polimorfismo Conformacional de Simple Cadena (SSCP). Se basa en la relación que existe entre la movilidad electroforética de la simple cadena de ADN y su estructura tridimensional. Un cambio en la secuencia nucleotídica de la cadena de ADN provoca un cambio conformacional, que a su vez, induce a un cambio de movilidad en la electroforesis detectable por esta técnica. Con la utilización de la técnica de SSCP permite identificar en las muestras de ADN cambios conformacionales diferentes a la variante normal que pueden corresponder a polimorfismos o mutaciones.7
Objetivo
Identificar los cambios conformacionales en el exón 3 y detectar polimorfismos en pacientes cubanos con diagnóstico clínico presuntivo de la EW.
Materiales y Métodos
Se realizó un estudio descriptivo, en el Centro Nacional de Genética Médica, durante el período 2007-2013, que incluyó 105 pacientes (43 mujeres y 62 hombres) con diagnóstico clínico de EW, los cuales asistían a las consultas en el Instituto Nacional de Gastroenterología. Estos pacientes dieron su consentimiento para participar en la investigación, de acuerdo con los principios éticos de la Declaración de Helsinki. Esta investigación fue aprobada por el Consejo Científico y el Comité de Ética del Centro Nacional de Genética Médica y el Instituto Nacional de Gastroenterología.
Los casos se seleccionaron por los siguientes criterios:
Criterios de inclusión: Individuos que se diagnosticaron con EW, según Ferenci y col, 20038 y que estos brindaran su disposición de participar en esta investigación, lo cual se evidencia a través de la firma del consentimiento informado.
Criterios de exclusión: Pacientes con EW que no estuvieran de acuerdo con participar en este estudio.
Criterio de salida: Quienes manifestaran su deseo de retirar su participación en la investigación, cualquiera que fuera el momento, aún después de haber dado su autorización previa.
Las variables analizadas fueron: frecuencia alélica del polimorfismo p.L456V, cambio conformacional a) para la variante normal, cambio conformacional b) para la presencia del polimorfismo p.L456V en estado heterocigótico y el cambio conformacional c) en estado homocigótico. Las manifestaciones clínicas fueron clasificadas en: hepáticas, neurológicas, psiquiátricas y sus combinaciones.
La evaluación de las manifestaciones clínicas fue realizada por un equipo multidisciplinario (gastroenterólogos, genetistas, neurólogos y bioquímicos, psiquiatras), se siguieron los criterios establecidos de diagnóstico de la enfermedad.
Se seleccionó el exón 3 del gen atp7b, para la detección de cambios conformacionales y la identificación de polimorfismos. A todos los pacientes se les tomó una muestra de sangre y se extrajo el ADN mediante el método de precipitación salina9 a partir de 10 mL de sangre periférica con ácido etildiaminotetraacético, EDTA, (56 mg/mL). Las condiciones para la amplificación del exón 3 mediante la técnica de Reacción en Cadena de la Polimerasa (PCR), las condiciones fueron: 100ng de ADN, 10(F) 5'-AGT CGC CAT GTA AGT GAT AA-3' y (R) 5'-CTG AGG GAA CAT GAA ACA A-3', 1mM de dNTPs (Boehringer), 10X tampón PCR, 15mM de MgCl2, 1U de Taq polimerasa (Amplicen), en un volumen de 25 μL.
Posteriormente se realizó la electroforesis SSCP. Se mezcló 3,5 μL con una solución de parada de Bromofenol azul (0,05% BFA, 10mM NaOH, 95% formamida, 20 mM EDTA) y 1 μL del producto amplificado, en un volumen final de 7μL. Se aplicó en un gel de acrilamida comercial (GeneGel Excel 12,5/24 Kit). La visualización del ADN se realizó por el método de tinción con plata, siguiendo las instrucciones del juego comercial: kit PlusOne DNA Silver Staining (Amersham Biosciences, 2007).
El polimorfismo p.L456V fue determinado por comparación de las corridas electroforéticas mediante la técnica de SSCP de controles positivos heterocigóticos y homocigóticos del polimorfismo p.L456V con el producto amplificado obtenido de la técnica de PCR del exón 3 del gen atp7b.
Los controles positivos fueron las muestras de ADN de los pacientes heterocigóticos y los homocigóticos para el polimorfismo p.L456V que se donaron por investigadores de los laboratorios Ospedale Regionale per le Microcitemie, Cagliari, en Italia y el Hospital Universitario de Munster, en Alemania.
Se calculó la media y desviación estándar de la edad de los pacientes en años. Se determinó la frecuencia del polimorfismo p.L456V como: Número de alelos que presenta el polimorfismo p.L456V/Número de alelos totales. Se expresó en porcentaje.
Resultados
Los pacientes que presentaron el polimorfismo p.L456V, la edad fueron de 31,6 años con una desviación estándar de 14 años. El intervalo fue de 11 años (mínimo) a 58 años (máximo).
Los pacientes estudiados que mostraron el polimorfismo p.L456V estaban distribuidos en 10 provincias y el municipio especial Isla de la Juventud. (Tabla).
Tabla. Representación por provincias y en el municipio especial de los pacientes con la EW
| Provincias | Pacientes |
|---|---|
| Pinar del Río | 12 |
| Isla de la Juventud | 7 |
| La Habana | 27 |
| Mayabeque | 5 |
| Matanzas | 2 |
| Cienfuegos | 1 |
| Villa Clara | 2 |
| Santi Spíritus | 2 |
| Camagüey | 1 |
| Santiago de Cuba | 3 |
| Guantánamo | 2 |
Se identificó los cambios conformacionales denominados b y c con el uso de la técnica SSCP. (Figura 1). El cambio conformacional denominado a correspondió a la variante normal. El cambio conformacional b y c correspondieron a la presencia del polimorfismo p.L456V estado heterocigótico y homocigótico respectivamente.

Fig. 1 SSCP del exón 3 del gen atp7b en pacientes y controles positivos que presentan el polimorfismo p.L456V.
Se detectaron 42 pacientes (40%) que presentaron el cambio conformacional b , 22 pacientes (21%) con el cambio conformacional c y 41 pacientes (39%) con el cambio conformacional a . La presencia del polimorfismo p.L456V es en un total de 64 pacientes. (Figura 2).
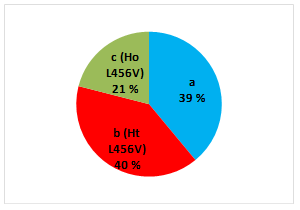
Fig. 2 Frecuencia de los cambios conformacionales del exón 3 y el porcentaje. Los cambios conformacionales denominados b y c fueron pacientes heterocigóticos y homocigóticos para el polimorfismo p.L456V respectivamente.
De los 105 pacientes cubanos con diagnóstico clínico presuntivo de la EW, se identificaron 42 pacientes heterocigóticos y 22 homocigóticos para el polimorfismo p.L456V por la técnica SSCP. La frecuencia alélica del polimorfismo p.L456V en pacientes cubanos estudiados en esta investigación fue de 41%.
El polimorfismo p.L456V es consecuencia del cambio de Guanina por Citocina que provoca un cambio del aminoácido Leucina por Valina en la posición 456 de la proteína ATP7B. Este polimorfismo se localiza entre el dominio de unión al cobre 4 y 5. El cambio provocado por esta variante polimórfica no afecta la función de la proteína transportadora de cobre, ATP7B, en humanos y se identifica en diversas poblaciones con una frecuencia mayor que 1%.3-5
Entre las principales manifestaciones clínicas en los pacientes que presentaron el polimorfismo p.L456V, fueron más frecuentes las hepáticas (78%), a continuación de las manifestaciones mixtas: hepáticas y neurológicas (11%), las neurológicas (9 %) y las mixtas neurológicas y psiquiátricas (2%).
Se identificó en cuatro pacientes con el polimorfismo p.L456V la presencia de los anillos de Kayser-Fleischer, lo que representa 6,3% de los pacientes que se estudiaron, el mismo constituye un criterio de diagnóstico clínico de la enfermedad. Las manifestaciones clínicas de los mismos eran dos pacientes con manifestaciones hepáticas y neurológicas, un paciente con manifestaciones neurológicas y un paciente con manifestaciones neurológicas y psiquiátricas.
Discusión
La edad de los pacientes con el polimorfismo p.L456V es similar a lo que se informa en la literatura internacional.1
En la distribución por provincias de los pacientes con el polimorfismo p.L456V se observó que el mayor número de pacientes era de La Habana y Pinar de Río. La mayoría de los pacientes con este polimorfismo se concentró en la región occidental del país.
Se han informado más de 130 polimorfismos en el gen atp7b en pacientes con la EW. La frecuencia del polimorfismo p.L456V obtenida en este estudio es similar a lo que se informa en China,10 y como se esperaba, es mayor que 1%. En estudios posteriores se analizará el impacto de este polimorfismo en los pacientes cubanos con diagnóstico presuntivo de la EW. La frecuencia de este polimorfismo es muy baja en la India 1,8%,11 el cual se comporta como una variante rara y es diferente a lo que se informó en diversas poblaciones estudiadas.3,4,5,6 Si se analiza el polimorfismo p.L456V con programas bioinformáticos como el SIFT y el Polyphen-2 predicen que es tolerable y benigna respectivamente.4
El polimorfismo p.L456V se halló en América,6) en Asia,3,10,11,12 en Europa13 y en África no se ha estudiado. Una posible explicación de su presencia en la población cubana es por el origen étnico, pues 72% de genes son de origen caucásico.14 En diferentes países se determina el polimorfismo p.L456V, aunque no informan las frecuencias 6,15,16,17 por lo que no se pueden comparar los resultados con esta investigación y en algunos casos, no construyen los haplotipos con el uso de los diferentes marcadores moleculares y se desaprovecha la oportunidad de ampliar el diagnóstico molecular. En Turquía el polimorfismo p.L456V no se detectó; sin embargo, en el mismo exón 3, se informó el polimorfismo p.V446L.18
Para el diagnóstico molecular de enfermedades genéticas que posean uno o más genes que contienen un número elevado de exones, es necesaria una tecnología de avanzada para lograr el éxito del mismo. Existen diversos estudios moleculares en pacientes, tanto en edad pediátrica como en adultos, con la EW.19,20 Sin embargo, siempre el médico que atiende al paciente con sospecha de la EW debe hacer un minucioso diagnóstico clínico.21
Aun cuando se determinan nuevas mutaciones en el gen atp7b,22,23,24 se realizan técnicas de microarrays25) y se utiliza la secuencia de última generación;26 es necesario, en países en vías de desarrollo que estudien enfermedades genéticas, por Biología Molecular, que se incorpore la búsqueda de los polimorfismos, para ofrecer el servicio de diagnóstico molecular por métodos indirectos.
En Cuba se trabaja en el establecimiento de la estrategia para el diagnóstico molecular de la EW. Se tiene experiencia en la identificación de polimorfismos en el exón 10,27 en el intrón 928) y en el exón 13.29,30 Con los polimorfismos que se detectaron y el p.L456V en los pacientes cubanos con diagnóstico presuntivo de la EW permitirá la construcción de los haplotipos en las familias cubanas donde haya un individuo con diagnóstico clínico de la enfermedad y permitirá la realización del diagnóstico molecular por métodos indirectos.
Esta investigación tiene limitaciones que expliquen algunas de las diferencias observadas con otros estudios. Una de ellas es el tamaño de muestra que aunque en ese estudio el número de pacientes es considerable en países como Irán12 fue pequeña y la población de referencia no es la misma, ni tan siquiera similar a estudios que se replican. Además, existen diferencias étnicas entre las poblaciones, donde se identificó asociación de estos polimorfismos, con respecto a la cubana, debido a la constitución genética.














