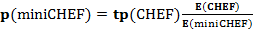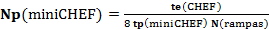INTRODUCCIÓN
Pasteurella multocida es una bacteria bacilar, Gram negativa, oxidasa positiva, no motil, no formadora de endospora, y anaerobia facultativa. Se divide en tres subespecies fundamentales: P. multocida subsp. multocida, P. multocida subsp. gallicida, y P. multocida subsp. septica basado fundamentalmente en su capacidad de fermentar azúcares como dulcitol y sorbitol (Mutters et al., 1985; Orynbayev et al., 2019). Este género bacteriano tiene un amplio rango hospedero que va desde mamíferos como conejos y ganado bobino y porcino hasta aves y reptiles (Pedersen et al., 2003; Petersen et al., 2001). Pasteurella multocida se considera un patógeno oportunista o secundario y puede encontrarse en el tracto respiratorio superior de animales tanto sanos como enfermos (Stahel et al., 2009; Kim et al., 2019). Hasta la fecha, se han reportado grandes pérdidas económicas debido a infecciones provocadas por esta bacteria en animales de cría. Es el microorganismo patógeno que con más frecuencia se aísla de conejos, y su prevalencia ha sido reportada entre el 7% y cerca del 100% en las infecciones del tracto respiratorio superior de estos animales (Stahel et al., 2009; Jaglic et al., 2006). P. multocida es igualmente causante de infecciones en humanos (Liu et al., 2003; Holst et al., 1992). Estas, en la mayoría de los casos, están directamente relacionadas con mordeduras de mascotas o inhalaciones de la bacteria, provocando neumonía o dermatitis (Holst et al., 1992; Kim et al., 2019).
Debido a la alta prevalencia y gravedad de las infecciones provocadas por esta bacteria en algunos animales, muchos han sido los trabajos encaminados a la caracterización de este microorganismo (Boyce et al., 2002; Ewers et al., 2006; Peng et al., 2019). La técnica de Electroforesis de Campo Pulsante (ECP), se considera el “estándar de oro” para el estudio molecular de P. multocida (Goering, 1993; García, 2010; Deng et al., 2015). Diversos autores han analizado brotes de pasteurelosis tanto en animales como en humanos utilizando esta técnica (Pedersen et al., 2003; Lainson et al., 2002; Kędrak et al., 2007). Aunque presenta ciertas ventajas sobre otros métodos de tipificación molecular, es una técnica que requiere tiempo, personal calificado y gran cantidad de reactivos de laboratorio. La extracción del ADN cromosomal de P. multocida toma al menos 20h de trabajo y la separación posterior de los fragmentos de ADN, resultado de la digestión con enzimas de restricción, toma de 20-40h de electroforesis (Gunawardana et al., 2000; Ivana et al., 2010; Kehrenberg et al., 2001). En total, el procedimiento de obtención de la huella digital molecular de este microorganismo requiere entre 40-60h de trabajo.
Si se utiliza el sistema de ECP miniatura Guefast06 (Neuronic, SA; Cuba; Sánchez-Enamorado et al., 2015) el tiempo de electroforesis puede reducirse a 5-7 horas. Este permite obtener resoluciones entre las moléculas de ADN que, una vez estandarizados, son similares a los que se obtienen con los sistemas convencionales (Lopez-Canovas et al., 2009, 2006, 2003). Hasta el momento no se ha reportado un protocolo estándar de ECP en minicámaras que garantice la obtención de patrones de bandas de P. multocida comprables a los reportados en la literatura, obtenidos en los sistemas convencionales de ECP.
Tampoco se han llevado a cabo estudios que involucren la selección de la enzima de restricción más efectiva para la discriminación entre los patrones de banda de cepas de P. multocida relacionadas entre sí. Este es uno de los parámetros claves en el éxito de los procedimientos de tipificación molecular mediante ECP (Goering, 2010), lo cual sería importante desde el punto de vista epidemiológico, para el análisis de brotes infecciosos provocados por esta bacteria. La obtención de patrones con isorresolución entre los sistemas de ECP convencional y el miniaturizado y la selección de la enzima de restricción que favorezca la discriminación entre patrones de bandas usualmente se realizan mediante ensayos de prueba y error. Esto implica un gasto excesivo de reactivos y enzimas que encarecen el proceso. Los programas computacionales PFGE Simulator (Plasencia et al., 2013) y GuefastScan (Santos et al., 2013), incluidos en el Sistema de ECP Guefast06 (Neuronic SA, Cuba) facilitan el diseño y análisis experimental, a la vez que garantizan el ahorro de reactivos y tiempo en el laboratorio.
Los objetivos del presente trabajo fueron obtener un protocolo estándar para la tipificación de P. multocida y determinar las enzimas de restricción que permiten la mejor discriminación entre las diferentes cepas de esta bacteria.
MATERIALES Y MÉTODOS
Selección de los parámetros de electroforesis tiempos de pulso (tp) y cantidad de pulsos (Np)
El diseño de las condiciones de tiempos de pulso (tp) y cantidad de pulsos (Np) para obtener patrones de bandas del ADN de P. multocida en el sistema Guefast06 (Neuronic SA, Cuba) se realizó in silico con las herramientas del software PFGE Simulator (Plasencia et al., 2013). El programa, mediante ecuaciones de migración de fragmentos de ADN lineal, predice la posición de cada una de las bandas en un gel virtual (López-Canovas et al., 1998; Riverón et al., 2008; Riverón et al., 2000). Las condiciones iniciales de tp y Np se calcularon a partir de las reportadas por Gunawardana et al., (2000), usando las siguientes ecuaciones:
donde tp (miniCHEF), E (miniCHEF), Np (miniCHEF) y N (rampas) son el tiempo de pulso, el campo eléctrico, la cantidad de pulsos y el total de cambios de tiempo de pulsos o rampas, respectivamente, que se aplicará en el sistema miniatura de ECP Guefast06. Mientras que tp (CHEF), E (CHEF) y te (CHEF) son el tiempo de pulso, el campo eléctrico y el tiempo de electroforesis reportados en la literatura para obtener los patrones de bandas de ADN de P. multocida con los sistemas convencionales de ECP (Gunawardana et al., 2000).
Las simulaciones se realizaron asumiendo un campo eléctrico de 10 v/cm, temperatura de 20˚C, y tampón TBE 0.5 X (Tris 44.5 mM, ácido bórico 44.5 mM, ácido etilendiaminotetraacético (EDTA) 1 mM) como buffer de corrida. Las condiciones de tp y Np seleccionadas in silico para miniCHEF se comprobaron experimentalmente bajo las mismas condiciones de campo eléctrico, temperatura y tampón de corrida utilizando el patrón de peso molecular concatámeros del fago Lambda y se ajustaron mediante ensayos de prueba y error.
SELECCIÓN IN SILICO DE LA ENZIMA DE RESTRICCIÓN QUE MEJOR DISCRIMINA LAS CEPAS DE P. MULTOCIDA
Se accedió a la base de datos del Centro Nacional para la Información Biotecnológica de EUA disponible en www.ncbi.nlm.nih.gov/gene y se descargaron las secuencias del genoma completo de ocho cepas de P. multocida. Cada secuencia fue cargada en el programa PFGE Simulator (Plasencia et al., 2013) y cortada de manera virtual, con cada una de las 134 enzimas de restricción de la base de datos del software. Se obtuvo la cantidad y tamaño de los fragmentos de restricción resultantes de la digestión virtual. Se preseleccionaron las enzimas que producían entre 10 y 40 fragmentos. Además, se simularon los patrones de bandas de ADN (pulsotipos) que se generarían con el uso estas enzimas bajo las condiciones de ECP previamente seleccionadas. Los pulsotipos virtuales resultantes se introdujeron en el programa GuefastScan (Santos et al., 2013). Se compararon todos los pulsotipos virutales generados y se determinó el coeficiente de DICE (Dice, 1945) para cada par de ellos.
CEPAS
Se tomaron siete cepas aisladas de brotes infecciosos del sistema respiratorio superior en cerdos (PM Granma, PM Thelma, PM 129, PM 248, PM I, PM II, PM III). Estas fueron previamente identificadas mediante métodos convencionales como P. multocida en el cepario de la empresa cubana LABIOFAM. Dichas cepas se sembraron en placas con agar y 5% de sangre de carnero desfibrinada y se incubaron 24h a 37˚C. Se seleccionó una colonia única, se sembró en Caldo Triptona Soya (Merck) y se incubó igualmente a 37˚C, con régimen de aireación 1/10 y una agitación de 200rpm, hasta pasar la fase exponencial del crecimiento.
PREPARACIÓN DE MUESTRAS DE ADN PARA ECP
La suspensión celular obtenida, luego del crecimiento en medio líquido de las siete sepas de P. multocida, se centrifugó 10min a 4°C, 2500rpm. Las células se lavaron dos veces con 1mL NaCl 0,15M. Posteriormente, 2x109 unidades formadoras de colonias (UFC) se mezclaron con 1 mL de 1,5% agarosa de bajo punto de fusión (Sigma, EEUU). Los bloques se formaron al añadir la mezcla en los moldes del Sistema de ECP Guefast06 (Neuronic SA, Cuba) y se transfirieron a 1 mL de tampón de lisis (0,01M Tris, 0,1M EDTA), 1% w:v Sarcosyl, 1% v:v Nonidet P-40, 4M urea, pH 9,5) por 2h a 45ºC. Los bloques se lavaron 2 veces a 45°C con agua destilada por 1 minuto y 6 veces con tampón TE (10 M Tris and 0,5mM EDTA pH=8,0) a 45°C por 15min cada uno. Se almacenaron hasta su uso en TE a 4°C.
DIGESTIÓN DE LOS MINIBLOQUES CON ENZIMAS DE RESTRICCIÓN
Cada bloque se lavó tres veces con tampón TE-0,5X por 15min y se equilibró con 100µL del tampón de restricción de cada enzima (New England Biolabs, MA, USA). Cada minibloque se incubó con 10U de cada una de las endonucleasas de restricción seleccionadas previamente in silico, a la temperatura recomendada por el fabricante durante 2h. La reacción de digestión se detuvo añadiendo 1mL de TE a cada minibloque.
SEPARACIÓN DE LOS FRAGMENTOS DE RESTRICCIÓN EN MINI-ECP
Los fragmentos de restricción de las siete cepas de P. multocida se separaron mediante ECP, usando la minicámara CHEF del Sistema Guefast06 (a 10 v/cm, con agarosa al 1,5% y bajo las condiciones electroforéticas de tp y Np seleccionadas previamente. El tampón de corrida empleado fue TBE 0,5X (44,5mM Tris, 44,5mM ácido bórico y 1mM EDTA, pH=8,3) y la temperatura se mantuvo a 20°C.
La tinción de las moléculas de ADN se realizó mediante la incubación de los minigeles en una solución de bromuro de etidio 0,5μg/mL durante 30min. Posteriormente, se destiñeron por 30min en agua y se fotografiaron al iluminarlos con luz ultravioleta.
Para determinar cuál enzima tuvo mejor poder discriminativo para la comparación de estas cepas, se escanearon las imágenes de los patrones de banda resultantes con ayuda de las herramientas del programa GuefastScan (Santos et al., 2013) y se calculó el coeficiente de DICE para cada par de patrones.
ANÁLISIS ESTADÍSTICO
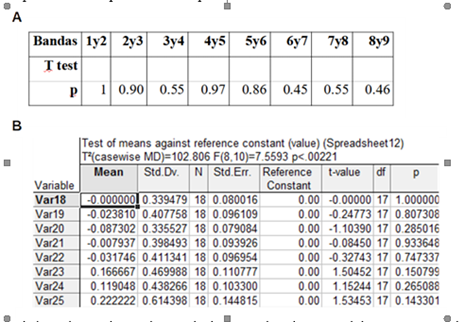
Para determinar las condiciones de tp y Np que permitieran obtener pulsotipos en miniCHEF, en comparación con los sistemas convencionales, se realizó una corrección geométrica de las imágenes experimentales. La corrección se aplicó de manera que coincidieran la mayor cantidad de bandas posibles, tomando como referencia a los patrones de peso molecular (concatámeros de fago Lambda) con respecto a los reportados en la literatura por Gunagwardana et al., 2000 y Jaglic et al., 2006. Mediante el empleo del programa GuefastScan (Santos et al., 2013) se calculó la resolución entre cada par de bandas de las 9 bandas más pequeñas del patrón de peso molecular concatámeros de fago Lambda como la diferencia entre las distancias de migración de cada par de bandas en el patrón, dividido entre el ancho de bandas (0.07cm). Se compararon los valores de resoluciones obtenidas en la cámara de miniCHEF con los valores de resolución calculados a partir de las imágenes reportadas en la literatura (Gunawardana et al., 2000; Jaglic et al., 2006), a través de las pruebas estadísticas t-student y T2 de Hotelling. Todas las pruebas estadísticas fueron realizadas con nivel de significación de 0.05 en el programa Statistica 10.0 Software Package (StatSoft, Sudáfrica).
RESULTADOS Y DISCUSIÓN
Selección de condiciones electroforéticas de tp y Np para P. multocida en miniCHEF
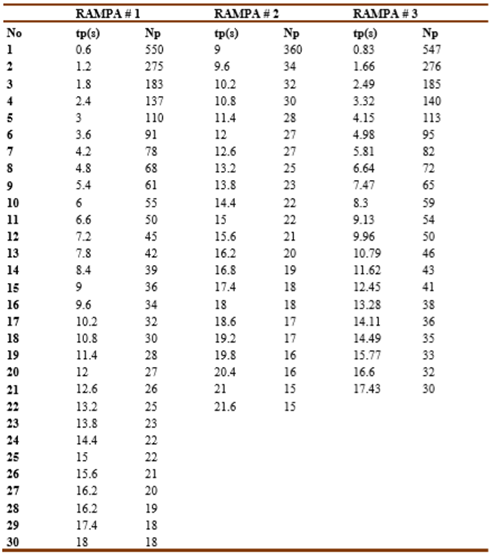
Las condiciones de tp y Np, calculadas a partir de las reportadas por Gunawardana et al., (2000), se relacionan en la Tabla 1 (RAMPA #1). Esta primera condición experimental generó un patrón virtual diferente al obtenido en el sistema de ECP convencional (resultados no mostrados). Después de ajustar de manera virtual dichas condiciones electroforéticas, mediante ensayos de prueba y error, se obtuvo la RAMPA #2 que se relaciona en la Tabla 1.
Tabla 1 Condiciones electroforéticas ensayadas en miniCHEF para obtener patrones de bandas del ADN de P. multocida comparables a los reportados en la literatura
Con el empleo de la RAMPA #2 (tabla 1) se obtuvo de manera virtual, un patrón de bandas del concatámero del fago Lambda similar al obtenido experimentalmente por Gunawardana et al., (2000) (Fig 1A y D, respectivamente). Sin embargo, el ensayo experimental de estas condiciones no brindó patrones similares a los referentes de la literatura (Fig 1B y D, respectivamente). Esta diferencia entre las resoluciones obtenidas in silico y las reales, podría explicarse por el tipo de agarosa utilizada. Las ecuaciones que describen la migración del ADN lineal en el programa PFGE Simulator están diseñadas para agarosa de alta electroendosmosis (López-Cánovas et al., 1998), mientras que la utilizada en el presente trabajo experimental fue agarosa de baja electroendosmosis. Los resultados obtenidos ponen en evidencia la influencia directa que tienen los distintos tipos de agarosa empleadas en los experimentos de electroforesis sobre la migración de las moléculas de ADN en los geles (Johnson et al., 1980).

Fig. 1 Patronesde bandas de concátameros del fago lambda obtenidos en minicámara CHEF, a 10v/cm, en agarosa 1.5%, TBE 0.5X a 20ºC mediante simulación (A), de manera experimental con la RAMPA #2 (B) y la RAMPA #3 (C), o con el sistema convencional de ECP (D).
La RAMPA #3 (tabla 1) resultó del ajuste de los parámetros experimentalmente mediante ensayos de prueba y error. Esta permitió obtener patrones de bandas del concatámero de fago Lambda similares a los reportados por otros autores en ECP convencional como Gunawardana et al., 2000, y Jaglic et al., 2006 (Fig 1C y D). Al comprar la resolución de todas las bandas en los patrones experimentales, no se encontraron diferencias significativas, ni en el análisis estadístico banda a banda usando la prueba T-student (Tabla 2 A), ni con la prueba multivariada T2 de Hotelling (Tabla 2 B).
López Cánovas et al., (2009) habían logrado previamente la isorresolución del ADN de Salmonella serotipo Braenderoup con las minicámaras CHEF y el sistema convencional de ECP. Sin embargo, estos autores necesitaron combinar los resultados de diferentes experimentos en miniCHEF para lograr patrones de bandas que fueran comparables a los que se obtienen con la ECP convencional. En contraste, en el presente trabajo se logró obtener isorresolución entre los sistemas miniatura y convencional de ECP, en una única condición experimental, tomando como referencia el patrón de peso molecular del concatámero del fago Lambda. De esta manera, se redujo el tiempo para obtener los resultados electroforéticos a solo 5h.
Selección de la enzima de restricción que mejor discrimina las cepas de P. multocida
Apa I, Bsp 120 I, Eag I, Asc I y Eco 0109 I fueron las enzimas que produjeron virtualmente entre 10 y 40 fragmentos (Fig 2). Este rango de fragmentos pudiera ser adecuado para ser resuelto en un minigel (5 x 7 x 0.5 cm; largo x ancho x alto), y suficiente para establecer comparaciones entre pulsotipos diferentes (López Cánovas et al., 2009). De estas, Apa I y Bsp 120 I son isoesquizómeros, por lo que solo se tomó en consideración Apa I para los análisis subsiguientes.
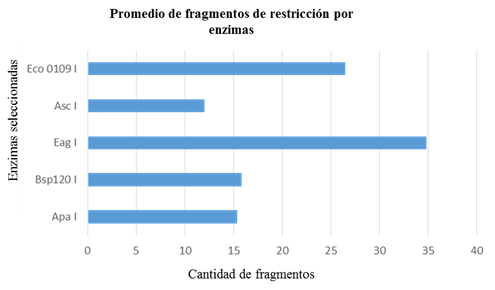
Fig. 2 Promedio cantidad de fragmentos de restricción que generan las enzimas a partir de la digestión virtual de los ocho genomas disponibles de P. multocida.
Los pulsotipos obtenidos como resultado de la digestión virtual con Eag I y simulación de la separación de los fragmentos de restricción utilizando la RAMPA #3, presentaron las menores diferencias entre las ocho cepas estudiadas (mayor coeficiente de DICE, Figura 3). Mientras que, los pulsotipos con mayores diferencias se obtuvieron usando la enzima Asc I, de manera que esta última permitió discriminar mejor las cepas utilizadas en el análisis in silico. Estos resultados contrastan con la literatura pues la enzima Apa I es la que se ha usado con mayor frecuencia para la diferenciación de cepas de P. multocida (Sanchez-Enamorado et al., 2015; Marois et al., 2009; Donnio et al., 1999, Pedersen et al., 2003).
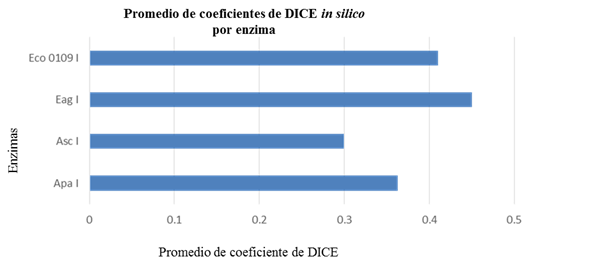
Fig. 3 Promedio de coeficientes de DICE calculado a partir de la comparación de los patrones virtuales de las ocho cepas de P. multocida
Los pulsotipos obtenidos experimentalmente, luego de la restricción del genoma de siete aislados de P. multocida con las enzimas Apa I, Asc I y Eag I y aplicando en miniCHEF las condiciones de tp y Np de la RAMPA #3, presentaron entre 8 y 16 bandas bien resueltas para las dos primeras enzimas (Fig 4). Los pulsotipos generados por Apa I tuvieron como promedio 12 bandas bien resueltas (Fig 4A), los generados por Asc I, 8 bandas (Fig 4B); sin embargo, no se obtuvieron patrones con digestión completa de 6 de las 7 cepas estudiadas con la enzima Eag I (Fig 4C). Este comportamiento ha sido reportado en la literatura para cepas que se consideran no tipificables bajo ciertas condiciones; por ejemplo, cuando el ADN bacteriano se encuentra metilado que impide la digestión eficiente de determinadas enzimas sensibles a estas modificaciones (Goering, 2010). Además, la enzima Apa I, permitió diferenciar cepas que Asc I no discriminó (Figura 4 A y B, señaladas en rectángulos rojos).
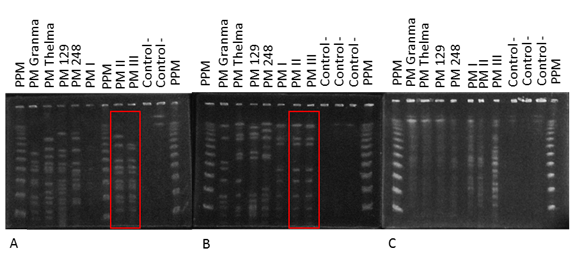
Fig. 4 Patrones de bandas del ADN de siete aislados de P. multocida obtenidos en miniCHEF con la RAMPA #3, luego de la digestión enzimática con Apa I (A), Asc I (B) o Eag I (C). El rectángulo rojo señala las cepas que se pudieron diferenciar con Apa I y no con Asc I.
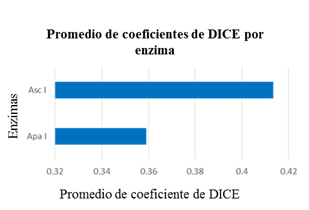
Los coeficientes de DICE, calculados al comparar los patrones de bandas obtenidos experimentalmente para las siete cepas estudiadas; evidenciaron que la enzima Apa I mostró como promedio, las mayores diferencias entre las siete cepas (menor coeficiente de DICE, Fig 5). Estos resultados contrastan con los obtenidos previamente en el análisis in silico, en el que la enzima más discriminativa resultó ser Asc I (Fig 3). La diferencia podría explicarse porque las cepas usadas en ambos análisis (experimental e in silico) fueron diferentes. Sin embargo, Apa I es la enzima que con más frecuencia se ha utilizado para la tipificación de P. multocida, según los reportes de la literatura (Sanchez-Enamorado et al., 2015; Marois et al., 2009; Donnio et al., 1999; García, 2010).
CONCLUSIONES
En este estudio se reportó por primera vez las condiciones experimentales para la tipificación de P. multocida utilizando la tecnología miniaturizada de ECP con las que se obtuvieron patrones de bandas de ADN homólogos y comparables a los reportados en la literatura. De esta manera el tiempo de electroforesis se redujo a 5h. Adicionalmente, se determinó que las enzimas Apa I y Asc I son las que mejor discriminan diferentes cepas de P. multocida. La rapidez del análisis genómico sería determinante en el contexto de brotes infecciosos producidos por esta bacteria para el tratamiento oportuno de los pacientes (animales y humanos). Al mismo tiempo, las condiciones experimentales reportadas en el presente trabajo permitirían compartir la información genética de los brotes entre diferentes laboratorios, sin importar el sistema de ECP del que dispongan, lo que permitiría establecer estrategias de vigilancia epidemiológica regionales o globales más eficientes. Sin embargo, el análisis de un mayor número de cepas se haría necesario para corroborar los resultados obtenidos en el presente trabajo.