Introducción
La familia Hyphomicrobiaceae, del orden hyphomicrobiales y la clase alphaproteobacteria, constituye un grupo fenotípicamente heterogéneo de bacterias gram negativas, que comprende especies de interés médico, biotecnológico y medioambiental.1 Esta familia incluye a los géneros Caenibius, Dichotomicrobium, Filomicrobium, Hyphomicrobium, Limoniibacter, Methyloceanibacter, Methyloligella, Pedomicrobium, Prosthecomicrobium, Seliberia y Rhodomicrobium, los cuales están validados por el Código Internacional de Nomenclatura de Procariotas (ICNP, por sus siglas del inglés International Code of Nomenclature of Prokaryotes).2
El género Rhodomicrobium está conformado por tres especies, R. udaipurense, R. vannielii y R. lacus, que incluyen cepas que habitan en ambientes extremos y son importantes para la biorremediación y el tratamiento anaeróbico de desechos. Rhodomicrobium udaipurense JA643T, por ejemplo, presenta genes que codifican enzimas involucradas en la degradación de compuestos aromáticos.3 Del mismo modo, Rhodomicrobium vannielii presenta la maquinaria enzimática requerida para la oxidación del hierro, y Rhodomicrobium lacus puede habitar en ambientes alcalinos.4,5 Adicionalmente, todas las especies de Rhodomicrobium son capaces de fijar dinitrógeno. La información genómica disponible indica que presentan genes que codifican molibdeno-hierro nitrogenasa y hierro-hierro nitrogenasa; Rhodomicrobium vannielii presenta además una nitrogenasa de vanadio-hierro.4,5,3
El desarrollo de plataformas de secuenciación de alto rendimiento o nueva generación Next-Generation Sequencing (NGS), ha permitido la secuenciación de genomas completos y un incremento acelerado en el uso de los datos ómicos. La disponibilidad de genomas completamente secuenciados para un gran número de especies bacterianas, constituye una oportunidad para la taxonomía y el diagnóstico molecular. La comparación de secuencias homólogas de proteínas de diferentes especies mediante el alineamiento múltiple, permite la identificación de marcadores moleculares CSI (del inglés Conserved Signature Indels, inserciones y deleciones) que son utilizados para estimar relaciones filogenéticas y para la demarcación de grupos específicos de organismos en términos moleculares.6
A pesar de que el género Rhodomicrobium ha sido ampliamente estudiado, pocas investigaciones han estado dirigidas a la estimación de marcadores genéticos o bioquímicos que distingan a sus miembros de otros grupos de bacterias. Teniendo en cuenta que el mismo comprende especies de gran relevancia, el presente estudio se basa en la estimación de indeles de tipo inserción en secuencias de proteínas, que pudieran ser útiles en la identificación y clasificación de especies, y para futuros estudios bioquímicos o funcionales en las enzimas de interés.
Materiales y métodos
Se analizaron las proteínas ADN ligasa NAD+ dependiente y Serina ARNt sintetasa, pertenecientes a especies del orden hyphomicrobiales. Las secuencias homólogas de las mismas fueron obtenidas a partir de la base de datos UniprotKB/Swiss-Prot (https://www.uniprot.org), y el resultado fue enriquecido mediante búsquedas en bases de datos disponibles en el sitio NCBI (National Center of Biotechnology Information, http://www.ncbi.nlm.nih.gov); empleando la herramienta BLASTp (Basic Local Alignment Search Tool) y utilizando la secuencia de Rhodomicrobium vannielii como secuencia de entrada.7,8 El resultado de la búsqueda con el programa Blastp fue analizado, con el objetivo de seleccionar secuencias altamente similares a las del género Rhodomicrobium, sobre la base de los siguientes valores: Expected value (E value) < 0,001; Identity >35 %, y Bit score > 50.9
Las secuencias escogidas fueron organizadas en conjuntos, archivadas en formato FASTA y posteriormente alineadas mediante el programa MUSCLE.10 Los alineamientos obtenidos se analizaron mediante inspección visual para identificar las inserciones, considerando relevantes aquellas flanqueadas por regiones conservadas y con igual longitud en todas las especies que la comparten, como sugieren los autores de la metodología.6
Se escogieron genomas completos de especies comprendidas en la familia Hyphomicrobiaceae a partir del Centro de Recursos Bioinformáticos de Bacterias y Virus (BV-BRC, http://www.bv-brc.org).11 Para ser incluidos, los genomas fueron evaluados teniendo en cuenta parámetros como calidad del 100 %, además de consistencias fina y gruesa mayor a 95. Los genomas completos que no fueron encontrados en el BV-BRC, fueron obtenidos de la base de datos Genome del NCBI, y anotados manualmente.
El alineamiento de los genomas se realizó igualmente con el programa MUSCLE, y posteriormente se realizó análisis filogenómico por el método de Máxima Verosimilitud a partir del programa RAxML, el cual está integrado al BV-BRC, y se utilizaron 1 000 réplicas de bootstrap.12,13 Las especies utilizadas como grupo externo fueron: Rhodobacter capsulatus DSM 1710, Rhodospirillum rubrum ATCC 11170 y Caulobacter segnis ATCC2175.
Resultados y discusión
En el presente trabajo se identificaron inserciones en las proteínas ADN ligasa NAD+ dependiente y Serina ARNt sintetasa, las cuales fueron evaluadas para estimar si estas constituyen marcadores moleculares útiles para estudios moleculares en el género Rhodomicrobium.
En cada figura, el nombre científico es seguido por el número de acceso de la secuencia en la base de datos. Los puntos muestran identidad con el aminoácido de la primera secuencia en el alineamiento, y los espacios representan brechas que indican ausencia de la inserción la cual está señalada por un cuadro. La figura 1 muestra parte del alineamiento de la proteína Serina ARNt sintetasa, en la que se puede observar una inserción exclusiva del género Rhodomicrobium, que permite diferenciar a sus miembros del resto de las bacterias analizadas. En la figura 2 se muestra una inserción de seis aminoácidos en la proteína ADN ligasa NAD+ dependiente, la cual está presente en las secuencias de Rhodomicrobium vannielii y R. udaipurense, pero está ausente en R. lacus y en el resto de las especies analizadas.
No existen reportes de marcadores moleculares de tipo inserción distintivos del género Rhodomicrobium, por lo que estos resultados pudieran ser utilizados en posteriores estudios para la identificación de características bioquímicas y fisiológicas exclusivas del mismo. Los estudios funcionales y de modelación estructural han revelado, que los indeles están presentes mayormente en los bucles superficiales de las proteínas, y se plantea que los mismos desempeñan un papel esencial en las bacterias portadoras.14,15,16
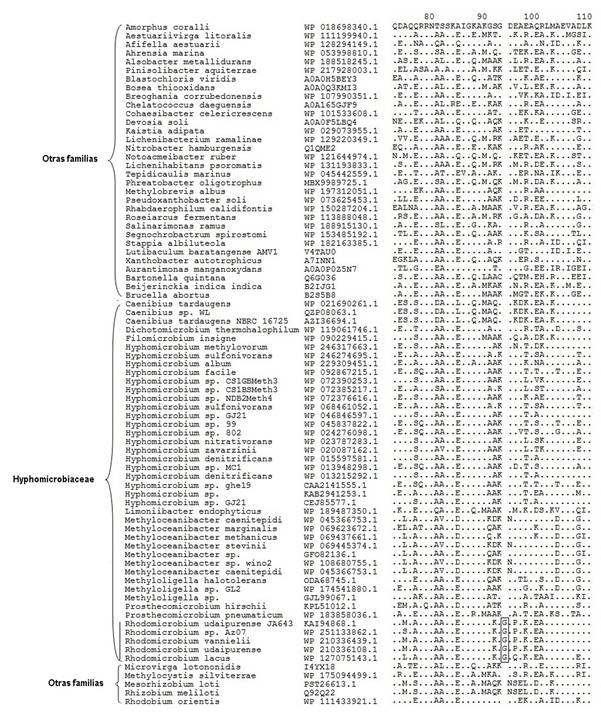
Fig. 1 Alineamiento de la proteína Serina ARNt sintetasa que muestra inserción de un aminoácido característica del género Rhodomicrobium
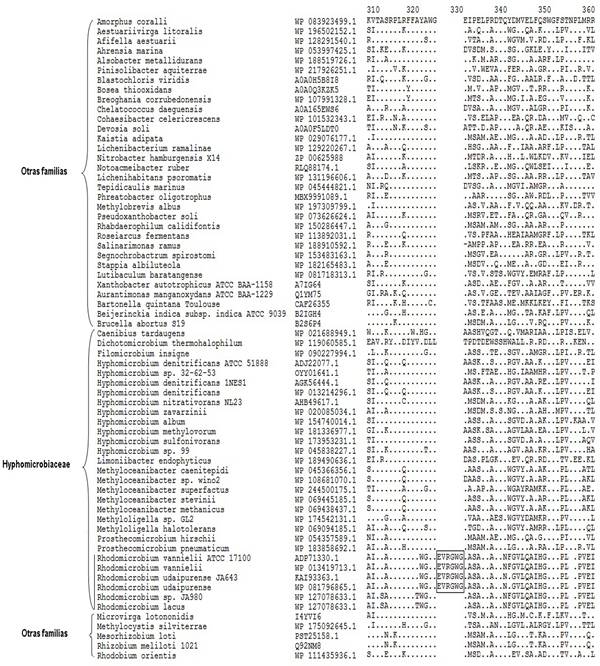
Fig. 2 Alineamiento de la proteína ADN ligasa NAD+ dependiente que muestra inserción de seis aminoácidos en Rhodomicrobium vannielii y R. udaipurense, ausente en R. lacus y el resto de las bacterias analizadas
El género Rhodomicrobium es reconocido por su capacidad de crecer en ambientes extremos, por su versatilidad fisiológica e importancia ecológica en los ciclos del carbono, nitrógeno y azufre. El mismo incluye cepas que degradan químicamente una gran variedad de compuestos orgánicos, y son importantes para la agricultura al tolerar las altas temperaturas que alcanza la materia orgánica durante el proceso de compostaje. Actualmente, la aplicación de estas bacterias se ha extendido a otras áreas como la ganadería, avicultura, porcicultura, así como para el reciclaje de residuos y el tratamiento de agua y efluentes.17
La bacteria R. udaipurense JA643T, por ejemplo, es una cepa sicrotolerante que sobrevive en altas concentraciones de metales.3 Esta cepa presenta genes que codifican enzimas monooxigenasas, dioxigenasas y peroxidasas involucrados en la degradación de compuestos aromáticos heterocíclicos. Por otro lado, R. vannielii ATCC 17100 y Rhodomicrobium sp. R_RK_3 han sido identificadas como bacterias capaces de precipitar metales disueltos como el hierro.4,18
La figura 3 muestra el árbol de Máxima Verosimilitud; los números en los nodos internos se corresponden con los valores de soporte de Bootstrap. La inserción de un aminoácido presente en la enzima Serina ARNt sintetasa es exclusiva de Rhodomicrobium, y soporta la monofilia de ese género. Este resultado es congruente con el análisis filogenómico realizado, y corrobora que el género Rhodomicrobium forma un grupo monofilético soportado por un valor de bootstrap de 100 % (figura 3), como fue reportado en estudios previos.5 De esta manera, la inserción constituye un marcador distintivo de Rhodomicrobium que puede ser utilizado como un carácter molecular complementario en estudios taxonómicos y filogenéticos.
Del mismo modo, la inserción hallada en la enzima ADN ligasa NAD+ dependiente, es congruente con el análisis filogenético. Esta inserción está presente en las especies Rhodomicrobium vannielii y R. udaipurense, pero ausente en R. lacus. En el árbol filogenético R. vannielii ATCC 17100 y R. udaipurense JA643 forman un grupo hermano, soportado por un valor de bootstrap de 100 %, y R. lacus JA980 está ubicado en una rama aparte. Este hecho sugiere que la mutación involucrada en la inserción ocurrió en el ancestro común de las dos especies que lo portan, señalando a R. lacus como una especie más antigua que R. vannielii y R. udaipurense.
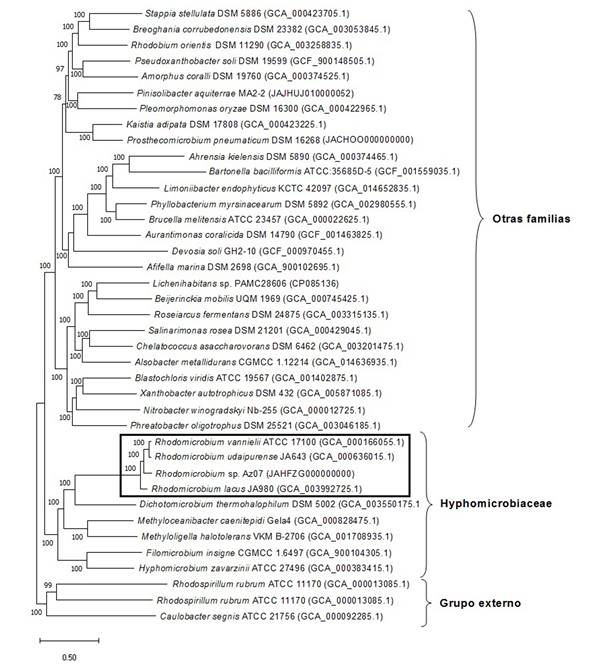
Fig. 3 Árbol filogenómico inferido por el método de Máxima Verosimilitud a partir de genomas completos de 39 especies del orden hyphomicrobiales
Los resultados pueden tener significado práctico en el diseño de protocolos de diagnóstico basados en técnicas de PCR (Reacción en Cadena de la Polimerasa), así como para la identificación in silico de especies conocidas o nuevas, mediante búsqueda por similitud con el programa BLASTp. Los marcadores indeles del tipo inserción son muy utilizados en estudios taxonómicos y evolutivos como caracteres moleculares distintivos a determinados grupos. Actualmente se utilizan como complemento a los estudios filogenómicos para la descripción de nuevas especies bacterianas.19,20
En el desarrollo de la sistemática bacteriana, resulta de gran valor la identificación de marcadores moleculares o características exclusivas de los diferentes grupos de bacterias. La molécula 16S ARNr es muy conservada, y a pesar de que es muy útil para el análisis de grupos distantes a nivel de phylum, resulta difícil su utilización para la diferenciación de especies, debido a que no presenta variaciones considerables para la distinción a este nivel 21; todo lo cual corrobora una vez más el valor de los marcadores moleculares del tipo inserción como complemento a los métodos tradicionales para la clasificación de bacterias.
Conclusiones
El análisis de las proteínas permitió la identificación de inserciones, que pueden ser utilizadas como marcadores moleculares que complementan los estudios taxonómicos y filogenéticos en Rhodomicrobium. La inserción de un aminoácido en la proteína Serina ARNt sintetasa soporta la monofilia de Rhodomicrobium, y permite una distinción a nivel de género; mientras que la inserción de seis aminoácidos en la ADN ligasa NAD+ dependiente confirma que R. lacus es un grupo hermano de R. vannielii y R. udaipurense. Los datos presentados representan un aporte teórico que sirve de base para futuros estudios bioquímicos y funcionales en las proteínas evaluadas.














