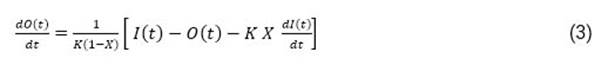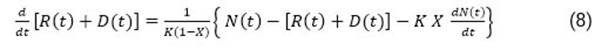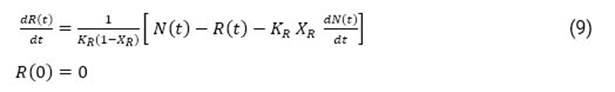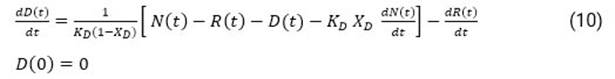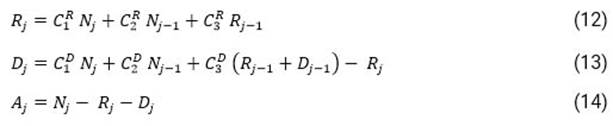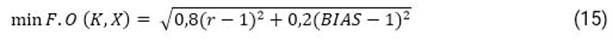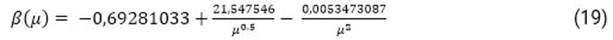Introducción
Los modelos son descripciones simplificadas de la realidad que involucran características que se consideran importantes para la comprensión del fenómeno en estudio. En particular, los modelos matemáticos son descripciones, traducidas a un lenguaje muy preciso, que permiten establecer las consecuencias de los supuestos que se hacen en ocasiones.1 En este universo de modelos, los epidemiológicos son considerados una de las herramientas más poderosas para analizar y comprender la propagación y el control de epidemias. El análisis de la dinámica de transmisión de las enfermedades infecciosas permite arribar a modelos más robustos y contribuir a enlentecer su transmisión.2,3) Desde los inicios del siglo xx, una gran cantidad de trabajos han sido publicados sobre la construcción de modelos epidemiológicos para enfermedades contagiosas, entre las que puede mencionarse la polio, la malaria, la influenza, el VIH/sida, entre otras.4-8 De acuerdo con Anderson y May9, estos modelos abarcan desde simples modelos de ajuste de curvas, modelos compartimentados MSEIR, MSEIRS, SEIJR, SIR, SIRS, SEIR, SEIS, SI, SIS etc. hasta complejos modelos estocásticos.10-17 También la disponibilidad de grandes bases de datos ha hecho posible el uso de modelos matemáticos complejos para analizar datos.18
Dentro de las enfermedades emergentes del siglo XXI se encuentra el Síndrome Respiratorio Agudo Severo (SARS, por sus siglas en inglés). De acuerdo con Zhong et al.19 y Naheed et al.2, esta es una enfermedad viral respiratoria causada por un coronavirus llamado SARS-CoV. El primer caso con SARS fue diagnosticado en noviembre 2002, en la provincia china de Guangdong. A finales de febrero de 2003, la epidemia de SARS se expandió alrededor del mundo, cuando un médico de Guangdong infectó a varias personas en un hotel en Kowlon. También se conoce que el SARS se propagó a través de vuelos aéreos. De acuerdo con estimados de la Organización Mundial de la Salud (WHO, por sus siglas en inglés), 8450 personas fueron infectadas y 810 fallecieron debido a SARS en 33 países de los 5 continentes. Aunque este brote de la epidemia fue controlado a finales de 2003, se reportaron brotes aislados de SARS en Singapur, Taiwan y China.20 Según reportan Naheed et al.2, la principal causa de esta epidemia fue la liberación accidental de SARS-CoV a partir de muestras de laboratorio. Los animales infectados por la cepa de SARS-CoV también infectaron humanos, y a inicios de 2004 se volvieron noticia nuevos casos de esta enfermedad. Ambos incidentes mostraron el peligro del brote de SARS, sobre todo en un futuro cercano, ya sea por SARS-CoV evolucionando de animales o por muestras de los virus procedentes de laboratorios. Naheed et al.2 también plantearon la posibilidad de que, bajo circunstancias especiales, este virus podía convertirse en el primer SARS-CoV humano, con la habilidad de transferirse de animales a humanos.
Solo una década más tarde, otro coronavirus patógeno, conocido como síndrome respiratorio del Medio Oriente (MERS-CoV) se extendió por los países de esa región.21 Recientemente, a finales de 2019, en Wuhan, un centro de negocios emergente de China, tuvo lugar un brote de un nuevo coronavirus que mató a más de 1800 personas e infectó a más de 70 000 en los primeros 50 días de la epidemia. El nuevo virus fue llamado por los investigadores chinos en la etapa inicial de la epidemia como coronavirus de Wuhan o 2019-nCov (2019 novel coronavirus), y luego el Comité Internacional de Taxonomía de los Virus (ICTV, por sus siglas en inglés) lo nombró como SARS-CoV-2, y a la enfermedad como COVID-19.22 Como ya se planteó con anterioridad, el SARS-CoV se diseminó en los 5 continentes, con una tasa de mortalidad del 9 %.
El SARS-CoV-2, hasta la fecha que se escribe esta contribución, ha afectado a 185 naciones del mundo, infectando a más de 4,8 millones de personas, con una tasa de mortalidad de 6,7 % y una tasa de transmisión superior a la del SARS-CoV. En una reciente investigación Shereen et al.23 estudiaron las características biológicas e infecciosas de SARS y MERS, con especial atención a la COVID-19. La aparición de este nuevo coronavirus, como en ocasiones anteriores, ha motivado a la comunidad científica a trabajar, no solamente en el área clínica asistencial, sino en otras ramas, entre las que se encuentra la modelación epidemiológica.3,24,25
En esta contribución se propone un modelo inspirado en procesos hidrológicos para el pronóstico de escenarios de infección de la COVID-19 que relaciona la transición dinámica de personas contagiadas por la COVID-19 hacia recuperadas y fallecidas, y es de destacar que esta aplicación no ha encontrado precedentes en la literatura consultada. Para desarrollar el modelo se empleó el principio de analogía con procesos hidrológicos, en particular el flujo en ríos. Este modelo puede ser considerado de una estructura simple ya que presenta pocos parámetros y requiere la solución de dos ecuaciones diferenciales ordinarias. Además, basa su implementación en la utilización de asistentes matemáticos conocidos. Por último, en este artículo se realiza la demostración de las bondades del modelo con su aplicación al pronóstico de la evolución de la pandemia de la COVID-19 en Cuba.
Métodos
Modelo epidemiológico inspirado en un proceso hidrológico
Ecuaciones
El modelo que se propone en esta contribución está inspirado, por analogía, en un proceso natural, como es el tránsito hidrológico en ríos o embalses, los cuales actúan como sistema ante el ingreso y salida de determinados volúmenes de agua en cierto periodo de tiempo. Todo fenómeno hidrológico puede ser sintetizado matemáticamente como un modelo de entradas y salidas (figura 1A). Sin entrar en detalles de su formulación, el modelo que se presenta está basado en el método de Muskingum, propuesto por McCarthy en 1938 y que puede ser consultado con profundidad en Chow26, HEC27 y Szymkiewicz (28.
En el método originalmente propuesto por McCarthy, las diferencias entre los flujos de agua de entrada y de salida, ordenadas de las funciones de entrada I(t) y de salida O(t), dependientes del tiempo, son iguales a las tasas de almacenamiento en el río, representadas en la figura 1B por el área sombreada. Como puede observarse en dicha figura, existen procesos de retardo y atenuación a la vez. Esto se explica por la existencia de almacenamiento en el tramo en estudio. Por ejemplo, si el punto A representa determinado volumen, el mismo demorará cierto tiempo en salir (punto C). De forma análoga, para un mismo instante de tiempo se registra a la entrada y a la salida volúmenes diferentes (puntos B y C).
En sentido general, todo proceso está sometido a cambios continuos, particularmente en el tiempo. Aplicando el principio de analogía, considerando flujos, pero asociados a cantidades de personas contagiadas por determinada enfermedad, por ejemplo la COVID-19, cuyo ciclo de incubación es de 14 días, el proceso puede esquematizarse tal y como se muestra en figura 1C.
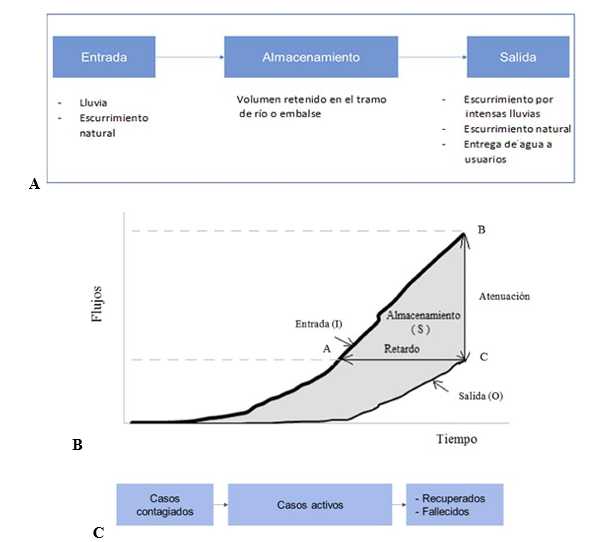
Fig. 1 . A) Sistema que caracteriza procesos hidrológicos. B) Relación entre flujos de entrada, salida y almacenamiento. C) Sistema que puede caracterizar el contagio de una enfermedad.
A partir de lo ilustrado en las figuras 1A-1C dicho proceso se puede representar matemáticamente por:
Por otro lado, de acuerdo con McCarthy, el almacenamiento puede expresarse a través de la siguiente función:
siendo X un factor de ponderación adimensional que determina el peso relativo de los flujos de entrada y de salida, cuyo valor puede estar dentro del intervalo de 0 a 1. Si el almacenamiento solamente es función del flujo de la variable de salida O t , el factor de peso X = 0. Por otro lado, K es el tiempo de retención, el cual es un indicador del proceso de retardo. Al sustituir la derivada de la ecuación (2) en (1) y despejar la variación de la función de salida en el tiempo se obtiene:
sujeta a la condición inicial O (0) = 0. La ecuación (3) se integra por un método numérico en diferencias finitas de tipo explícito, descrito en detalle por Szymkiewicz28. De esta manera, dicha ecuación conduce al siguiente sistema de ecuaciones algebraicas:
siendo j el índice de nivel de tiempo (j = 1, 2, M). Las constantes C 1 , C 2 y C 3 vienen dadas por las expresiones siguientes:
donde ∆𝑡 es el paso de tiempo.
Aplicando el principio de analogía para la epidemia COVID-19, se considera en esta propuesta que existe un flujo de casos confirmados 𝑁 𝑡 , que en la figura 1 se corresponde con el flujo de entrada, y un flujo de salida, en este caso correspondiente a casos recuperados 𝑅 𝑡 y fallecidos 𝐷(𝑡). Por tanto, se puede plantear que 𝑁 𝑡 =𝐴 𝑡 +𝑅 𝑡 +𝐷 𝑡 , siendo A, R y D los casos activos, recuperados y fallecidos, respectivamente, acumulados en el instante de tiempo t.
Teniendo en cuenta lo anterior, de la ecuación (3) se puede obtener por sustitución:
Sin embargo, conocidos los contagios 𝑁 𝑡 , la ecuación (8) presenta dos incógnitas, en este caso, los flujos de casos recuperados y fallecidos. Para darle solución a este inconveniente, inicialmente el flujo de casos recuperados se obtiene considerando que no hay fallecidos, y para esto la ecuación (8) se transforma en:
A partir de esta consideración, con posterioridad el flujo de casos fallecidos podrá determinarse según:
siendo (X R , K R ) y (X D , K D ) parámetros de ajuste para los flujos de casos recuperados y fallecidos respectivamente. Finalmente, una vez resueltas las ecuaciones (9) y (10), podrá cuantificarse el flujo de casos activos 𝐴 𝑡 acumulados en el instante de tiempo t según:
Aplicando el esquema en diferencias finitas de la ecuación (4) a las ecuaciones (9) y (10) se obtendrá:
Nótese que las constantes C 1 , C 2 y C 3 se distinguen por los supraíndices R y D ya que las ecuaciones (12) y (13) dependen de los parámetros (X R , K R ) y (X D , K D ), respectivamente.
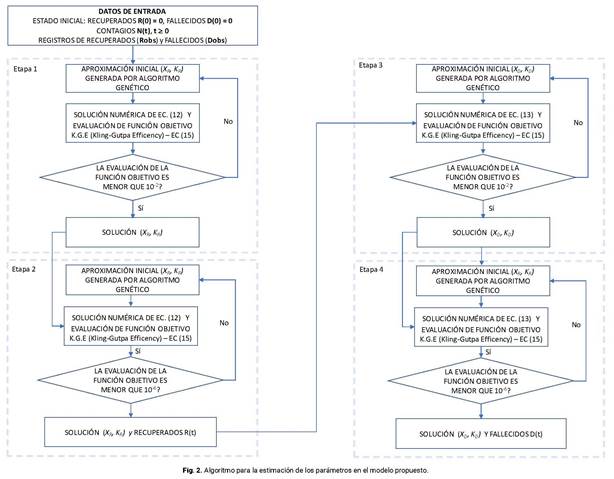
En esta contribución, el cálculo de los parámetros de cada una de las dos ecuaciones se realiza a partir de la solución de un problema inverso, de forma secuencial, o sea, primero se determinan los parámetros de la ecuación (12) y luego los de la ecuación (13). Para realizar este cálculo se estableció un algoritmo iterativo el cual fue programado en el asistente matemático MatLab. De esta forma, teniendo en cuenta que los valores finales de los parámetros (X R , K R ) y (X D , K D ) se determinan por ajuste, se estableció como criterio para finalizar el cálculo iterativo la minimización de la función objetivo de KGE (Kling-Gutpa Efficency), propuesta por Gupta et al :29
siendo r el coeficiente de correlación entre simulaciones y registros; y BIAS, la relación entre las medias de las simulaciones y los registros. Estos últimos son de los recuperados o fallecidos, según sea la ecuación que se esté ajustando.
Para estimar los parámetros antes mencionados minimizando la función objetivo dada por la ecuación (15), las ecuaciones (12) y (13) deben ser acopladas a un algoritmo para la estimación óptima de parámetros. En esta contribución se empleó un algoritmo genético implementado en el asistente matemático MatLab, a través de la función GA. Detalles de este algoritmo y sus aplicaciones, pueden consultarse en Goldberg30 y Conn et al.31),(32
Para la aplicación del algoritmo genético, se realizó la estimación de parámetros en dos etapas. La primera de ellas se caracterizó por realizar una estimación débil, tomando en cuenta que la tolerancia en la evaluación de la función objetivo fue alta (Tol = 10-2). De esta manera se generó la aproximación inicial de la segunda etapa donde se vuelve a aplicar el algoritmo genético, para realizar una estimación más robusta, considerando una tolerancia Tol = 10-6. En la figura 2 se presenta el algoritmo del procedimiento aplicado.
Modelo de transición para la simulación de los acumulados de casos confirmados
La serie de registros acumulados de casos confirmados 𝑁 𝑡 de la COVID-19, hasta una fecha determinada, puede ser ajustada por un modelo de transición, considerando que la reducción de los contagios por día conduce a la formación de un máximo de casos confirmados acumulados. Por tanto, a través de la utilización del ajuste anterior, la serie de casos confirmados puede ser extendida en el tiempo hasta alcanzar el máximo y de esta forma pronosticar un máximo posible de casos confirmados.
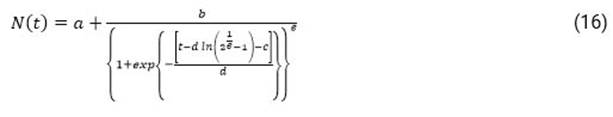
Para determinar el modelo de transición que mejor ajusta los datos se propone el programa TableCurve-2D, desarrollado por Systat_Software_Inc.33, el cual brinda a los investigadores la posibilidad de encontrar el modelo ideal para datos de estructura compleja. Este sistema tiene una biblioteca incorporada que incluye un amplio espectro de modelos lineales y no lineales. Dentro de estos modelos se encuentran varias ecuaciones de transición, como por ejemplo, el modelo pentaparamétrico Asymmetric Sigmoid (Sigmoide asimétrico, SA) cuya estructura es:
siendo a, b, c, d y e, los parámetros de ajuste. El modelo de transición presentado puede caracterizar la evolución de la epidemia de forma asimétrica en el tiempo, caracterizada por una rama de rápido ascenso y otra de lento descenso.
No es menos cierto que un modelo diferente podría haberse empleado. Por ejemplo, Ramón-Hernández et al.25 emplearon técnicas de inteligencia artificial empleando diferentes funciones de transición a partir de distribuciones de casos activos de tipo trapecio y campana. Sin embargo, en este sentido no existe contradicción al respecto, ya que son alternativas para la generación de contagios, como ya se explicó, para extender la serie temporal de casos confirmados y estimar el número máximo de contagiados, desconocido a priori.
Modelo estocástico para la generación de escenarios de casos confirmados de la COVID-19
Para la generación de escenarios fue utilizado un modelo estacionalmente integrado, cuya media móvil estacional se hizo corresponder con un ciclo de incubación de la enfermedad (14 días). Este tipo de modelo, conocido sintácticamente como ARIMA (p,D,q), donde p, D y q son parámetros del mismo, puede verse en detalle en Box et al., 34 y se encuentra implementado en el asistente matemático MatLab.
Los modelos estacionalmente integrados facilitan al modelador la generación de diversos escenarios, pero está reconocido que usualmente involucran cierta dificultad. Por esta razón, con el objetivo de realizar un análisis de sensibilidad y escoger aquellos valores que garantizaran escenarios convergentes, de los parámetros involucrados (p, D y q) se varió solamente uno de ellos, el parámetro q, que representa el grado polinomial de la media móvil, cuyo dominio de la variable es 𝑞= 1,2,…, 𝑞 ∗ , siendo 𝑞 ∗ el grado máximo del polinomio en correspondencia con el número de observaciones o registros que caracterizan a la serie temporal de casos confirmados. El criterio para su determinación puede encontrarse en Box et al.34
De esta forma, para determinar el valor de q se calibraron los parámetros del modelo utilizando del total de datos de casos confirmados hasta la fecha de la realización del pronóstico, una cantidad igual al total de los datos menos los datos correspondientes a los últimos 14 días (un ciclo de incubación de la enfermedad). Una vez concluida esta primera etapa, se estableció el pronóstico de casos confirmados de los últimos 14 días, realizando m simulaciones de Montecarlo del modelo ARIMA para cada uno de los valores de q, lo cual generó m patrones de valores esperados. Hay que señalar que, de ser necesario, es recomendable introducir un cambio de variables durante el proceso de ajuste del modelo ARIMA para garantizar que los residuos se ajusten a una distribución gaussiana. En caso de implementar un cambio de variables, una vez realizado el pronóstico, los valores simulados se recalculan utilizando la inversa de la ecuación utilizada. Como tercer paso se chequeó la convergencia entre los valores de los datos de los casos confirmados durante los últimos 14 días y las simulaciones, para cada valor de q, con vistas a descartar aquellos valores que no garantizan dicha convergencia. Luego, de los valores de q aceptados se tomó el valor con la varianza mínima. De esta forma, de las m simulaciones realizadas con el valor de q seleccionado, se tomaron los patrones de valores esperados correspondientes a las ramas mínima (N - ), media (N o ) y máxima (N + ), con vistas a pronosticar tres escenarios, uno favorable, otro poco favorable y uno crítico. A continuación se realizó el ensamblaje de los registros de contagios utilizados y los escenarios favorable, poco favorable y crítico (mínimo -N - , medio N o y máximo N + ), generados con el modelo ARIMA en el periodo de incubación (14 días). Por último, las tres series de registros acumulados se reajustaron al modelo de transición seleccionado (SA en esta contribución) para obtener finalmente los tres escenarios de casos confirmados, cuya serie temporal se extendió hasta alcanzar un valor aproximadamente constante. Esto último permitió estimar el valor máximo de casos confirmados en cada uno de los escenarios.
Estimación de escenarios de casos activos de la COVID-19 en Cuba
Relación número de pacientes recuperados/número de pacientes fallecidos
Un aspecto de interés durante el desarrollo de la epidemia de COVID-19 es el indicador (β), que representa la relación entre el número de pacientes recuperados R y fallecidos D. Para calcular esta variable se planteó la siguiente relación adimensional:
Este parámetro es dinámico, de manera que su valor puede variar en el tiempo. Aunque no es el único indicador, tal y como se verá más adelante, puede constituir un criterio para evaluar la capacidad de detección y la velocidad de respuesta de un sistema de salud ante la propagación de una epidemia.
Implementación del modelo propuesto para la estimación de posibles escenarios de contagio de la COVID-19 en Cuba a partir de la evolución de otros países del mundo
Con vistas a aprovechar la ventaja relativa que tiene el hecho de que la propagación de la epidemia de coronavirus se haya producido desde el Lejano Oriente hacia América, se utilizaron los datos sobre la epidemia de países que estaban en una etapa más avanzada de enfrentamiento a la misma con respecto a Cuba. Para ello se recopilaron los datos publicados en https://pomber.github.io/covid19/timeseries.json de los casos confirmados, pacientes recuperados y fallecidos, correspondientes al desarrollo de la epidemia por la COVID-19, hasta el 29 de abril de 2020, de un grupo de países incluidos entre los de mayor desarrollo económico social, con el objetivo de minimizar el sesgo de los datos sobre la epidemia. Este grupo se conformó con los 20 países de mayor índice de desarrollo humano, según datos del Informe sobre Desarrollo Humano del PNUD35y los 20 países con mayor Producto Interno Bruto36. De ese grupo de países fueron excluidos Liechtenstein y San Marino, por tener una población relativamente pequeña; y se incluyó a China, por ser el país de origen de la epidemia y tener un buen desempeño en el control de esta; y a 3 países desarrollados afectados tempranamente por la epidemia, uno asiático y dos europeos (Corea del Sur, España e Italia). Para todos estos países se calcularon el indicador (β), propuesto anteriormente, y el valor de la letalidad por la siguiente ecuación:
Seguidamente se establecieron cuatro cuartiles a partir de los valores de β de los países estudiados hasta el 16 de abril de 2020, utilizando la función cuartil de la hoja de cálculo de Excel. Además, para comprobar las bondades de los modelos propuestos se estimaron los parámetros de las ecuaciones (12) y (13) para cada uno de los países seleccionados. Teniendo en cuenta que el proceso de estimación de los parámetros se encuentra sujeto a las restricciones de estos, se concibió un rango suficientemente amplio para K R y K D (entre 10 y 5000). Para el parámetro X se consideró el caso particular de X = 0 en el cálculo de los parámetros del modelo, utilizando los datos hasta el 16 de abril de 2020, y de esta manera la variabilidad de los casos activos en el modelo viene dada por las variables de salida, en este caso, los acumulados de recuperados R y fallecidos D, en el transcurso de la epidemia. Posteriormente se calcularon los casos activos A(t) obtenidos, utilizando el modelo propuesto (ecuaciones 12, 13 y 14), con los parámetros calculados para cada país, y los datos reportados para los casos confirmados.
A continuación, se realizó el pronóstico de tres escenarios de cantidad de posibles casos confirmados con la COVID-19, utilizando los datos de Cuba hasta el 29 de abril de 2020. Para ello se utilizó primeramente el modelo ARIMA, implementado en el asistente matemático Matlab, donde se calibraron los parámetros del modelo y se determinó el dominio de q con los datos hasta el 16-abr-2020 (fecha correspondiente a 14 días antes del final de la serie de datos disponible). Para establecer el pronóstico para un ciclo de incubación, a partir del 16-abr-2020, se realizaron 100 simulaciones de Montecarlo del modelo ARIMA que generaron los patrones de valores esperados. Luego se seleccionó el modelo ARIMA que presentó la varianza mínima, y de él las ramas de valores esperados mínima (N - ), media (N o ) y máxima (N + ), los cuales constituyen en última instancia el pronóstico de escenarios favorable (mínimo-N - ), poco favorable (medio-N o ) y crítico (máximo-N + ). Posteriormente se realizó el ensamblaje de los registros de contagios hasta el 16-abr-2020 con los pronósticos de los escenarios favorable, poco favorable y crítico (mínimo-N - , medio-N o y máximo-N + ), generados con el modelo ARIMA en el periodo de incubación de 14 días. Estas tres series generadas después del ensamblaje fueron ajustadas con el modelo de transición SA, seleccionado por medio del programa TableCurve-2D.
Teniendo en cuenta que en el momento que se realiza este estudio se cuenta con los datos del desarrollo de la epidemia en otros países que llevan mucho más tiempo que Cuba enfrentando la pandemia, se incluyó este factor en el pronóstico de posibles escenarios de casos activos para Cuba, a través de la utilización de los datos de los países incluidos en el mismo cuartil que Cuba. En este sentido, para evaluar posibles escenarios del comportamiento de los casos activos A, se consideraron los valores extremos de ( de ese cuartil y uno intermedio cercano al valor de Cuba.
De esta forma, para realizar la estimación de los posibles escenarios para Cuba de casos activos de la COVID-19, se realizó la modelación del pronóstico de casos confirmados de tres escenarios (favorable, poco favorable y crítico), extendido a 120 días con el modelo transición, utilizando el modelo epidemiológico propuesto (ecuaciones 12, 13 y 14), para los tres países seleccionados y se comparó con los datos de casos activos hasta el 29 de abril de 2020 presentados por Cuba.
Por último, en este trabajo se realizó una recalibración del modelo propuesto para Cuba con los datos hasta el día 29 de abril de 2020, donde además se incluyó en el proceso de ajuste los parámetros de peso X R y X D , que con anterioridad habían sido fijados a cero para los datos hasta el 16 de abril de 2020, con vistas a evaluar la influencia de la evolución temporal del parámetro ( en la estimación de los escenarios de casos activos y comparar estos escenarios con el comportamiento real de casos activos para Cuba hasta el 22 de mayo de 2020.
Resultados y discusión
Datos sobre el desarrollo de la epidemia COVID-19 en el mundo
En la tabla 1 se muestran los países seleccionados para este estudio, ordenados en cuartiles a partir del indicador 𝛽, con sus correspondientes datos hasta el día 16 de abril de casos confirmados, recuperados, fallecidos, el valor de la letalidad y los T días transcurridos después de detectado el primer caso en cada país hasta el 16 de abril de 2020.
Vale la pena recordar que el indicador 𝛽 es dinámico, por tanto, su valor puede variar en el tiempo. Este indicador y el de la letalidad podrían ser usados para evaluar la eficacia y eficiencia de un sistema de salud ante la propagación de la epidemia. Por ejemplo, un valor bajo de 𝛽 y un valor alto de 𝜇, después de transcurridos 28 días (dos ciclos de incubación de la enfermedad), desde que fue detectado el primer caso en un país, podría sugerir una reacción tardía de las autoridades en el enfrentamiento de la epidemia y una posible ineficacia del sistema de salud para atender a los pacientes.
Aunque lo ideal sería que no existieran fallecidos, en ocasiones resulta inevitable por causas multifactoriales. Si 𝛽→0, quiere decir que el número de fallecidos es mucho mayor que el número de recuperados (𝐷≫𝑅). Lógicamente, eso no es deseable y un sistema de salud debe concentrar todos sus esfuerzos por revertir ese escenario a uno más optimista. Una situación particular es aquella en que 𝛽=1, lo cual indica que el número de recuperados es igual al número de fallecidos (𝑅=𝐷), lo cual tampoco es un escenario deseable. Por tanto, el éxito de un buen enfrentamiento a la epidemia y su propagación es que el número de recuperados sea mucho mayor que el número de fallecidos (𝑅≫𝐷) y que el valor de la letalidad sea el menor posible.
De la tabla 1 se puede inferir que, como regla general, bajas tasas de letalidad ( se corresponden con elevados valores del indicador ( y viceversa. Para los países estudiados la relación funcional puede ser establecida a partir del siguiente modelo de ajuste, con un coeficiente de correlación r = 0,84:
Tabla 1 Datos por países hasta el 16 de abril de 2020
| Cuartil | País | ( | T(días)* | ||||
|---|---|---|---|---|---|---|---|
| 1 | Reino Unido | 104 145 | 375 | 13 759 | 13,211 | 0,027 | 77 |
| Holanda | 29 383 | 311 | 3327 | 7,572 | 0,093 | 50 | |
| Irlanda | 13 271 | 77 | 486 | 1,106 | 0,158 | 48 | |
| Noruega | 6896 | 32 | 152 | 0,346 | 0,211 | 51 | |
| Suecia | 12 540 | 550 | 1333 | 3,034 | 0,413 | 77 | |
| Bélgica | 34 809 | 7562 | 4857 | 13,953 | 1,557 | 73 | |
| USA | 667 801 | 54 703 | 32 917 | 4,929 | 1,662 | 87 | |
| 2 | Italia | 168 941 | 40 164 | 22 170 | 13,123 | 1,812 | 77 |
| Francia | 147 091 | 33 327 | 17 941 | 12,197 | 1,858 | 84 | |
| España | 184 948 | 74 797 | 19 315 | 10,443 | 3,872 | 76 | |
| Japón | 8626 | 901 | 178 | 2,064 | 5,062 | 92 | |
| Cuba | 862 | 171 | 27 | 3,132 | 6,333 | 36 | |
| Canadá | 30 808 | 9698 | 1257 | 4,080 | 7,715 | 82 | |
| Luxemburgo | 3444 | 552 | 69 | 2,003 | 8,000 | 48 | |
| 3 | Dinamarca | 7074 | 3203 | 321 | 0,731 | 9,978 | 50 |
| Suiza | 26 732 | 15 900 | 1281 | 4,792 | 12,412 | 52 | |
| Alemania | 137 698 | 77 000 | 4052 | 2,943 | 19,003 | 81 | |
| Austria | 14 476 | 8986 | 410 | 2,832 | 21,917 | 52 | |
| Finlandia | 3369 | 1700 | 75 | 0,171 | 22,667 | 79 | |
| 4 | China | 83 403 | 78 401 | 3346 | 4,012 | 23,431 | 131 |
| Emiratos Árabes Unidos | 5825 | 1095 | 35 | 0,601 | 31,286 | 79 | |
| Corea del Sur | 10 613 | 7757 | 229 | 2,158 | 33,873 | 88 | |
| Australia | 6462 | 2355 | 63 | 0,975 | 37,381 | 82 | |
| Catar | 4103 | 415 | 7 | 0,171 | 59,286 | 48 | |
| Singapur | 4427 | 683 | 10 | 0,226 | 68,300 | 85 | |
| Nueva Zelanda | 1401 | 770 | 9 | 0,642 | 85,556 | 49 | |
| Islandia | 1739 | 1144 | 8 | 0,018 | 143,000 | 49 |
* Datos disponibles a partir del día 22 de enero de 2020 (la corrección de los datos de más de 86 días se realizó a partir de lo reportado por Wu et al.37
Aplicación del modelo epidemiológico propuesto para el caso de Cuba teniendo en cuenta el desarrollo de la pandemia en el mundo
Estimación de los parámetros de las ecuaciones de variación de los casos recuperados y fallecidos en el tiempo
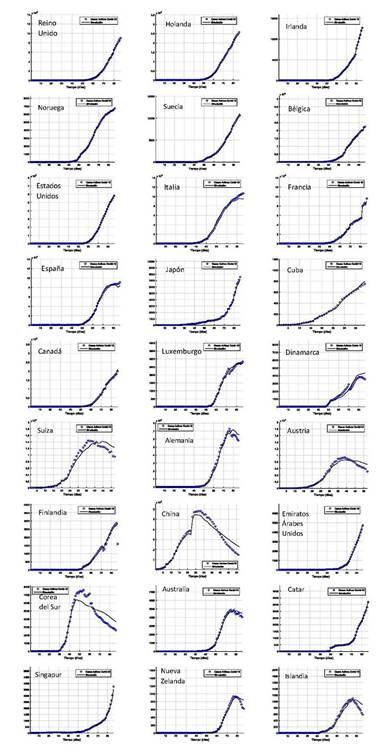
Los resultados de la estimación de los parámetros de las ecuaciones (12) y (13), obtenidos para cada uno de los países se muestran en la tabla 2. Nótese los valores alcanzados por la función objetivo en la estimación óptima de K R y K D . En la figura 3 se pueden observar las curvas de casos activos A(t) obtenidas por medio de la ecuación (14), utilizando el modelo propuesto con los parámetros calculados para cada país (tabla 2) y los datos reportados para los casos confirmados. En esta figura se evidencian las excelentes correlaciones alcanzadas utilizando el modelo propuesto.
Tabla 2 Resultados del proceso de estimación de los parámetros del modelo propuesto
| Cuartil | País | 𝑭.𝑶 ( 𝑲 𝑹 ) Ec. (15)K
| 𝑭.𝑶 ( 𝑲 𝑫 ) Ec. (15) K
|
||
|---|---|---|---|---|---|
| 1 | Reino Unido | 0,103046 | 1,30E+03 | 1,04E-02 | 38,9927 |
| Holanda | 0,084679 | 859,8368 | 0,00567181 | 89,1652 | |
| Irlanda | 0,0667629 | 2,40E+03 | 1,90E-03 | 241,14 | |
| Noruega | 0,0398507 | 3,38E+03 | 7,58E-03 | 806,0732 | |
| Suecia | 0,0219634 | 419,774 | 0,00973823 | 108,9731 | |
| Bélgica | 0,00364253 | 43,898 | 0,00696408 | 26,5661 | |
| USA | 0,00248315 | 142,7707 | 0,00040104 | 86,2059 | |
| 2 | Italia | 0,00746797 | 47,3454 | 0,00364024 | 29,4759 |
| Francia | 0,0066408 | 29,2167 | 0,00514222 | 19,2965 | |
| España | 0,0017138 | 23,9648 | 0,001641 | 17,2849 | |
| Japón | 0,024008 | 70,0512 | 0,0110803 | 59,4023 | |
| Cuba | 0,0121301 | 35,4738 | 0,00373333 | 30,584 | |
| Canadá | 0,00208606 | 30,2271 | 0,011497 | 26,7095 | |
| Luxemburgo | 0,0471844 | 87,7838 | 0,00272821 | 76,9165 | |
| 3 | Dinamarca | 0,0270506 | 35,8258 | 0,00679627 | 30,4315 |
| Suiza | 0,0108043 | 24,0608 | 0,00895449 | 21,5647 | |
| Alemania | 0,00379918 | 20,5445 | 0,0180689 | 19,3591 | |
| Austria | 0,0151884 | 26,9772 | 0,0125662 | 25,373 | |
| Finlandia | 0,143983 | 95,4236 | 0,00638518 | 83,1164 | |
| 4 | China | 0,0105394 | 28,2859 | 0,00621201 | 25,35 |
| Emiratos Árabes Unidos | 0,014732 | 45,7203 | 0,00727325 | 43,7104 | |
| Corea del Sur | 0,0213491 | 36,2999 | 0,0213491 | 34,7219 | |
| Australia | 0,0140114 | 54,6539 | 0,00407557 | 52,2541 | |
| Catar | 0,00737075 | 114,7778 | 0,0345566 | 111,9783 | |
| Singapur | 0,00635873 | 36,5679 | 0,00635873 | 35,9953 | |
| Nueva Zelanda | 0,0196002 | 28,6184 | 0,123256 | 28,2954 | |
| Islandia | 0,0189579 | 29,6632 | 0,0189579 | 29,2354 |
Generación de escenarios de posibles casos confirmados de la COVID-19 para Cuba
Durante la generación de tres escenarios de posibles casos confirmados acumulados con la COVID-19, utilizando el modelo ARIMA con los datos hasta el 16 de abril de 2020, los valores del grado máximo del polinomio q que garantizaron la convergencia de la serie temporal estuvieron en el intervalo de 1 a 6. Anteriormente a la realización de las simulaciones se llevó a cabo un cambio de variables, como había sido recomendado anteriormente, para garantizar que los residuos, durante el proceso de ajuste del modelo ARIMA, tuvieran una distribución gaussiana. La ecuación utilizada para realizar el cambio de variables en este caso fue la siguiente:
siendo 𝑁 el promedio diario de los casos confirmados en Cuba hasta el 29 de abril de 2020. Los valores simulados, una vez realizado el pronóstico, fueron recalculados empleando la relación inversa
Teniendo en cuenta las consideraciones anteriormente expuestas, y posterior al procesamiento de los datos en el asistente matemático MatLab, el modelo resultó estructurado como ARIMA (0, 1, q), arrojando los resultados que se muestran en la figura 4.
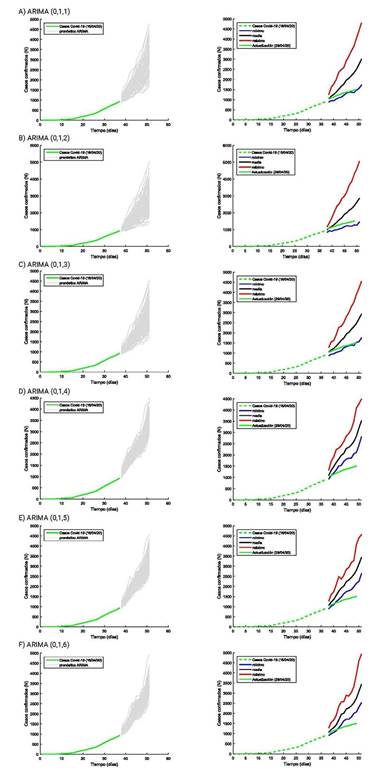
Fig. 4 Escenarios de contagio correspondiente a un ciclo de incubación de COVID-19 con modelo ARIMA (0, 1, q).
A partir de estos resultados se descartaron los valores de q en el intervalo de 4 a 6, por no garantizar la convergencia entre los valores registrados hasta el 29 de abril de 2020 y las simulaciones. De los modelos ARIMA con valores de q en el intervalo de 1 a 3, se seleccionó el que presentó la varianza mínima, en este caso el modelo ARIMA (0,1,2). En la figura 4b, a la derecha, se muestran las ramas correspondientes a los escenarios favorable (mínimo - N - ), poco favorable (medio - N o ) y crítico (máximo N + ).
La serie de registros acumulados de contagios de la COVID-19 hasta el 16 de abril de 2020, extendida inicialmente a un ciclo de incubación (14 días), se ajustó con el modelo de transición SA (ecuación 9), considerando que la reducción de los contagios por día, conduce al máximo de casos confirmados acumulados.
El modelo de transición seleccionado por medio del programa TableCurve-2D, para el ajuste de las series ensambladas con las extensiones obtenidas por el modelo ARIMA (0,1,2), muestra excelentes bondades de ajuste, tal y como se muestra en la tabla 3 y la figura 5. Nótese en esta última figura que se ha realizado una extrapolación a 120 días para estimar el número máximo de contagiados.
Tabla 3 Parámetros de ajuste de la curva de contagio de tipo SA para cada escenario.
| Curva de contagios | Parámetros | Valor | Error estándar | Intervalos de confianza 95 % | ||||||
|---|---|---|---|---|---|---|---|---|---|---|
| N_ | a | 2,6995 | 9,9102 | -17,2488 | 22,6478 | |||||
| b | 1476,8264 | 59,2728 | 1357,5165 | 1596,1363 | ||||||
| c | 34,5730 | 0,7198 | 33,1242 | 36,0219 | ||||||
| d | 35,9462 | 1,8215 | 32,2797 | 39,6126 | ||||||
| e | 3,4455 | 0,6226 | 2,1924 | 4,6987 | ||||||
| R2 | 0,997620 | |||||||||
| Curva de contagios | Parámetros | Valor | Error estándar | Intervalos de confianza 95 % | ||||||
| N
|
a | -99,7804 | 12,1061 | -124,1487 | -75,4122 | |||||
| b | 3626,7684 | 658,6780 | 2300,9183 | 4952,6185 | ||||||
| c | 44,0121 | 2,4684 | 39,0434 | 48,9808 | ||||||
| d | 2,4684 | 1,3322 | -0,2131 | 5,1499 | ||||||
| e | 0,1877 | 0,1050 | -0,0236 | 0,3990 | ||||||
| R2 | 0,999245 | |||||||||
| Curva de contagios | Parámetros | Valor | Error estándar | Intervalos de confianza 95 % | ||||||
| N
|
a | 13,3624 | 21,7584 | -30,4350 | 57,1598 | |||||
| b | 6421,2584 | 919,4884 | 4570,4242 | 8272,0926 | ||||||
| c | 45,5927 | 1,5001 | 42,5730 | 48,6123 | ||||||
| d | 3,5955 | 1,2262 | 1,1274 | 6,0636 | ||||||
| e | 0,4607 | 0,1846 | 0,0891 | 0,8323 | ||||||
| R2 | 0,99773 | |||||||||
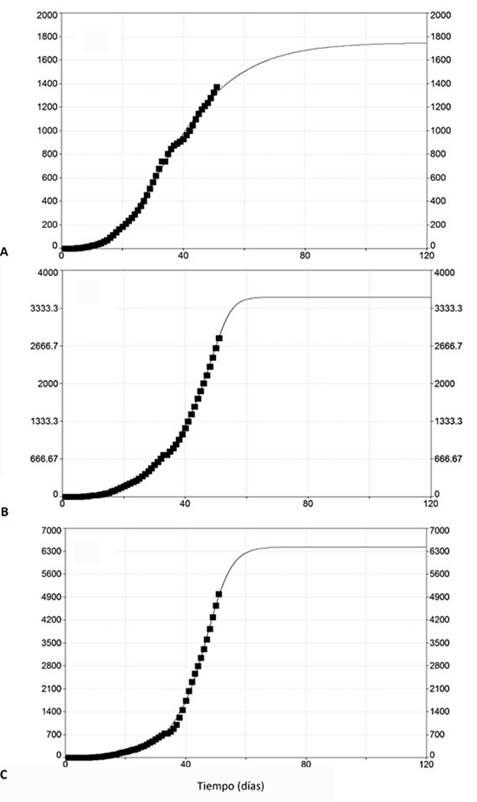
Fig. 5 Pronóstico extendido a 120 días a partir del ensamblaje de los registros de casos confirmados hasta el 16 de abril de 2020 y los escenarios de un periodo de incubación (14 días): a) mínimo -N - , b) medio N o y c) máximo N + , generados con modelo ARIMA (0,1,2)
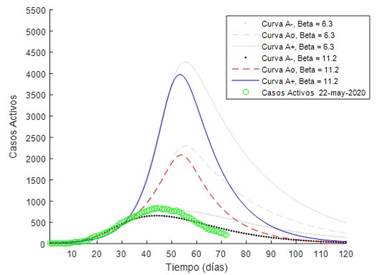
Como se mostró anteriormente, para la fecha de referencia 16 de abril de 2020, Cuba se encontraba ubicada en el cuartil 2. En este sentido, para evaluar posibles escenarios del comportamiento de los casos activos A, se consideraron los valores extremos de ( en el segundo cuartil, en este caso, ( = [( min , ( max ] = [1,82, 8,0], correspondientes a Italia y Luxemburgo, respectivamente. Un escenario intermedio e interesante es el correspondiente a Japón (( = 5,1), pues el indicador que relaciona recuperados y fallecidos hasta la fecha de referencia, era similar al de Cuba (( = 6,3). Vale la pena recordar que dicho parámetro es dinámico, ya que puede variar en el tiempo según el comportamiento de los acumulados de pacientes recuperados y fallecidos. Para la fecha del 29 de abril de 2020, el indicador ( para Cuba ascendió a 11,2; casi duplicando el reportado para el 16 de abril de 2020. Esto refleja la efectividad de los tratamientos y las medidas adoptadas por el gobierno y el sistema de salud cubano en el enfrentamiento de la COVID-19.
De esta forma para realizar la estimación de los posibles escenarios para Cuba de casos activos de la COVID-19, se utilizaron las ecuaciones (5-7) y (12-14) correspondientes al modelo propuesto, y los valores de los parámetros de las ecuaciones K R y K D , correspondientes a los países seleccionados del segundo cuartil (tabla 2 , países: Italia, Japón, Luxemburgo), con los registros de referencia hasta el 16 de abril de 2020:
Además, se introdujo el pronóstico extendido a 120 días con el modelo de transición SA, para el cual fueron generados los comportamientos de contagio N_,N o y N + , y en consecuencia se obtuvieron las curvas de casos activos A_,A o y A + , tal y como se observa en las Fig. 6 A), B) y C), respectivamente. Los registros de casos activos para Cuba hasta el 29 de abril de 2020 muestran un comportamiento dentro de la familia de curvas 𝐴 − para diferentes valores del indicador ( (figura 6C), mostrando una tendencia de los datos hacia la curva de mayor relación entre recuperados y fallecidos, lo cual se traduce en que la letalidad ((%) se ha comportado dentro de los valores más bajos del cuartil 2 donde se encuentra Cuba.
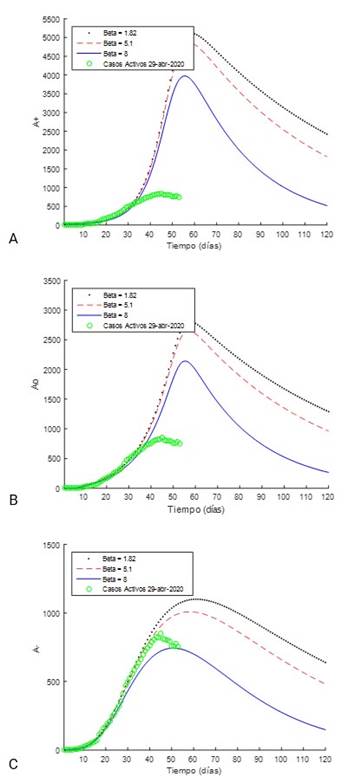
Fig. 6 Pronóstico extendido a 120 días de los casos activos para los escenarios de contagio_ a) máximo N + , b) medio N o y c) mínimo N - , generados con el modelo ARIMA (0,1,2), con fecha de referencia 16 de abril de 2020, teniendo en cuenta diferentes valores de (.
Teniendo en cuenta lo anterior, si realizamos un análisis más exhaustivo se puede apreciar que existe un desfasaje de 7 días en la estimación del pico de la curva, y que para el día 29 de abril de 2020 los registros descienden ligeramente por debajo del límite inferior de los escenarios preestablecidos (figura 7). Lo anterior puede ser un indicador de que los parámetros del modelo merecen ser reajustados, esta vez otorgándole mayor relevancia al parámetro X, sin riesgo de que este proceso implique grandes cambios del análisis conducido con anterioridad, pero buscando un mejor desempeño en la predicción de la evolución temporal de la COVID-19 en Cuba. En la tabla 4 se reportan los resultados obtenidos donde se aprecia una notable reducción de la función objetivo y una modificación apreciable de los parámetros.
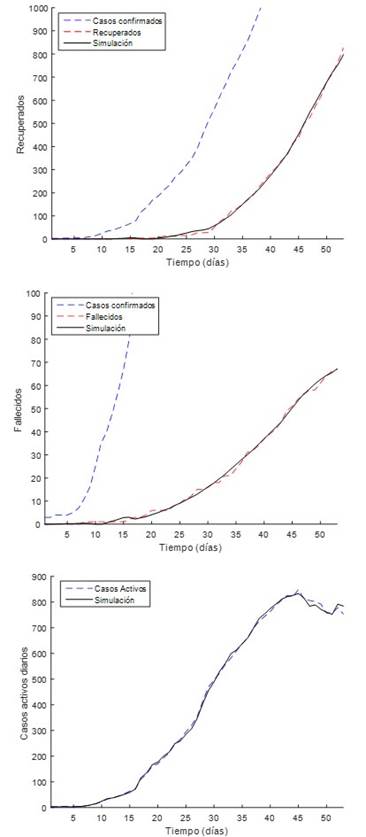
Fig. 7 Comparación entre los registros y las simulaciones de casos recuperados, fallecidos y activos de COVID-19 hasta el 29 de abril de 2020 en Cuba.
Tabla 4 Resultados del reajuste de parámetros del modelo propuesto
| Fecha registros | X
|
K
|
F.O (K
|
X
|
K
|
F.O (K
|
|---|---|---|---|---|---|---|
| 16 de abril de 2020 | 0,0000 | 35,4738 | 0,012130100 | 0,0000 | 30,584 | 0,00373333 |
| 29 de abril de 2020 | 0,2742 | 21,5861 | 0,000715188 | 0,2860 | 19,4338 | 0,00149226 |
En la figura 7 se observan las excelentes correlaciones alcanzadas. Nótese la reducción de K D en un 39,1 % y de K R en un 36,4 %, lo cual indica un incremento en el número de casos recuperados, pero también de los fallecidos. En este último caso, la letalidad para el 16 de abril de 2020 era de 3,13 %, mientras que para el 29 de abril de 2020 se presentaba un ligero incremento hasta el 4,1 %.
En la figura 8 se presentan las comparaciones entre los registros actualizados hasta el 22 de mayo de 2020 y las curvas de casos activos para los ajustes realizados con las fechas de referencia 16 de abril de 2020 (valores de ( = 6,3 - curvas de color gris) y 29 de abril de 2020 (valores de ( = 11,2). Manteniendo los escenarios de contagio definidos con anterioridad, puede observarse claramente como las curvas de casos activos se modificaron en cuanto a la amplitud, duración y simetría. Nótese que en todos los escenarios se redujeron los máximos esperados. Además, existe convergencia en cuanto al número de casos al cabo de los 120 días. Esto es de esperar ya que no son límites fijos, sino que se van reajustando durante el transcurso de la epidemia. Con los parámetros del modelo reajustado se pone de manifiesto que los registros transitan ligeramente por encima de la curva A - , existiendo muy buena coincidencia en los días en que se registran valores máximos, y es de esperar que exista convergencia en la duración, aun cuando durante el desarrollo de esta investigación la rama de descenso de la curva de registros aún no se encontraba totalmente desarrollada. La naturaleza dinámica de los intervalos A_,A o y A + permite reducir la incertidumbre que acompaña este tipo de problemas, y por tanto, contribuye a elevar la seguridad en la toma de decisiones para el enfrentamiento a este tipo de epidemias.
Conclusiones
El modelo propuesto inspirado en un proceso hidrológico permite pronosticar la acumulación de casos confirmados, fallecidos, recuperados y activos en su evolución a través del tiempo dentro de diferentes escenarios de la pandemia de la COVID-19, lo cual fue comprobado con la aplicación al caso de Cuba y podría utilizarse de la misma forma para otros países de América que tienen también un retardo con respecto a los países seleccionados para este estudio. El análisis de la evolución de la pandemia a nivel global con respecto a la relación entre recuperados y fallecidos ubicó a Cuba en el segundo cuartil entre los países de mayor desarrollo económico y social, a pesar de la diferencia entre el tiempo transcurrido de desarrollo de la epidemia en Cuba y en el resto de los países seleccionados. Por último, este modelo propuesto podría ser de utilidad para los tomadores de decisiones que trabajan en el enfrentamiento de la pandemia para pronosticar posibles escenarios de desarrollo de la misma, a través de la actualización periódica de los resultados obtenidos por medio de su implementación, teniendo en cuenta que el desarrollo de la pandemia es un proceso dinámico.