INTRODUCCIÓN
El Plan de Acción Mundial (PAM) publicado por la Organización Mundial de la Salud (OMS) en 2015 reconoce la importancia de la lucha contra la resistencia a los antimicrobianos (RAM), 1 respaldado por la Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO) y la Organización Mundial de Sanidad Animal (OIE). La Colaboración Tripartita reconoce que el desafío de la RAM debe abordarse con un enfoque de "Una salud”, OH por sus siglas en inglés (One Health) pues el desarrollo y la propagación de la RAM no respetan las fronteras entre sectores y, por tanto, requiere una colaboración intersectorial. 2,3
Escherichia coli comensal no patógena es un marcador establecido de contaminación fecal, también constituye un reservorio de genes de resistencia a los antimicrobianos (ARG). La plasticidad genómica de las cepas de E. coli patógenas y comensales contribuye a su desempeño como vehículo de ARG y justifica su utilización como “indicadora’’ en los planes de vigilancia integrada con el enfoque “Una salud’’. Los ARG en E. coli se consideran un reflejo de la presión selectiva debido al uso de antibióticos. 4) Aunque el conocimiento sobre E. coli-R-C3G en animales es limitado, especialmente en países de ingresos bajos y medios. En la actualidad prevalecen BLEE tipo CTX-M que transitaron de hospitales a la comunidad. 5
La resistencia a C3G en E. coli es comúnmente causada por la producción de β-lactamasas de espectro extendido (BLEE) codificadas por las familias de genes blaCTX-M, blaSHV y las enzimas AmpC del tipo blaCMY. Estos genes se localizan con frecuencia en elementos genéticos móviles, lo que contribuye a su transferencia a otras cepas. El análisis de la RAM en E. coli posibilita dilucidar su origen y vínculos epidemiológicos entre cepas derivadas de animales destinados a la producción de alimentos, el medio ambiente y la salud humana. Existen evidencias sobre el papel de las aves de corral y los cerdos como reservorio de E. coli-R-C3G en diferentes países del mundo. 6,7 En Cuba se informan CTX-M (15, 27, 32 y 55) en humanos, 8) pero no existen datos derivados de la producción animal. El trabajo tuvo como objetivos determinar la ocurrencia E. coli-R-C3G en aves y cerdos destinados a la producción de alimentos; definir los tipos de betalactamasas que portan las cepas; establecer rasgos de la epidemiología molecular de las cepas.
MÉTODOS
Aislamiento e identificación de E. coli-R-third generation cephalosporins en aves y cerdos. Selección de cepas y evaluación de la susceptibilidad in vitro
Se realizó un estudio descriptivo transversal dirigido a la caracterización molecular de E. coli-R-C3G procedentes de aves y cerdos de la región occidental de Cuba. Durante el período 2013-2015, se colectaron un total de 434 muestras de hisopados cloacales de aves sanas de granjas de gallinas ponedoras y una de pollos de engorde, ubicadas en la provincia Mayabeque, Cuba. La distribución de las muestras, es la siguiente 2013 (n = 139), 2014 (n = 145) y 2015 (n = 150). Durante el período 2016-2018 se tomaron 215 muestras de exudados rectales de cerdos sanos, mostrando la siguiente distribución: 2016 provincia Matanzas, municipio Matanzas (granja n = 42 y matadero n = 10); 2017 municipio Calimete (granja n = 42); 2018 provincia La Habana, municipio La Lisa (matadero n =57), municipio Caimito (matadero n = 12); provincia Mayabeque San José de las Lajas (granja n = 52). Las muestras se cultivaron en medios selectivos para Enterobacteriaceae R-C3G (chromID ESBL; bioMérieux, Marcy l’Etoile, Francia) y medio chromID CARBA (bioMérieux). E. coli se confirmó por espectrometría de masas MALDI-TOF. Las cepas se conservaron en glicerol a 20 ºC.
La selección de cepas de E. coli-R-C3G representativas de ambas colecciones derivadas de aves y cerdos, se basó en la relación genética por reacción en cadena de la polimerasa para elementos palindrómicos repetitivos (rep-PCR, de sus siglas en inglés) según condiciones descritas por Vila, J.1996 9 y el sitio de procedencia. Las cepas seleccionadas se utilizaron para la evaluación de la susceptibilidad in vitro y la caracterización genética. Los valores de concentración mínima inhibitoria (CMI) de 14 antibióticos para 32 cepas procedentes de aves y 27 de cerdos, se determinaron mediante microdilución en caldo Mueller-Hinton, utilizando placas de susceptibilidad EUVSEC Sensititre® (Thermo Fisher Scientific, Waltham, EE. UU.), siguiendo las directrices del fabricante. Se utilizaron los criterios propuestos por Comité Europeo para Pruebas de Susceptibilidad a los Antimicrobianos (EUCAST) 10 y por el Instituto de Estándares Clínicos y de Laboratorio (CLSI). 11
Caracterización genética de las cepas derivadas de aves
La identificación de genes de resistencia a antibióticos se realizó por microarreglos de ADN usando ArrayStrip_microarrays AMR08. Los genes de resistencia a β-lactámicos se confirmaron mediante PCR y secuenciación de ADN. 12,13 Las sustituciones de aminoácidos en la región determinante de la resistencia a la quinolina de GyrA y ParC se detectaron como se describió por Cano et al. 14 La tipificación por múltiples locus de tipos de secuencias (MLST, de sus siglas en inglés multi locus sequence typing) se realizó según el esquema de Achtman y la base de datos proporcionada por el University College Cork, Irlanda (http://mlst.ucc.ie/mlst/dbs/Ecoli). La similitud genética de los aislamientos E. coli-R-C3G se determinó mediante electroforesis en gel de campo pulsado (PFGE) usando la enzima XbaI.13 Los aislados se sometieron a tipificación de replicón de plásmido utilizando el kit comercial de tipificación basado en PCR (PBRT) (Diahtheva, Italia). PBRT determina los grupos Inc de las principales familias de plásmidos en Enterobacteriaceae.15,16
Caracterización genética de las cepas derivadas de cerdos. Secuenciación de genomas completos y análisis in silico
Se extrajo ADN de 27 cepas mediante el kit AMPure XP (Beckman Coulter). La concentración se cuantificó en el fluorómetro Qubit® 3.0 (Invitrogen) y se ajustó a 1,5 μg de ADN. La secuenciación se realizó mediante la tecnología Illumina en la plataforma NovaSeq6000 (Illumina). Las lecturas se sometieron a control de la calidad mediante la herramienta FASTQC 0.11.7 (https://github.com/s-andrews/FastQC). Los genomas se ensamblaron de novo mediante SPAdes v3.12 y se visualizaron mediante Geneious v10.1.2 (Biomatters). Los genomas se analizaron in silico con las herramientas bioinformáticas en línea del Centro de Epidemiología Genómica (http://www.genomicepidemiology.org) ResFinder v3.1 detección de genes adquiridos y mutaciones de resistencia antibiótica, MLST v2.0 basados en el esquema de Achtman para E. coli y PlasmidFinder v2.0 para detectar grupos de incompatibilidad (grupos o tipos Inc) plasmídica.
Análisis de la estructura circular completa de un genoma representativo de la colección de aislados
Se seleccionó un aislado (PK6 blaCTX-M-32). el ADN genómico se fragmentó usando el dispositivo MinION Mk1B (Oxford Nanopore Technologies). La lectura automática de bases se realizó mediante la herramienta Guppy basecaller (v2.3.7) y Guppy barcoder (v2.3.7), respectivamente (Oxford Nanopore Technologies). El genoma de la cepa PK6 se analizó in silico con las herramientas bioinformáticas descritas anteriormente.
RESULTADOS
Del total de muestras de aves (434), se recuperaron 62 aislamientos de E. coli-R-C3G, en todas las granjas investigadas, 22 de gallinas y 40 de pollos de engorde. Con relación a las muestras colectadas de cerdo (215), se confirmó la presencia de 96 cepas de E. coli-R-C3G, 65/136 derivadas de granjas y 31/79 de mataderos, mostrando la siguiente distribución Matanzas 15/52 (28,8 %), Calimete 28/42 (66,7 %), La Lisa 20/57 (35 %), Caimito 8/12 (66,7 %) y San José 25/52 (48 %). Acorde a sus perfiles de rep-PCR y la procedencia se seleccionaron 32 cepas de la colección de aves y 27 derivadas de cerdos, para la tipificación en cuanto a susceptibilidad antimicrobiana, características genéticas y epidemiología molecular.
Todas las cepas de E. coli recobradas de aves y cerdos fueron sensibles a colistina, carbapenemes y resistentes a cefotaxima en la prueba de susceptibilidad in vitro. También mostraron resistencia a otras clases de antibióticos. Las cepas de aves mostraron los siguientes porcentajes de resistencia: tetraciclina (81,0 %), sulfametoxazol (59,0 %), trimetoprim (56,0 %), ácido nalidixico (72,0 %), ciprofloxacina (50,0 %), chloramphenicol (34,0 %) y gentamicina (28,0 %). Las cepas de origen porcino también mostraron altos porcentajes de resistencia a tetraciclina (94,8 %), sulfametoxazol (75,0 %), trimetoprim (69,8 %) chloramphenicol (44,8 %), ácido nalidixico (51,0 %), ciprofloxacina (31,3 %), gentamicina (27,1 %) y tigeciclina (4,2 %).
En ambas colecciones de cepas y en correspondencia con los perfiles de resistencia fenotípica se encontraron genes que soportan la resistencia a estas familias de fármacos: sulfonamidas (sul1, sul2, sul3), trimetoprima (dfrA1, dfrA7, dfrA12, dfrA14, dfrA17), tetraciclinas [tet (A), tet (B)] y cloranfenicol (cmlA1, floR). Algunos de los aislamientos también contenían los genes de resistencia a la estreptomicina strA y strB, el gen de resistencia a la estreptomicina/espectinomicina aadA4, y a macrólidos mph(A) y mph (D).
La resistencia a las fluoroquinolonas en las cepas se asoció con la sustitución de aminoácidos en la región que determina resistencia a quinolonas (QRDR, de sus siglas en inglés quinolone resistance- determining regions) que se localiza en las topoisomerasas. Las cepas de aves mostraron los siguientes cambios en la región QRDR, en Gyrasa A S83-L (n = 15), D87-N (n=13), D87-Y (n = 1), ParC S80-I (n= 12) y E84-G (n = 2). Además, se encontraron los genes qnr y aac(6)-Ib-cr, que confieren bajos niveles de resistencia a las fluoroquinolonas en 17 y 15 cepas respectivamente (figura1). En 9 cepas de origen porcino que mostraron altos niveles de resistencia a ciprofloxacina y ácido nalidíxico se encontraron las siguientes sustituciones de aminoácidos en la región QRDR, GyrA (S83L, D87N, D87Y), ParC (S80I) y además en ParE (S458A). Mientras en 13 cepas con una susceptibilidad disminuida se encontraron solo genes qnrB19 y qnrS1, sin mutaciones en la región QRDR de las topoisomerasas. Hubo 4 aislados que contenían qnrB19 o qnrS1 y exhibieron baja resistencia frente a ácido nalidíxico (figura 2).
La caracterización de los genes de resistencia a C3G reveló que 57/59 cepas de E. coli aisladas de ambas especies contenían algún alelo del gen blaCTX-M. En E. coli de origen aviar se notificaron las variables: blaCTX-M-1 (n = 28), blaCTX-M-15 (n = 4) y blaLAP-2 (n = 1) (figura 1) y en las cepas derivadas de cerdos blaCTX-M-32 (n = 17), seguido de blaCTX-M-15 (n = 5) y blaCTX-M-55 (n = 4). En todas las cepas de cerdos que portaban el gen blaCTX-M-55 se detectó también el gen blaTEM-1B. Otro gen relacionado con la resistencia a C3G que se identificó en cepas de porcino fue blaCMY-2 que codifica para la variante pAmpC CMY-2 (figura 2).
Las cepas de aves predominantes fueron positivas para BLEEs tipo CTX-M-1, pertenecieron a 19 tipos de secuencias (ST), albergaban plásmidos de varios grupos de incompatibilidad y se detectaron en el 85 % (6/7) granjas. Por el contrario, los aislados que contenían CTX-M-15 pertenecían a sólo 2 ST relacionadas filogenéticamente, las ST410 y ST90, aunque procedían de diferentes granjas (figura 1).
El análisis por MLST de las cepas de cerdos mostró una alta diversidad clonal; se encontraron 16 ST de las cuales las ST9360, ST9361 y ST9362 representaron nuevas ST, que se depositaron en la base de datos Enterobase (http://enterobase.warwick.ac.uk/species/ecoli/). El gen blaCTX-M-32 fue predominante, se encontró en cepas pertenecientes a 12 ST distribuidas en granjas y mataderos de todas las provincias estudiadas. Los genes blaCTX-M-15 y blaCTX-M-55 se encontraron solamente en dos ST cada uno (ST162, ST648) y (ST457, ST2599), respectivamente. El gen blaCMY-2, se encontró en dos cepas pertenecientes a la ST648, uno de ellas también contenía el gen blaCTX-M-15 (figura 2). En cuanto a la diversidad de replicones de plásmidos en las cepas de origen porcino, se detectaron 13 grupos Inc, siendo IncX1 el tipo más predominante y algunos aislamientos albergaron 2 o más tipos de Inc (figura 2).
El genoma completo de la Cepa PK6 de cerdos constó de 5 cóntigos circulares con una suma de 4 789 128 pb y un contenido de G + C del 50,8 %, 4582 secuencias codificantes. Los genes (QnrB19, TEM- 1B, Tet(B), GyrA (S83L), ParC (S80I) de resistencia a antibióticos se detectaron en el cromosoma, mientras en el plásmido IncX1 pRHEcCUB-1 de 42 683 pb se confirmaron los siguientes genes AadA1, AadA2, CmlA1, DfrA12, QacH2, Sul3 (In640); CTX-M-32. En este plásmido además se detectó un gen de resistencia a desinfectantes de compuestos de amonio cuaternario. 17
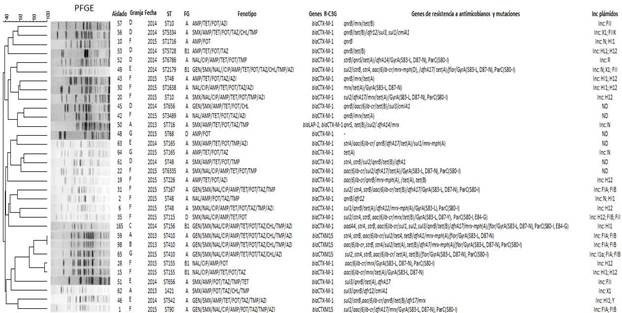
Fig. 1 . Características genéticas y árbol filogenético construido a partir del patrón de electroforesis en gel de campo pulsado (PFGE) de 32 aislados de E. coli resistentes a cefalosporina de tercera generación (Ec-R-C3G) de aves de corral en la provincia Mayabeque. Cepa: cepa Id); las granjas se representan con las letras de la A-G; ST: tipo de secuencia; PhG: filogrupo; Inc plásmidos: grupos de incompatibilidad de plásmidos. Antibióticos AMP: ampicilina; CAZ: ceftazidima; CTX: cefotaxima; CIP: ciprofloxacina; NAL: ácido nalidíxico; GEN: gentamicina; SMX: sulfametoxazol; TMP: trimetoprima; TET: tetraciclina; CHL: cloranfenicol; AZI: azitromicina. Genes y funciones de resistencia a los antimicrobianos: genes d β-lactamasa de espectro extendido blaCTX-M para resistencia a cefalosporinas de tercera generación, aadA4: gen de nucleotidiltransferasa de aminoglucósidos para resistencia a estreptomicina y espectinomicina; cmlA1, gen de eflujo de cloranfenicol; flor, gen de resistencia al florfenicol / cloranfenicol; dfrA1, dfrA5, dfrA17 y drfA19 = gen de dihidrofolato reductasa para la resistencia a trimetoprima; tet(A), tet(B), gen de eflujo de tetraciclina; strA, strB, gen de estreptomicina fosfotransferasa; sul1, sul2, sul3 = gen dihidropteroato sintasa para la resistencia a la sulfonamida; mrx-mph (A), mrx-mph (D) grupo de genes de inactivación de macrólidos; mph (A), mph (D) macrólidos fosfotransferasas; proteína hidrófoba mrx con función desconocida; aac(6')- Ib-cr, gen aminoglucósido N (6') - acetiltransferasa-cr para resistencia a amikacina, kanamicina y quinolona; qnrB, qnrS, gen de protección de la ADN girasa para un bajo nivel de resistencia a las fluoroquinolonas. GyrA (S83-L), (D87-N), (D87-Y) y ParC (S80-I), (E84-G), sustituciones de aminoácidos en topoisomerasa GyrA y ParC confiere altos niveles de resistencia a las fluoroquinolonas.

Fig. 2 Árbol filogenético basado en el método Neighbor-Joining (NJ) y en el esquema de cgMLST estable para la especie E. coli que incluye 2513 genes, de 27 aislados secuenciados de E. coli-R-C3G procedentes de cerdos sanos en Cuba. La figura además incluye la distribución de los tipos de secuencia (ST) (n = 12), tipos de incompatibilidad (Inc) (n = 13), genes de resistencia a antibióticos (n = 39) y de virulencia (n = 9) presentes en los aislados secuenciados de. E. coli-R-C3G. Leyenda: Antibióticos: C3G, cefalosporinas de tercera generación; AMP, ampicilina; EST, estreptomicina; KAN, kanamicina; GEN, gentamicina; SUL, sulfonamidas; QNL, quinolonas; TET, tetraciclinas; CLF, cloranfenicol; TMP, trimetoprim; Otros: macrólidos, lincosamidas, fosfomicina. Lugares de muestreo: CLM/G: Granja de Calimete; LLA/M/G: Matadero de La Lisa (cerdos procedentes de granja en La Lisa); MAT/G: Granja en Matanzas; MAT/S: Matadero en Matanzas; SJL/G: Granja en San José de Las Lajas; LLA /M-CAI / G: Matadero en La Lisa (cerdos procedentes de granja en Caimito).
DISCUSIÓN
Las infecciones por bacterias productoras de BLEE se asocian a estancias hospitalarias prolongadas y cada vez se informan con más frecuencia a nivel comunitario. 18 Sin embargo, aún no se estima la carga global de estas infecciones en los animales de granja. La transmisión puede ocurrir de persona a persona, o de fuentes animales a personas a través de la cadena alimentaria. 4 Por tanto, el papel potencial de los animales como reservorio de estos microorganismos para los seres humanos hace necesario un enfoque interdisciplinario de "Una salud" para mantener esta amenaza bajo control. 3
En este estudio se demostró la ocurrencia de cepas comensales de E. coli-R-C3G, en muestras de aves y cerdos sanos, lo cual coincide con informes generados en otros países que mostraron su prevalencia en la crianza animal. 19,20 Este es el primer informe en Cuba que notifica en E. coli de aves las variantes CTX-M-1 y CTX-M-15 y en cepas de cerdos CTX-M-32, CTX-M-15 y CTX-M-55. Ambas colecciones de cepas mostraron altos niveles de resistencia a tetraciclinas y fluoroquinolanas, antibióticos clínicamente importantes en medicina humana y veterinaria. Las cepas bacterianas resistentes a C3G se aíslan de animales de producción, aun cuando se crían en ausencia de cefalosporinas. Esto ocurre por la coselección de los genes de resistencia a antibióticos no betalactámicos que se localizan en los mismos elementos genéticos que transportan genes para la expresión de BLEE. 21 Por tanto el uso de tetraciclinas, aminoglucósidos, sulfonamidas y trimetoprim, que en Cuba están autorizados para la producción animal, 22 podría favorecer la coselección de estas cepas. La observancia de cepas indicadoras de E. coli, a través de su fondo genético para la RAM pueden ser un reflejo del uso de los antibióticos, requeriendo implementar medidas para preservar su efectividad. 23
El tipo blaCTX-M-1 fue abundante en E. coli de aves, esta tendencia también se ha observado en Europa. 24,25,26 Aunque el grupo CTX-M-1 es amplio y comprende una diversidad de subtipos, que requieren de estudios adicionales basados en técnicas de secuenciación de última generación para esclarecer con precisión cuales albergan las cepas de aves. Sin embargo, en E. coli de cerdos blaCTX-M-32 fue dominante. Es posible que las cepas que albergan ambas variantes blaCTX-M-1 y blaCTX-M-32 en aves y cerdos respectivamente, aprovechen una ventaja selectiva particular, por colonización o adaptación metabólica al aparato digestivo en estos animales.
Los resultados indicaron que cepas de E. coli CTX-M están extendidas en instalaciones de producción de aves y cerdos en la región occidental, lo cual no parece ser un evento esporádico. La diversidad de las cepas, su baja tasa de clonalidad, incluso dentro de una misma granja, la presencia del mismo grupo de incompatibilidad plásmidica y el tipo de BLEE en diferentes granjas, señala como la vía más activa de propagación la transferencia horizontal de elementos genéticos móviles con limitaciones a estos sitios o quizás por la adquisición de múltiples fuentes.
Los mecanismos de transmisión de bacterias productoras de β-lactamasas aún no están esclarecidos, por ellos es necesario considerar posibles fuentes de contaminación múltiples, como el ambiente, las moscas, heces de otros animales y humanos. 27,28 Como los cerdos y las aves de corral forman parte de líneas de producción en las que se aplican medidas de bioseguridad, podría ser menos probable que se introduzcan cepas procedentes de una amplia variedad de fuentes en su entorno, de ahí la importancia de reforzar las medidas de higiene y saneamiento en estas instalaciones. (29 La presencia de E. coli R-C3G en exudados cloacales de aves supone un riesgo para la producción y manipulación de huevos, principal renglón derivado de la producción aviar en Cuba. El huevo y las heces comparten el mismo entorno dentro y fuera del ave. Estos resultados indican fortalecer las buenas prácticas de higiene y saneamiento para minimizar el riesgo de propagación de determinantes de la resistencia a los antimicrobianos.
Este estudio permitió obtener y recopilar información preliminar sobre la diversidad de E. coli portadora de blaCTX-M y sus respectivas variantes genéticas en aislados de pollos y cerdos. El tipo de secuencia 131, la más relevante desde el punto de vista clínico en los seres humanos, no se encontró. Sin embargo, se detectaron cepas de las ST 410, 10 y 648 de importancia clínica en humanos que han sido informadas en otras partes del mundo 7,20,21 y en Cuba. 8
Recientemente, Quiñones y col. (2020) 8 informaron la presencia de los genes blaCTX-M-32, blaCTX-M-15, blaCTX-M-55 y blaCMY-2 en cepas de E. coli resistentes a C3G recobradas de infecciones extrainstestinales en humanos en Cuba, la variante blaCTX-M-15 se encontró en el mayor porcentaje de los aislados. Interesantemente la variante blaCTX-M-32, se detectó en un paciente en Matanzas, también mostró predominio en las muestras de cerdos colectadas de esta provincia. Algunos autores han sugerido que CTX-M-55 y CTX-M-32 proceden de fuentes animales, incluidos ganado bovino y porcino. 30 Los aislados de ST410 recobrados en aves y de ST 648 y 162 de cerdos contienen CTX-M-15, el tipo de BLEE predominante en E. coli humana a nivel mundial. 31
Uno de los elementos más importantes a considerar en la diseminación de la RAM es la presencia de genes de resistencia localizados en plásmidos autotransferibles. En este estudio se detectaron diferentes tipos de replicones en las cepas multirresistentes de E. coli productoras de BLEE derivados de aves y cerdos. Además el genoma circular ensamblado de la cepa PK6 corroboró la localización del gen blaCTX-M-32 en el plásmido pRHEcCUB-1, experimentos de conjugación serían necesarios para esclarecer la capacidad para su diseminación.
La relación genética entre cepas de diferentes orígenes y los elementos genéticos que contienen CTX- M aún no se ha investigado en Cuba; tal investigación requeriría la caracterización basada en el genoma completo de los aislados con énfasis en plásmidos e información epidemiológica a gran escala. (32,33 En este estudio se detectaron diferentes tipos de replicones en las cepas multirresistentes de E. coli productoras de BLEE derivados de aves y cerdos. Además, el genoma circular ensamblado de la cepa PK6 corroboró la localización del gen blaCTX-M-32 en el plásmido pRHEcCUB-1, experimentos de conjugación serían necesarios para esclarecer la capacidad para su diseminación. La secuenciación de genomas completos es de gran utilidad en los programas de vigilancia integrada, permite distinguir bacterias con patrones de resistencia idénticos, pero causados por diferentes mecanismos, facilitando un nivel de detalle que refinará la comprensión de la resistencia. 34
Conclusiones
Los datos muestran que genes de resistencia a múltiples antibióticos, junto a los genes BLEE se albergan en diversos linajes de E. coli comensal para cerdos y aves. Aunque se observa diseminación de algunos clones, se propone como principal vía para la propagación de los genes de resistencia la transferencia horizontal. Estos hallazgos son indicativos de posibles fallos en la gestión del uso de antibióticos en el sector de la producción animal o de falta de higiene durante el proceso.














