INTRODUCCIÓN
El receptor del factor de crecimiento epidérmico (EGFR, del inglés epidermal growth factor receptor) es uno de los principales blancos para el desarrollo de terapias antitumorales.1 Entre estas se encuentran los anticuerpos monoclonales (AcM), de los cuales solo se han aprobado para uso clínico el nimotuzumab (2002), el cetuximab (2004), el panitumumab (2006) y el necitumumab (2015).2 A pesar de que todos reconocen la región extracelular del EGFR (reEGFR), el resultado clínico en términos de eficacia y seguridad difiere. Las propiedades de los anticuerpos que determinan sus efectos son el origen de los dominios constantes (especie e isotipo), y la afinidad y especificidad epitópica fina de sus dominios variables.
El desarrollo de resistencia a la terapia con cetuximab y panitumumab producto de mutaciones en los epitopos reconocidos por los mismos,3-7 y el hecho de que la susceptibilidad a las mutaciones difiera entre los AcM, a pesar de que reconozcan epitopos estrechamente relacionados, determina la necesidad de un estudio detallado de cada epitopo. Si bien se ha logrado comprender las bases moleculares del comportamiento de otros anticuerpos,8-11 la interfaz nimotuzumab-EGFR no se había caracterizado experimentalmente con anterioridad. Su exploración completaría el escenario molecular de los AcM anti-EGFR utilizados en la clínica y contribuiría a seleccionar la mejor opción de tratamiento.
Por otro lado, la afinidad impacta directamente sobre la toxicidad y el efecto antitumoral. De los AcM anti-EGFR en la clínica, el nimotuzumab es el menos tóxico.12 Este perfil de seguridad tan favorable se ha atribuido a su afinidad intermedia.13 No obstante, el beneficio del tratamiento con nimotuzumab está sesgado hacia el grupo de pacientes con altos niveles de EGFR.14 Existen evidencias de que la alta afinidad de los AcM anti-EGFR está asociada con un mayor beneficio en la supervivencia de los pacientes.15 La ventana de afinidad ideal, que resulte en un balance óptimo entre la efectividad antitumoral y la mínima toxicidad, no se conoce. Disponer de variantes de un AcM con afinidades diferentes dirigidas contra el mismo epitopo sería útil para explorar esta dependencia. Las variantes del nimotuzumab con mayor afinidad podrían tener una mayor aplicabilidad en el diagnóstico y tratamiento de tumores EGFR positivos.
Las investigaciones que se compilan en el presente trabajo estuvieron dirigidas a caracterizar la interfaz funcional de unión entre el nimotuzumab y el EGFR,16 y obtener variantes del nimotuzumab con mayor afinidad por el receptor que conservaran su especificidad epitópica fina.17
MÉTODOS
Obtención y caracterización de las variantes del dominio III sobre fagos
El gen que codifica para el dominio III del EGFR humano (DomIII) se clonó en el vector fagomidio pHAB18 y sirvió de molde para las reacciones de mutagénesis de Kunkel modificada19,20 con oligonucléotidos degenerados. Los fagos presentadores del DomIII se produjeron y purificaron según un protocolo descrito.21 La antigenicidad de las variantes del DomIII sobre fagos se evaluó por ELISA en placas recubiertas con los AcM anti-EGFR y el AcM 9E10 (antietiqueta c-myc). Los fagos unidos se detectaron con el AcM anti-M13 conjugado a la peroxidasa de rábano picante (HRP, del inglés horseradish peroxidase) (GE Healthcare, EUA).
Construcción de bibliotecas, selección y caracterización de fagos presentadores de las regiones variables del nimotuzumab
Los genes que codifican para las regiones variables de las cadenas pesada (VH) y ligera (VL) del nimotuzumab se clonaron en los vectores fagomidios pHAB (formato de fragmentos de anticuerpo de cadena única, scFv, del inglés single chain Fv)16 y pCS-1(formato Fab).17 Los mismos sirvieron de molde para la construcción de las bibliotecas mediante mutagénesis de Kunkel modificada19,20 con oligonucleótidos degenerados. La selección de los fagos presentadores de los fragmentos del nimotuzumab se realizó contra una proteína recombinante correspondiente a la reEGFR inmovilizada.22 Los niveles de presentación y las propiedades de unión de las variantes se evaluaron por ELISA sobre placas recubiertas con el AcM 9E10 y con la reEGFR. Los fagos unidos se detectaron con un AcM anti-M13-HRP (GE Healthcare, EUA).
Modelación in silico de la interacción nimotuzumab-dominio III
Las simulaciones de acoplamiento se realizaron con el programa Rosetta Commons 3.5.23 Las estructuras del Fv del nimotuzumab24 y del complejo cetuximab/EGFR8 se superpusieron, y el complejo Fv nimotuzumab/EGFR resultante se tomó como punto de partida para el acoplamiento a ciegas. Se realizaron varios ciclos de perturbación y filtrado, según los criterios de calidad típicos del acoplamiento y los datos experimentales. Adicionalmente se realizaron experimentos de mutagénesis in silico para escoger el modelo más compatible con los estudios funcionales.16
Obtención y caracterización de los AcMs K4 y K5
Los genes que codifican para las VH mutadas de los AcMs K4 y K5 se construyeron por mutagénesis de Kunkel modificada,19,20 para combinar las mutaciones seleccionadas por mostrar mayor reactividad por la reEGFR. Los genes de las VH (original y mutadas) y de la VL original se clonaron en los vectores de expresión para células de mamíferos pSV-gpt (que codifica para la región constante IgG1humana) y pSV-hyg (región constante kappa humana), respectivamente.25 Con las construcciones genéticas resultantes se electroporaron células de mieloma NS0 para obtener clones productores de los 3 AcM. Se purificaron los anticuerpos mediante cromatografía de afinidad por proteína A. A partir de los AcM purificados se obtuvieron los fragmentos Fab mediante digestión con papaína con el juego de reactivos Pierce Fab preparations (Thermo Scientific, EUA). Los parámetros cinéticos kon, koff y la constante de equilibrio de disociación KD se determinaron con un BIAcore T200, sobre un chip biosensor CM5 con la reEGFR inmovilizada. Los sensorgramas se analizaron con el software de evaluación 3.0 del instrumento. Los datos se ajustaron al modelo de interacción 1:1.
Producción y caracterización de las variantes de la reEGFR en células de mamíferos
Los genes que codifican para la reEGFR humano y de ratón se clonaron en el vector pCSE2.5-His-Xp.26 Las variantes de la reEGFR se construyeron con el juego de reactivos Quickchange II site-directed mutagenesis (Agilent Technologies, EUA). Los plasmidios resultantes se utilizaron para transfectar transitoriamente células HEK293-6E en suspensión, según un protocolo descrito,26 y obtener las proteínas de interés, las cuales se purificaron por cromatografía de afinidad a metales inmovilizados (IMAC). Para evaluar el reconocimiento de la reEGFR por las variantes, se recubrieron placas con nimotuzumab, AcMK4 y AcMK5. Las moléculas unidas se detectaron con un anticuerpo de ratón anti-etiqueta 6xHis (Dianova, Alemania), seguido de un anticuerpo anti-Fc de ratón-HRP (Sigma, EUA).
Ensayo de inhibición de la fosforilación del EGFR
Se incubaron células de las líneas MDA-MB-468 y H125 (con alta y moderada expresión del EGFR, respectivamente) con los anticuerpos anti-EGFR. Se retiró el medio y las células se estimularon durante 10 min con 100 ng/mL de EGF humano. A partir de las células tratadas se prepararon lisados celulares. Se transfirieron iguales cantidades de proteínas a una membrana de PVDF (Sigma, EUA). El EGFR fosforilado y el total se detectaron con los anticuerpos producidos en conejo anti-Phospho EGFR (Y1068) (Cell Signaling, EUA) y anti-EGFR (sc-03) (Santa Cruz Biotechnology, EUA), respectivamente, seguidos del anticuerpo anti-conejo-HRP (Cell Signaling, EUA). La señal se desarrolló con un sustrato quimioluminiscente (Santa Cruz Biotechnology, EUA).
RESULTADOS
La exploración de una región antigénica amplia delineó el mapa funcional del epitopo reconocido por el nimotuzumab, la manipulación del DomIII, correctamente plegado sobre fagos filamentosos fue la base de la estrategia de mapeo (figura 1). La evaluación por ELISA de un panel de variantes con mutaciones individuales interespecies (humano/ratón) culminó con la identificación del residuo H359 como clave para la formación del epitopo del nimotuzumab. El perfil de tolerancia a las mutaciones en la vecindad de este aminoácido permitió definir un mapa funcional del epitopo en su conjunto, formado por los aminoácidos S356-H359, contiguos en la secuencia primaria del Dom III y el residuo R353 (figura 2A). Los residuos S356 y H359 fueron absolutamente críticos para la formación del epitopo. Otros residuos (F357, T358 y R353), aunque no fueron imprescindibles para el reconocimiento por el AcM, solo se sustituyeron por residuos que comparten propiedades con el aminoácido original, lo que indicó una contribución funcional de sus interacciones. El papel de la R353 (localizada dentro del epitopo estructural del cetuximab) en la unión del nimotuzumab mostró que ambos epitopos se solapan (figura 2B), lo que explica la competencia entre estos AcM.24 Por otro lado, los residuos S356, F357 y T358 están involucrados directamente en la interacción del nimotuzumab, del EGF27 y del TGF-α28 con el DomIII (figura 2C), lo que explica el efecto inhibitorio del nimotuzumab sobre la activación del EGFR mediada por sus ligandos.
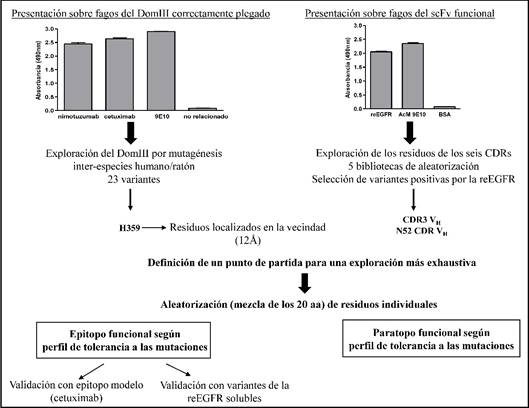
Fig. 1 Representación esquemática de la estrategia de mapeo funcional de la interfaz de unión nimotuzumab-DomIII (epitopo/paratopo).

Fig. 2 Interfaz funcional de unión nimotuzumab-DomIII. El DomIII del EGFR se muestra en cintas blancas y superficie semitransparente. Las regiones VH y VL del nimotuzumab se muestran en cintas y superficie semitransparente de colores cian y rosado, respectivamente. A) El epitopo reconocido por el nimotuzumab, según los experimentos de mutagénesis se muestra en rojo. B) Localización relativa del epitopo funcional del nimotuzumab (rojo) y el epitopo estructural del cetuximab (amarillo) según PDB 1YY9. El residuo R353, que pertenece a ambos epitopos, se resalta en naranja. C) Solapamiento entre el epitopo funcional del nimotuzumab y los sitios de unión a los ligandos. La interfaz entre el DomIII del EGFR y el EGF y TGF-α (residuos a 5Å del EGF y TGF-α en los complejos receptor/ligando, estructuras PDB 1IVO y 1MOX respectivamente) se coloreó en azul. Los residuos S356, F357 y T358 (resaltados en magenta) están involucrados en la unión de ambos ligandos y del nimotuzumab. El resto de los residuos del epitopo del nimotuzumab se muestran en rojo. D) Representación del paratopo del nimotuzumab a partir de la estructura del fragmento Fv (PDB 3GKW). En verde se muestran los residuos de los CDR2 y 3 de la VH críticos para la unión. El resto de los residuos explorados de la VH y de la VL que no contribuyen a la unión se muestran en azul marino y carmelita, respectivamente. E) Representación en cintas de la estructura del complejo nimotuzumab/DomIII según el modelo computacional seleccionado. El epitopo estructural predicho está en violeta, e incluye al epitopo funcional (rojo). Las imágenes se generaron con el programa Pymol.
El primer paso fue la presentación exitosa sobre fagos filamentosos del antígeno y del paratopo (en forma de fragmento variable de cadena única, scFv) del nimotuzumab. El segundo paso fue la identificación dentro del antígeno y del paratopo de una zona reducida que con una alta probabilidad contribuyera energéticamente en la unión. Una vez identificada la región candidata se exploró extensivamente mediante mutagénesis sitio-específica, con el objetivo de delinear el mapa de interacciones epitopo/paratopo. En el caso del epitopo, la realización de estudios adicionales de mapeo de un epitopo modelo (el del anticuerpo cetuximab) y la recapitulación de los resultados obtenidos en fagos con proteínas solubles, contribuyó a validar los resultados.
Paralelamente, se exploraron los residuos incluidos en el epitopo del cetuximab.8 La falta de tolerancia a múltiples sustituciones confirmó el papel clave de la K443, K465 e I467 en la unión del cetuximab4,29 y validó el método de mapeo del presente trabajo. La mayoría de las mutaciones mostraron un efecto divergente sobre la unión del nimotuzumab y el cetuximab al Dom III, lo que indicó que la especificidad fina de estos AcM es diferente.
Los resultados obtenidos con fagos se complementaron con estudios de mutagénesis sobre la reEGFR recombinante producida en células de mamífero, con un adecuado plegamiento y modificaciones postraduccionales.
Se confirmó que los reemplazos conservativos S440T, K443H y S356T, F357L eliminaron específicamente el reconocimiento por el cetuximab y el nimotuzumab, respectivamente (figura 3A). La reconstrucción exitosa de los epitopos del nimotuzumab (R359H) y del cetuximab (M467I) sobre una proteína no reconocida (reEGFR de ratón, figura 3B), constituyó la prueba definitiva de la confiabilidad de los resultados del mapeo.
La exploración por mutagénesis combinatoria e individual mostró el papel dominante de los residuos del CDR3 de la cadena pesada dentro del paratopo del nimotuzumab en el reconocimiento por el antígeno.
El paratopo del nimotuzumab (representado por el scFv presentado sobre fagos) se exploró mediante mutagénesis combinatoria e individual (ver figura 1). El estudio combinatorio concluyó que la VL y la mayoría de las posiciones de los CDR1 y 2 de la VH no tienen un papel crítico en la unión, pues aceptaron múltiples residuos con diferentes propiedades físicas y químicas. El grupo de variantes positivas al antígeno se distinguió por la presencia del residuo original en las posiciones 95, 96, 97, 98, 100A, 100B, 100C y 100E (CDR3 de la VH). Otros residuos del CDR2 y 3 de la VH (N52, F99 y D100), más que una estricta conservación del aminoácido original, mostraron una gran abundancia del mismo, solo reemplazado por residuos con propiedades químicas y topologías similares en cada caso. La relevancia del residuo N52 del CDR2 de la VH y de casi la totalidad de los residuos del CDR3 de la VH se comprobó mediante experimentos de mutagénesis individual, que confirmaron que los mismos contribuyen a la formación del paratopo funcional del nimotuzumab (ver figura 2D). La información obtenida a partir del mapeo funcional guió la generación de un modelo in silico de la interacción entre el nimotuzumab y el EGFR
La simulación a ciegas del acoplamiento entre el fragmento Fv del nimotuzumab y el DomIII del EGFR culminó con un modelo del complejo nimotuzumab reEGFR, en concordancia con los datos funcionales. Según este modelo, el grupo de residuos pertenecientes al epitopo funcional constituyen la parte principal del epitopo estructural predicho (ver figura 2E), y están semiacoplados en una cavidad superficial formada principalmente por el CDR3 de la VH, pero también por el CDR3 de la VL y en menor medida por el CDR1 de la VL (ver figura 2E).
La evolución in vitro del Fab derivado del nimotuzumab resultó en la obtención de AcM con mayor afinidad por el EGFR e igual especificidad A partir de la estrategia de maduración de la afinidad empleada (ver figura 4) se obtuvieron 2 AcM (K4 y K5) derivados del nimotuzumab. Los fragmentos Fab derivados de los 3 anticuerpos se compararon en términos de afinidad intrínseca, mediante BIAcore.
La figura 4 muestra un incremento modesto pero consistente en la afinidad de unión por la reEGFR inmovilizada para los AcMs K4 y K5 (3 y 3,6) veces con respecto al nimotuzumab. Si bien los nuevos residuos incorporados en el CDR2 de la VH de los anticuerpos de mayor afinidad establecieron nuevas interacciones, suficientes para modificar la afinidad, la especificidad fina del nimotuzumab se conservó. El análisis del reconocimiento de la colección de variantes mutadas del DomIII presentadas sobre fagos resultó en un perfil de tolerancia a las mutaciones muy similar para los 3 AcM. Igualmente, el estudio de pérdida y ganancia de reconocimiento con las variantes de la reEGFR producidas en células de mamíferos, corroboró que los nuevos anticuerpos conservaron la especificidad del nimotuzumab (figura 3).

Fig. 3 Mapeo epitópico mediante mutagénesis de la re-EGFR producida en células humanas HEK293-6E. La reactividad de las variantes mutadas de la reEGFR humana y de ratón (B) fusionadas al péptido etiqueta de 6 His, se evaluó por ELISA sobre placas recubiertas con nimotuzumab y cetuximab. Las proteínas unidas se detectaron con un AcM de ratón anti-6xHis y un anticuerpo anti-Fc de ratón conjugado a HRP. La reactividad del antígeno humano no mutado se utilizó como referencia para calcular la reactividad relativa (%) de las proteínas mutadas.

Fig. 4 Representación esquemática de la estrategia de evolución dirigida del paratopo del nimotuzumab. El primer paso fue la presentación exitosa sobre fagos filamentosos del paratopo (Fab) del nimotuzumab, el cual sirvió de molde para la construcción de bibliotecas donde se aleatorizaron los residuos de los CDR.
Los incrementos de afinidad se tradujeron en una mayor capacidad inhibitoria de los AcMs K4 y K5 sobre la señalización del EGFR.
Mediante experimentos de western blot se evaluó la capacidad de los AcM anti-EGFR de inhibir la fosforilación del EGFR en líneas con alta (MDA-MB-468) y moderada expresión del EGFR (H125). El incremento modesto en la afinidad de los AcM K4 y K5 (3 veces y 3,6 veces en comparación con el nimotuzumab, respectivamente) se tradujo en una mayor capacidad de inhibición de la fosforilación del EGFR mediada por el ligando en comparación con su anticuerpo predecesor (figura 5). Este efecto fue más evidente en la línea celular H125, donde redujeron la señalización en más del 80 % para todas las concentraciones evaluadas. Es interesante que la poca diferencia de afinidad entre los AcM K4 y K5 (1,2 veces) resultó en una actividad biológica distinguible, lo cual pudiera sugerir que el efecto de los AcM anti-EGFR sobre la cascada EGF/EGFR es muy sensible a pequeños cambios en su afinidad.

Fig. 5 Inhibición de la fosforilación del EGFR mediada por anticuerpos. Las células MDA-MB-468 y H125 se cultivaron durante 16 h en medio sin suero y luego se incubaron en ausencia o presencia de los anticuerpos a diferentes concentraciones. Las células se incubaron con EGF humano (100 ng/mL) durante 10 min. Las células no estimuladas se utilizaron como control negativo. Los lisados celulares se analizaron por western blot con anticuerpos específicos para la forma fosforilada del EGFR y para el receptor total. Para cada línea celular se muestra uno de los 2 experimentos independientes realizados.
Luego de 3 rondas de captura, recobrado y amplificación de los fagos positivos por la reEGFR, se seleccionaron los que presentaban mayor reactividad y se secuenció su material genético. Se combinaron las mutaciones seleccionadas en 2 moléculas (K4 y K5). Las construcciones genéticas se utilizaron para producir los nuevos AcM recombinantes en células de mamíferos. Mediante digestión con papaína se obtuvieron los fragmentos monovalentes Fab, ideales para los experimentos de medición de afinidad por BIAcore.
DISCUSIÓN
Mediante la combinación de la tecnología de presentación sobre fagos filamentosos y una estrategia de mutagénesis combinatoria y sitiodirigida16,30,31 se caracterizó por primera vez la interfaz de unión nimotuzumab-EGFR. La principal ventaja, respecto a otras estrategias de mapeo de anticuerpos anti-EGFR radica en el elevado número de variantes evaluadas durante la exploración del epitopo (162) y del paratopo (115), lo que condujo a resultados muy confiables. La estrategia de aleatorización empleada, donde se sustituyeron de manera individual los residuos a explorar por una mezcla de los 20 aa, permitió identificar los residuos energéticamente críticos en la interacción antígeno-anticuerpo, y determinar las características físico-químicas esenciales para la unión en cada posición. En otros estudios de mapeo epitópico de anticuerpos anti-EGFR se han asignado roles relevantes a los residuos con solo evaluar una sustitución.8,32-34 La rapidez con la que se generaron y evaluaron todas las variantes del DomIII se debió la presentación exitosa del DomIII sobre fagos, una contribución tecnológica del presente trabajo, pues hasta el momento no existían informes en la literatura de la presentación exitosa del EGFR o de alguno de sus dominios sobre fagos filamentosos. El campo del mapeo funcional de epitopos reconocidos por anticuerpos anti-EGFR estaba dominado por la presentación del EGFR o sus dominios sobre levaduras29,35 y células de mamíferos4 o la producción como proteínas solubles,8,32-34 tecnologías más lentas para el tamizaje de variantes.
La identidad del epitopo funcional determinado para el nimotuzumab está en concordancia con otros datos experimentales El alto grado de solapamiento entre los sitios de unión del nimotuzumab, del EGF27 y del TGF-α28 al Dom III, condición suficiente para impedir que el receptor adopte la conformación activa en presencia de los ligandos,36 explica el efecto antiproliferativo, proapoptótico y antiangiogénico del nimotuzumab.37,38 Además, el hecho de que el epitopo se localice hacia el extremo N-terminal del Dom III de la reEGFR, sugiere que la unión del nimotuzumab es incompatible con la conformación activa del receptor, por lo que bloquea la dimerización basal e inhibe la activación independiente del ligando.39 La identificación del epitopo funcional del nimotuzumab, más allá de explicar a nivel molecular sus efectos biológicos, reveló que su especificidad epitópica es única, comparado con los AcM anti-EGFR posicionados en el mercado (cetuximab, panitumumab y necitumumab). Este conocimiento completa el mapa de interacciones entre la reEGFR y los AcM terapéuticos. La información obtenida en el presente estudio abre la posibilidad de predecir el efecto de mutaciones en la región del Dom III que pudieran aparecer en los tumores, y escoger el tratamiento adecuado en dependencia del perfil de tolerancia de cada anticuerpo a las mutaciones. Adicionalmente, la información genética de la colección de variantes del DomIII generadas puede almacenarse por tiempo ilimitado, para el estudio de otros anticuerpos anti-EGFR.
La caracterización detallada de la interfaz de unión nimotuzumab-EGFR, permitió modificar el paratopo, proceso que culminó con 2 variantes del nimotuzumab con mayor afinidad que conservaron su especificidad epitópica fina. A pesar de que los incrementos de afinidad en el presente estudio fueron moderados, existieron 2 razones para continuar con estas variantes y no buscar incrementos adicionales en la afinidad. La primera fue que los incrementos modestos de afinidad se tradujeron en una mayor capacidad de las variantes mejoradas (AcMs K4 y K5) de inhibir la fosforilación del EGFR mediada por el ligando EGF, respecto al nimotuzumab, principalmente en las células con niveles moderados de expresión del EGFR. Este último es un escenario donde el nimotuzumab se encuentra en desventaja, probablemente porque requiere de una alta densidad del antígeno para una unión efectiva debido a su afinidad intermedia.40 De este modo las nuevas variantes serían una alternativa de tratamiento prometedora en situaciones donde el beneficio del nimotuzumab es limitado producto de la baja expresión del EGFR en los tumores. Adicionalmente, las evidencias preliminares provenientes de estudios imagenológicos experimentales (datos no publicados), sugieren que las diferencias de afinidad se traducen en una mayor acumulación de los nuevos anticuerpos marcados en los tejidos tumorales, por lo que podrían tener mejores propiedades para la detección in vivo de tumores. Esta tecnología abre las puertas a una amplia gama de aplicaciones como la selección de los candidatos adecuados para la terapia, el monitoreo de la respuesta y el desarrollo de resistencia a la inmunoterapia, así como en el campo de los teranósticos al fusionar los AcMs o las regiones variables de los mismos a radionúclidos con efecto terapéutico.41
La segunda razón para continuar con los nuevos AcM sin realizar optimizaciones adicionales del sitio de unión, es que los valores de afinidad muy altos no equivalen necesariamente a un mejor resultado clínico. De hecho, de los anticuerpos anti-EGFR que se encuentran en la clínica, el nimotuzumab (el de menor afinidad) es el que presenta un mejor perfil de seguridad,12 que permite su administración continuada.14 Este efecto favorable de la terapia con nimotuzumab se ha atribuido a su afinidad intermedia, que puede resultar en un adecuado balance entre la potencia antitumoral y la farmacodinámica.13 Según las predicciones por modelación matemática, existe la ventana de afinidad óptima para los anticuerpos anti-EGFR (valores de KD entre 10-8 y 10-9), donde los anticuerpos se localizarían preferentemente en el tumor mientras que la retención en los tejidos normales (baja expresión del EGFR) sería muy baja. Los nuevos AcM (K4 y K5) desarrollados en el presente trabajo, exhiben valores de afinidad que permanecen dentro del rango teórico ideal. Por tanto, explorar la posible utilidad de los AcM K4 y K5 merece especial atención. La búsqueda de anticuerpos con afinidad intermedia entre el nimotuzumab y el cetuximab condujo recientemente a la obtención del AcM Ame55, el cual presenta menor toxicidad y mayor eficacia que el cetuximab.42 Sin embargo, las diferencias en la especificad epitópica del cetuximab y el AcM Ame55 impiden establecer una relación directa entre la afinidad y las propiedades funcionales. Los AcM K4 y K5, que conservan la especificidad fina del nimotuzumab, serían herramientas útiles para estudiar en detalle la contribución de la afinidad al efecto anti-tumoral y a la toxicidad.
Conclusiones
El nimotuzumab reconoce un epitopo único respecto a otros anticuerpos aprobados en la clínica, y muestra una sensibilidad diferencial a las mutaciones del receptor. Las variantes del nimotuzumab de mayor afinidad K4 y K5 brindan alternativas adicionales para el diagnóstico y tratamiento de tumores, además de herramientas útiles para estudiar la vinculación entre la afinidad y los efectos biológicos del anticuerpo.














