Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.58 n.3 Ciudad de la Habana sep.-dic. 2006
Instituto de Medicina Tropical Pedro Kourí
Instituto de Medicina Tropical, Amberes, Bélgica
Centro de Investigación y Desarrollo de Medicamentos
PCR-RFLP/Hsp70 para identificar y tipificar Leishmania de la región neotropical
Lic. Ana Margarita Montalvo,1 Lic. Jorge Fraga,2 Lic. Jaqueline Aylema Romero,3 Lic. Lianet Monzote,4 Ing. Ivon Montano5 y Dr. Jean Claude Dujardin6
Resumen
Se realizó la estandarización de las condiciones de amplificación del gen que codifica para la proteína de choque térmico de 70 kDa (Hsp70) de Leishmania mediante la reacción en cadena de la polimerasa (PCR-Hsp70), así como el análisis posterior de la longitud de los fragmentos de restricción (RFLP) del producto amplificado, utilizando como molde ADN puro de una cepa de referencia de Leishmania mexicana. Se estudió la sensibilidad y especificidad analíticas de la PCR, así como la reproducibilidad, utilizando ADN de L. mexicana, L. amazonensis, L. guyanensis y L. lainsoni. Se obtuvo una banda de 1,3 Kpb, demostrándose la amplificación del gen que codifica para la Hsp70. Los patrones de bandas obtenidos tras la digestión enzimática, utilizando la enzima Hae III, permitieron establecer diferencias entre las especies estudiadas: L. guyanensis y L. lainsoni se diferencian entre sí y estas a su vez de L. mexicana y L. amazonensis, que mostraron un patrón de bandas común. La sensibilidad y especificidad analíticas de la técnica fueron adecuadas. Se demostró la factibilidad de identificar y tipificar especies del continente americano mediante la PCR-RFLP/Hsp70, y de utilizar la restricción enzimática del producto amplificado para distinguir entre Leishmania spp. y Trypanosoma cruzi, dándose un primer paso en el establecimiento de estos métodos moleculares en el laboratorio de referencia del instituto.
Palabras clave: PCR-RFLP, Leishmania, Hsp70, identificación, optimización.
El género Leishmania comprende protozoos parásitos pertenecientes a la Familia Trypanosomatidae, hemoflagelados y diploides, que son responsables de un conjunto de enfermedades conocidas como leishmaniosis.1 Diferentes formas clínicas de esta parasitosis pueden presentarse, en estrecha relación con la especie de Leishmania involucrada y la respuesta que resulta del equilibrio entre la inmunidad celular y humoral.2
La importante diversidad fenotípica de Leishmania ha ocasionado una compleja taxonomía con más de 20 especies descritas,3 la mayoría en América Latina. Dada la complejidad epidemiológica de algunas de sus regiones, con una distribución muy heterogénea de parásitos, los ciclos de transmisión de las diferentes especies pueden superponerse y varias especies pueden encontrarse en un mismo foco.4 Además, la intensa migración humana, así como el turismo, pueden llevar a la dispersión de Leishmania más allá de su distribución ecológica tradicional,5 todo lo cual justifica la necesidad de tipificar las especies infectantes.
Diversos métodos de caracterización se han aplicado al estudio de este género, como la electroforesis de isoenzimas,6 el análisis del ADN del kinetoplasto (ADNk),7 la técnica de ADN polimórfico amplificado al azar (RAPD)8,9 y el cariotipaje molecular.10 En los últimos años, la reacción en cadena de la polimerasa, seguida de la restricción del producto amplificado (PCR-RFLP) se ha erigido en una importante herramienta, no solo para la identificación, sino para la caracterización de especies de este género, en distintas zonas geográficas.11-13
Recientemente, García y otros14 reportaron la utilidad de la amplificación y posterior digestión del producto del gen que codifica para la proteína de choque térmico de 70 kD de Leishmania (Hsp70), para identificar varias especies del Nuevo Mundo, demostrando su aplicabilidad al estudio de muestras clínicas.
Tomando en cuenta estos antecedentes, los autores de este trabajo se propusieron optimizar en las condiciones cubanas, la PCR-RFLP/Hsp70, utilizando ADN puro de una cepa de referencia de Leishmania mexicana, así como estudiar la sensibilidad y especificidad analíticas de la técnica y su reproducibilidad, utilizando ADN de otras 3 especies de la región latinoamericana, con el fin de verificar su utilidad en nuestras condiciones, como paso fundamental en la implementación de técnicas moleculares útiles para la tipificación de especies de este parásito.
Métodos
Cepas: en el estudio se utilizaron las cepas de referencia de Leishmania que aparecen en la tabla 1.
Tabla 1 Cepas de referencia de Leishmania utilizadas en el estudio
| No. | Especie | Código de la cepa | Origen |
| 1 | Leishmania mexicana | MNYC/BZ/62/M379 | Brasil |
| 2 | Leishmania amazonensis | MHOM/BR/72/0016 | Brasil |
| 3 | Leishmania guyanensis | M4147 | Perú |
| 4 | Leishmania lainsoni | MHOM/BO/94/CUM88 | Bolivia |
Los parásitos fueron mantenidos en medio de Schneider (SIGMA, EE. UU.), suplementados con 10 % de suero fetal bovino y una mezcla de ampicilina y estreptomicina a 250 μg/mL y 100 UI, respectivamente, a 26 ºC de temperatura.
Obtención del ADN genómico: el ADN genómico se obtuvo de parásitos en su fase estacionaria de crecimiento. Se realizaron sucesivas extracciones con fenol/cloroformo como se ha descrito previamente.15 El sedimento de ADN se resuspendió en agua bidestilada estéril. El ARN presente en la muestra se digirió con la enzima ARNasa H (10 µg/mL) (Boehringer Mannheim, Alemania), incubándose durante 1 h a 37 ºC. La calidad del ADN obtenido se verificó por electroforesis en gel de agarosa 0,8 % en tampón de corrida tris-borato-EDTA 0,5X (TBE) (Tris borato 0,45M; EDTA 0,01 M; 0,5 X) con bromuro de etidio (0,5 mg/mL). También se utilizó ADN de Trypanosoma cruzi, Plasmodium falciparum, Toxoplasma gondii y Pseudomonas aeruginosa, donado por el Laboratorio de Biología Molecular del Departamento de Parasitología del Instituto de Medicina Tropical Pedro Kourí (IPK). Se realizó la lectura de la absorbancia a 260 y 280 nm de cada ADN obtenido. La relación entre ambas lecturas se empleó para estimar la pureza de la preparación.
Cebadores: los cebadores utilizados amplifican una región de 1,3 Kpb del gen que codifica la proteína Hsp70 reportados por García y otros14 y sintetizados por SIGMA Genosys (Inglaterra):
Hsp70 sense (5' GACGGTGCCTGCCTACTTCAA 3) y Hsp 70 antisense (5' CCGCCCATGCTCTGGTACATC 3)
Estandarización de la reacción en cadena de la polimerasa: para realizar la optimización se utilizó la cepa de L. mexicana MNYC/BZ/62/M379.
La mezcla de la PCR se realizó en un volumen total de 50 µL, que contenía 100 ng de ADN de L. mexicana como molde, 1x del tampón de amplificación de la enzima (tris-HCl 50 mM pH= 8,0; NaCl 100 mM; 0,1 mM EDTA; 1 mM DTT; glicerol 50 % y Tritón X-100 1 %) (Promega, EE. UU.) y 200 µM de cada dinucleótido trifosfato (Promega, EE. UU.). Además, fueron evaluadas las cantidades óptimas de: MgCl2: 1,5; 2,0; 2,5 y 3 mM, Taq ADN polimerasa (Promega, EE. UU.): 1,0; 1,5; 2,0 y 2,5 U, Cebadores: 0,2; 0,4; 0,6 y 0,8 µM. Se realizó un control negativo en todos los experimentos, que contenía todos los componentes de la mezcla de reacción, excepto ADN molde.
La reacción de amplificación se realizó en un termociclador (MJ Research PTC, EE. UU.), con el programa siguiente: desnaturalización inicial 94 ºC durante 5 min, seguida de: desnaturalización (94 ºC, 30 s), hibridación (61 ºC, 1 min) y extensión (72 ºC, 3 min) por 33 ciclos, seguido de una extensión final a 72 ºC por 10 min.
Los resultados fueron verificados mediante electroforesis en gel de agarosa 2 % en las condiciones ya descritas con anterioridad.
Reproducibilidad: la reproducibilidad se comprobó realizando la PCR en 2 d diferentes, utilizando ADN de 4 especies: L. mexicana, L .amazonensis, L. guyanensis y L. lainsoni. Los resultados fueron comprobados en corridas electroforéticas en gel de agarosa 2 %.
Sensibilidad analítica: este experimento permitió conocer, tanto la concentración de ADN molde que resulta útil en la reacción de amplificación, como su nivel de detección. Se realizaron diluciones dobles seriadas desde 140 ng y hasta 8 fentogramos/10 µL, utilizando ADN de L. mexicana y agua bidestilada estéril. Posteriormente, se realizó la PCR siguiendo las condiciones establecidas, en el mismo volumen final, y los productos de amplificación de algunas de las diluciones realizadas fueron observados en una corrida electroforética.
Especificidad analítica: para estudiar la especificidad de la PCR, se utilizó como molde 100 ng de ADN de Trypanosoma cruzi, como especie relacionada, y de Plasmodium falciparum, Toxoplasma gondii y Pseudomonas aeruginosa como especies no relacionadas. Se realizó la reacción de amplificación de la Hsp70 en las condiciones previamente estandarizadas para todos los casos. El resultado se visualizó en una corrida electroforética en gel de agarosa 2 % en las condiciones previamente descritas.
Precipitación del producto de PCR Hsp70. Los productos obtenidos en las diferentes reacciones de PCR con ADN de Leishmania spp. fueron precipitados. Para ello, se añadieron en cada caso 2 volúmenes de agua, 1/10 de NaAc 3M pH=5,4 y 2 volúmenes de etanol absoluto frío. Se mezcló y dejó reposar a - 20 ºC durante toda la noche. Posteriormente, se realizó una centrifugación a 1 000 g durante 15 min, se desechó el sobrenadante y se lavó con etanol 80 %, en iguales condiciones. El precipitado de ADN se dejó secar completamente y se resuspendió en 20 µL de agua bidestilada estéril. La calidad de la precipitación se chequeó a través de una electroforesis en gel de agarosa 2 %, en las condiciones de corrida ya descritas.
Digestión del producto de PCR (PCR-RFLP): para la restricción de los productos de PCR se utilizó la enzima Hae III (Promega, EE. UU.) de acuerdo con las instrucciones del fabricante. Las condiciones de digestión fueron estandarizadas, empleando diferentes concentraciones de enzima (1,5 y 2,0 U) y cantidades de producto de PCR precipitado (2, 3 y 5 µL). La mezcla se colocó a 37 ºC en baño termostatado toda la noche. A continuación, se realizó una corrida electroforética de todo el producto digerido en gel de agarosa de alta resolución 3 % (Promega, EE. UU.) a 50 V y en las condiciones de corrida ya descritas, con el fin de identificar los perfiles de bandas correspondientes a cada especie estudiada. Se realizó la comparación de los patrones, analizando las bandas presentes y ausentes en cada especie, tanto a través de la observación de la corrida electroforética, como de un diagrama en que se reprodujeron las bandas aparecidas en esta.
Resultados
En general, se define como óptima la concentración o cantidad del componente de la reacción que da lugar a bandas nítidas e intensas, a la menor concentración posible. Teniendo esto en cuenta, se reflejan en la tabla 2 las condiciones óptimas establecidas para cada parámetro estudiado, que pueden observarse individualmente en la figura 1 (A, B y C).
Tabla 2 Parámetros optimizados en la PCR-Hsp70
| Parámetro estudiado | Concentración óptima |
| Concentración de cebadores | 0,6 μM |
| Cantidad de enzima Taq ADN polimerasa | 1 U |
| Concentración de MgCl2 | 1,5 mM |
1A: Líneas 1, 2, 3, 4: concentraciones de 0,2; 0,4; 0,6; 0,8 µM de cebadores
1B: Líneas 1, 2, 3, 4: concentraciones de 1,5; 2; 2,5; 3 mM de MgCl2.
1C: Líneas 1, 2, 3, 4: cantidades de 1; 1,5; 2; 2,5U de Taq polimerasa MM marcador molecular, CN control negativo.
Fig. 1. Optimización de distintos parámetros de la PCR Hsp70 utilizando ADN de L. mexicana.
El nivel de detección alcanzado en el estudio de la sensibilidad analítica fue de 132 fentogramos, porque en diluciones posteriores no se observaron bandas en la corrida electroforética realizada (resultados no mostrados).
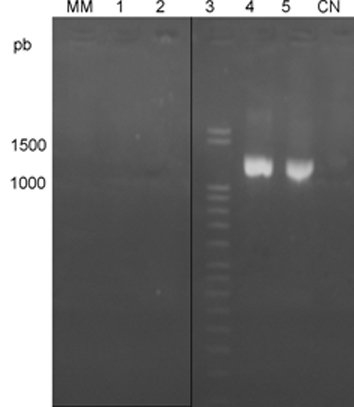
Una vez optimizadas las condiciones, los resultados fueron reproducibles cuando se realizó la PCR en 2 d diferentes y con ADN de las 4 especies (L. mexicana, L. amazonensis, L. guyanensis, L. lainsoni) (fig. 2). En todos los casos fueron observables productos de amplificación de la talla esperada: 1,3 Kpb.
Líneas 1 y 2: L. mexicana, Líneas 3 y 4: L. guyanensis Líneas 5 y 6: L. lainsoni Líneas 7 y 8 L. amazonensis. MM: marcador molecular
Fig. 2. Electroforesis en gel de agarosa de los productos de amplificación obtenidos al realizar la PCR en 2 d diferentes, utilizando ADN molde de 3 cepas.
En relación con la especificidad, se obtuvo producto de amplificación cuando se utilizó ADN molde de Leishmania spp. y de T. cruzi (fig. 3). Sin embargo, a pesar de que los otros protozoos evaluados también poseen el gen que codifica para la Hsp70, no se obtuvo amplificación cuando se utilizaron estos cebadores, así como tampoco con el ADN bacteriano.
MM: marcador molecular, Línea 1: L. mexicana, Línea 2: T. cruzi, Línea 3: P. falciparum, Línea 4: T. gondii, Línea 5: P. aeruginosa CN: control negativo
Fig. 3. Corrida electroforética de los productos de amplificación obtenidos utilizando diferentes ADN.
Los diferentes productos de PCR precipitados, fueron digeridos con la enzima HaeIII. De manera general, cantidades inferiores a los 5 µL de producto de PCR precipitado no fueron suficientes para lograr una digestión efectiva. En cuanto a la enzima, 1,5 U fue suficiente para obtener una buena digestión, por lo que concentraciones inferiores y superiores fueron desestimadas (datos no mostrados).
Los diferentes patrones de bandas obtenidos en la corrida electroforética, tras la restricción del producto amplificado y digerido con 1,5 U de enzima (fig. 4A), posibilitan la diferenciación entre algunas especies: L. mexicana y L. amazonensis produjeron un patrón de bandas idéntico, bien diferenciable del obtenido para L. guyanensis y L. lainsoni, individualmente. Así mismo, dado que se produjo amplificación de la Hsp70 con ADN de T. cruzi, se realizó la digestión del producto amplificado en las mismas condiciones, demostrándose que es perfectamente posible identificar ambos géneros de la familia Trypanosomatidae, de acuerdo con el patrón de bandas, que en el caso de T. cruzi resultó muy diferente al de cualquiera de los perfiles obtenidos para las especies de Leishmania estudiadas. Para facilitar la comprensión del análisis de las bandas, se muestra la representación esquemática de la corrida electroforética realizada (fig. 4B) donde puede corroborarse lo indicado.
Línea 1: L. amazonensis, Línea 2: L. mexicana, Línea 3: L. guyanensis, Línea 4: L. lainsoni, Línea 5: T. cruzi. MM marcador molecular
Fig. 4. Corrida electroforética de la digestión Hae III de distintos productos de PCR Hsp70. A. Electroforesis en gel de agarosa 3 %. B. Diagrama en que se reproducen los fragmentos apreciados en la corrida electroforética.
Discusión
En la leishmaniosis tegumentaria americana (LTA), que agrupa todas las variantes clínicas de la enfermedad cutánea que pueden presentarse en América Latina, la tipificación temprana y segura de los parásitos puede ser de importancia crítica para monitorear la evolución clínica, adecuar el tratamiento y evaluar los riesgos epidemiológicos. Lo anterior se apoya en el hecho de que síntomas similares pueden ser causados por otros patógenos, varias especies de Leishmania pueden ocasionar la misma presentación clínica16 y diferentes especies pueden requerir distintos tratamientos.17
De otra parte, un principio fundamental para el uso de la PCR continúa siendo su optimización, con el fin de garantizar una amplificación exitosa, ya sea cuando se establece por primera vez en un laboratorio, cuando se utilizan por primera vez un juego de cebadores, o incluso un tipo diferente de enzima polimerisadora.18
Los cebadores utilizados para amplificar el gen deseado cumplen los requisitos generales que determinan el éxito de la reacción19 y la calidad en la síntesis de estos ha sido demostrada con los resultados expuestos. En relación con su concentración, 0,4 µM (20 pmol/50 µL reacción) de cebadores fue adecuado para desarrollar la reacción de amplificación de la Hsp 70. Concentraciones superiores ensayadas produjeron bandas más intensas y concentraciones más bajas estarían en el límite recomendado para su uso, y favorecerían su posible agotamiento durante la reacción, razones que determinaron esta selección.
En el ensayo, la concentración óptima de MgCl2 resultó ser 1,5 mM. Concentraciones superiores produjeron amplificaciones igualmente nítidas a la observación electroforética, por lo que se escogió la menor concentración que produjera bandas definidas y reproducibles.
Respecto a la enzima Taq ADN polimerasa, la cantidad resulta esencial para la obtención de un producto de amplificación adecuado. Se considera óptimo un rango entre 0,5 y 2,5 U/50µL de reacción de acuerdo con las características de la enzima utilizada. Cantidades superiores conllevan a disminuir su especificidad.19 La utilización de 1 U de enzima de calidad Eppendorf en cada reacción, fue seleccionada como óptima, cuando se mantuvieron el resto de los parámetros constantes; a concentraciones superiores se obtuvieron bandas de igual intensidad, por lo que se escogió la menor cantidad de enzima con la que se obtuvo una banda nítida.
El análisis de la longitud de los fragmentos de restricción, desde su utilización por Cupolillo y otros,20 ha demostrado ser una herramienta prometedora para estudiar la diversidad molecular de especies de Leishmania.12,13,16 En este trabajo, se obtuvieron bandas de amplificación al realizar la PCR utilizando ADN de Leishmania spp. y T. cruzi. Como ha sido señalado, las proteínas de choque térmico están ampliamente distribuidas en la naturaleza21 y son muy heterogéneas,22 lo que ha servido de base para la caracterización interespecífica.
Sin embargo, tal como se reporta en la literatura, los cebadores utilizados posibilitan la amplificación a partir del ADN de T. cruzi, aspecto que puede ser resuelto al realizar la digestión posterior del producto (PCR-RFLP), porque como se demostró, ofrece patrones de bandas bien distinguibles entre ambas especies. Conociendo que a la par coinciden en su distribución geográfica numerosas especies de Leishmania spp. y T. cruzi,23 esta técnica podría demostrar las variaciones que existen entre los genes de ambos parásitos, y sería posible identificar, de forma paralela, infecciones por T. cruzi, que no son siempre clínicamente evidentes.24
Se debe señalar que entre las especies de Leishmania, no todas tienen la misma importancia epidemiológica ni clínica, por lo cual la imposibilidad de distinguir entre ellas no puede ser un análisis rutinario ni circunscrito solo al aspecto molecular.
En este trabajo, se pudo distinguir perfectamente entre L. guyanensis y L. lainsoni, con patrones de bandas idénticos a los reportados con anterioridad,14 y estas a su vez se diferenciaron de L. mexicana y L. amazonensis, que mostraron patrones comunes tras la digestión.
En relación con estas últimas, se puede señalar que García y otros14 obtuvieron un patrón de bandas tras la digestión con Hae III idéntico al encontrado por nosotros al realizar la PCR-RFLP/Hsp70 con ADN de L. amazonensis, pero no fue analizada la especie L. mexicana en aquel trabajo.
L. mexicana es responsable de una lesión generalmente única de evolución benigna, mientras L. amazonensis también se presenta como lesión única o múltiple, pero es reticente a la curación y tiene alta tendencia a evolucionar a la forma difusa;3 aunque también ha sido aislada en casos de leishmaniosis mucocutánea y visceral y su distribución geográfica es mucho más limitada.25
Estas características pudieran ayudar a su diferenciación, aunque el empleo combinado de varias enzimas de restricción pudieran facilitar esta, como ha ocurrido en el estudio de otras especies;16 así como el estudio adicional de otros marcadores entre ambas o incluso la combinación con otras herramientas, en dependencia de factores que abarcan desde la disponibilidad de enzimas hasta la posibilidad de contar con el aislamiento en cultivo.
Futuros estudios permitirán obtener perfiles que caractericen un mayor número de especies y sentar las bases para futuras aplicaciones en el contexto humano, por lo que se recomienda aplicar estas condiciones al estudio de otras especies del parásito circulantes en nuestra área geográfica, que puedan aportar información para un mejor control clínico y epidemiológico de los casos de leishmaniosis neotropical.
Agradecimientos
Esta investigación ha sido realizada con el apoyo financiero del Programa Belga de Cooperación a través del Proyecto Marco de fortalecimiento de la investigación: Institutional Collaboration between ITM Antwerp and INHEM - IPK- CUBA.
PCR-RFLP/Hsp70 for identification and tipification of Leishmania from the tropical region
Summary
The optimization of the PCR conditions for amplification of the gene coding for the 70kDa (HSp70) heat shock protein as well as the analysis of the restriction fragment length polymorphism (RFLP) were carried out. DNA from a reference strain of Leishmania mexicana was used as template. Analytical sensitivity and specificity, and reproducibility of PCR using DNA from L. mexicana, L.amazonensis, L. guyanensis and L. lainsoni were determined. A 1.3 kp band was obtained, which confirmed gene amplification. The band patterns derived from Haelll enzyme digestion allowed differentiating several species. L. guyanensis and L. lainsoni were different from each other, while L. mexicana and L. amazonensis, which shared a common pattern, were different from the other two species. Analytical sensitivity and specificity were adequate. The enzymatic restriction of the PCR product made it possible to differentiate Leishmania spp. from T. cruzi. The feasibility of identifying and typifying species from the American continent through PCR-RFLP/Hsp70 and of using enzymatic restriction of amplified product to distinguish Leishmania spp. from Trypanosoma cruzi was shown. This was the first step in implementing these molecular methods in the reference laboratory of the Institute.
Key words: PCR-RFLP, Leishmania, Hsp70, identification, optimization.
Referencias bibliográficas
1. WHO. Control of the Leishmaniasis. Report of a WHO Expert Committee,
2. Alvar-Ezquerra J, Moretti ML, Corachán-Cuyás M. Infecciones causadas por protozoos flagelados hemotisulares. En: Farreras P, Rozman C, eds. Medicina Interna. 14ª ed. Madrid: Ediciones Harcourt; 2000. p.2749-57.
3. Lainson R, Shaw JJ. Evolution, classification and geographical distribution. En: Peters W; Kendrick R, eds. The Leishmaniasis in Biology and Medicine.
4. Lucas CM, Franke ED, Cachay MI, Tejada A, Cruz ME, Kreuzer RD, et al. Geographic distribution and clinical description of leishmaniasis cases in
5. Victoir K, Bañuls J, De Doncker S, Cabrera L, Alvarez E, Arévalo J, et al. Direct Identification of Leishmania species in biopsies from patients with american tegumentary leishmaniasis. Trans R Soc Trop Med Hyg 2003;97:80-7.
6. Chance ML,
7. Gramiccia M, Smith DF, Angelici MC, Raedy PD, Gradoni L. A kinetoplast DNA probe diagnostic for L. infantum. Parasitology 1992;105:29-34.
8. Giuziani I, Dellagi K, Isamail RB. Random amplified polymorphic DNA technique for identification and differentiation of
9. Tybairenc M, Neubauer K, Barnabe C, Guerrini F, Skareckey D, Ayala FJ. Genetic characterization of six parasitic protozoa parity between random primer DNA typing and multilocus enzyme electrophoresis. Proc Nat Acad Sci USA 1993;90:1335-9.
10. Dujardin JC, Gajendran N, Arevalo J, Llanos Cuentas A, Guerra H, Gomez J, et al. Karyotype polimorphism and conserved characters in the Leishmania (Viannia) braziliensis complex explored with chromosome derived probes. Ann Soc Belge Med Trop 1993;73:101-18.
11. Marfurt J, Nasereddin A, Niederwieser I, Jaffe CL, Beck HP, Felger I. Identification and diferentiation of Leishmania species in clinical samples by PCR amplification of the miniexon sequence and subsequent restriction fragment length polymorphism analysis. J Clin Microbiol 2003;41:3147-53.
12. Quispe Tintaya KW, Xu Ying, Dedet JP, Rijal S, De Bolle X, Dujardin JC. Antigen genes for molecular epidemiology of leishmaniasis: polymorphism of Cysteine Proteinase b and surface metalloprotease Glycoprotein 63 in the Leishmania donovani Complex. J Infect Dis 2004;189:1035-43.
13. García AL, Kindt A, Quispe Tintaya WQ, Bermúdez H, Llanos A, Arévalo J, et al. American tegumentary leishmaniasis antigen gene polymorphism, taxonomy and clinical pleomorphism. Infect Genet Evol 2005;5:109-16.
14. García L, Kindt A, Bermúdez H, Llanos-Cuentas A, De Doncker S, Arévalo J, et al. Culture independent species typing of neotropical Leishmania for clinical validation of a PCR based assay targeting heat shock protein 70 genes. J Clin Microbiol 2004;42:2294-7.
15. Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning. A laboratory Manual.
16. Rotureau B, Ravel C, Couppié P, Pratlong F, Nacher M, Dedet JP, et al. Use of PCR restriction fragment length polymorphism analysis to identify the main
17. Croft SL, Yardley V, Hendrick H. Drug sensitivity of Leishmania species some unresolved problems.Trans R Soc Trop Med Hyg 2002;96:S127-9.
18. Amini B, Olyaee S, Sahabi F, Karimi M. PCR Optimization improving of human cytomegalovirus (HCMV) PCR to achieve a highly sensitivity detection method. Iranian J Biotechnol 2003;1:59-64.
19. Steffen C, Ankenbauer W, Poauer-Hofmann R, Bye S, Eeisen D, Frey B, et al. PCR Applications Manual 2nd ed. Mannheim: Roche; 1999. p.10-48.
20. Cupolillo E, Grimaldi GJr, Momen H. Intergenic region typing (IRT) a rapid molecular approach to characterization and evolution of Leishmania. Mol Biochem Parasitol 1995;73:145-55.
21. Rico AI, Angel SO, Alonso C, Requena JM. Immunostimulatory properties of the Leishmania infantum heat shock proteins Hsp70 and Hsp83. Mol Immunol 1999;36:1131-9. 22. Arora
23. Aznar C, López BP, Bradariz S, Mariette C, Liegeard P, Do Carmo DA, et al. Prevalence of anti R13 antibodies in human Trypanosoma cruzi infections. FEMS Immun Med Microbiol 1995;12:231-7.
24. Alberti E. Trypanosoma. En: Microbiología y Parasitologia Médicas. t.3. Llop A, Valdes Dapena MM, Zuazo JC, eds. Ciudad de La Habana:Ed. Ciencias Medicas; 2001. p.49-68.
25. Barral A, Pedral Sampaio D, Grimaldi GJr, Momen H, Mc Mahon Pratt D, De Jesús AR, et al. Leishmaniasis in Bahia, Brazil. Evidence that Leishmania amazonensis produces a wide spectrum of clinical disease. Am J Trop Med Hyg 1991;44:536-46.
Recibido: 22 de mayo de 2006. Aprobado: 18 de agosto de 2006.
Lic. Ana Margarita Montalvo. Instituto de Medicina Tropical Pedro Kourí, Apartado Postal 601, Marianao 13, Ciudad de La Habana, Cuba. Correo electrónico: amontalvo@ipk.sld.cu
1 Investigadora Auxiliar. Instituto de Medicina Tropical Pedro Kourí (IPK).
2 Máster en Parasitología. Investigador Agregado. IPK.
3 Reserva Científica. Centro de Investigación y Desarrollo de Medicamentos.
4 Máster en Farmacología. Aspirante a Investigadora. IPK.
5 Especialista en Parasitología. IPK.
6 Doctor en Ciencias. Profesor. Instituto de Medicina Tropical, Amberes, Bélgica.