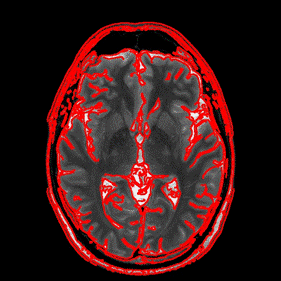
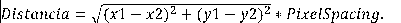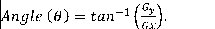Introducción
La neurociencia comprende una amplia gama de interrogantes acerca de cómo se organiza el sistema nervioso y cómo funciona para generar la conducta. Estos cuestionarios pueden explorarse por medio de las herramientas analíticas de la genética, la biología molecular y celular, la anatomía y la fisiología de los sistemas, la biología conductual y la psicología. (Purves et al., 2007)
La localización de las funciones cognitivas en el cerebro tiene una larga historia, que se remonta por lo menos a los primeros años del siglo XIX, con la fundación de la frenología. Esta ciencia constituyó una especie de “psicología de las facultades”, empeñada en atribuir características mentales específicas a las diferentes regiones del cerebro, hasta 27 distintas en esa época, en primer lugar, se enfatiza por primera vez la importancia de la superficie cerebral, el córtex. En segundo lugar, se postula que el cerebro no era un órgano unitario e indiferenciado, abriendo la puerta a un estudio más sistemático y, sobre todo, caracterizado por los medios científicos como centro de las funciones cognitivas del cuerpo.(Escera, 2004)
El cambio al siglo XX supuso la eclosión de los estudios neuroanatómicos y neurofisiológicos que fueron perfilando el surgimiento de la Neurociencia, en los años 60, como disciplina de síntesis. En la década de 1990 sucedieron los estudios citoarquitectónicos del córtex, que pusieron de manifiesto la existencia de una compleja organización anatómica, la identificación de varias decenas de áreas compuestas por células nerviosas de morfología diferenciada. Sin embargo, más adelante, algunos científicos cuestionaban que las diferencias morfológicas implicaban diferencias funcionales. A raíz de estos estudios los psicólogos cognitivos empezaban a dirigir sus miradas hacia la neurociencia y la neurociencia empezaba a interesarse más por el tipo de problemas que la psicología se planteaba. La necesidad de una nueva forma de entender las relaciones entre el cerebro y la mente se hacía palpable.(Snatiesteban y García, 2019) (Escera, 2004)
El desarrollo de las TIC`s en la actualidad han permitido profundizar en la exploración y el desarrollo de diferentes áreas de las ciencias y otros ámbitos en plena transformación brindando métodos, formas y herramientas para la aplicación de estos descubrimientos. Los programas informáticos se han convertido en una herramienta fundamental en el estudio de un órgano tan complejo como el cerebro (Escobar, 2020). Dando paso al surgimiento de la neuroinformática como uno de los campos más efervescentes y que siguen brindando nuevos descubrimientos. (Maria, 2012)
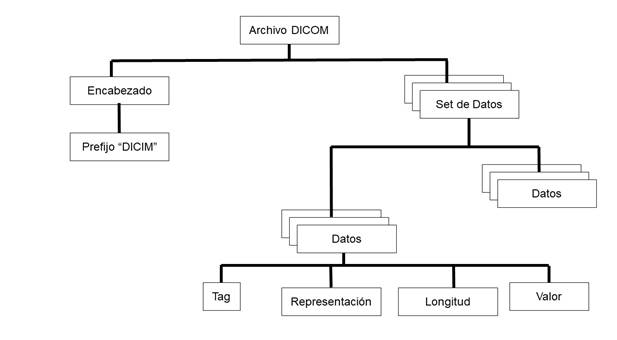
Los nuevos estudios y descubrimientos en este campo han arrojado un inmenso cúmulo de información y entre todas estas se encuentran las técnicas computacionales para obtención y manejo de datos cerebrales. Entre estos datos tienen un impacto mayor los datos de neuroimagen, obtenidos principalmente a través de Resonancias Magnéticas y Tomografías Digitales, de ahí la importancia de proponer soluciones sobre el manejo, gestión y procesamiento de datos de neurociencia. Para estos datos poder ser visualizados son convertidos a un formato estándar para su visualización, siendo el caso del formato DICOM. El formato DICOM ha sido impulsado por las organizaciones ACR y NEMA como un estándar para la transmisión de información y almacenamiento de imágenes médicas. Este estándar de archivos está formulado por numerosos elementos, cada cual compuesto por una etiqueta que lo identifica, un valor de representación que indica el tipo de dato almacenado, un campo que indica la longitud del dato y el dato propiamente dicho; este último proporciona información del paciente, del análisis realizado o de la imagen obtenida, dependiendo del valor de la etiqueta. En el archivo los elementos se escriben en orden ascendente de acuerdo al valor de la etiqueta que los identifica.(Forero, Sarmiento y Díaz, 2001)
La resonancia magnética (RM) se ha establecido como una herramienta muy valiosa en el diagnóstico e investigación de muchas áreas en la medicina gracias a su gran capacidad de proveer excelente caracterización y diferenciación de tejidos blandos de múltiples áreas del cuerpo.(Rivera, Puentes y Caballero, 2011). Por tanto, las resonancias magnéticas son unas de las técnicas más confiables de obtener imágenes cerebrales de pacientes para su posterior análisis.(Sánchez-Cedeño et al., 2023)
CNEURO es una institución cubana de investigación y desarrollo dedicado a la investigación traslacional y otras tecnologías médicas, que abarca desde la investigación básica hasta el desarrollo, producción y comercialización de tecnologías. Mostrando fuerte interés en la utilización de software nacional dedicado a la medicina. Entre las investigaciones más destacadas de esta organización se encuentran los estudios de resonancia magnética aplicado a distintos escenarios, entre ellos los diagnósticos y seguimiento de tumores cerebrales, mal formaciones congénitas y afecciones mediante factores externos como contusiones.(Cuba, 2020)
Esta entidad ejecuta investigaciones en una amplia gama de temas que incluyen neurociencia cognitiva, neuroinformática, neuroimagen funcional, análisis de señal bioeléctrica, modelación matemática, investigación neuroquímica, genética molecular e impresión 3D para dispositivos médicos.(«Empresa Centro de Neurociencias de Cuba - Empresa Centro de Neurociencias de Cuba», 2020)
Actualmente en la Universidad de las Ciencias Informáticas se encuentra en continuo desarrollo desde su creación el sistema XAVIA-PACS o sistema para el almacenamiento, transmisión y visualización de imágenes médicas como indican sus siglas. Es utilizado en algunos hospitales del país mostrando buenos resultados en su desempeño y despliegue pues cuenta con varias funcionalidades que permiten el manejo de imágenes médicas para la obtención de diagnósticos a partir de los datos visuales. Permitiendo la realización de mediciones, modelos tridimensionales, selecciones de áreas y perímetros de cualquiera de los elementos de estas imágenes. Cuenta además con una base de datos en la cual se organizan y almacenan todos los datos de los pacientes.
A pesar de los avances en el desarrollo de este sistema y su puesta en marcha no cuenta con un componente que permita realizar mediciones precisas sobre las imágenes de resonancia magnética con la finalidad de poder llevar un control de los avances de las enfermedades o cualquier otra afección que comprometa la salud de los pacientes. Para esta tarea se analizaron una serie de algoritmos localizados en las literaturas consultadas. Los autores del presente trabajo realizaron pruebas de los mismos para determinar su precisión en mediciones lineales.
Métodos o Metodología Computacional
Se realizó una investigación de los principales algoritmos aplicados para realizar las mediciones en imágenes médicas en el estándar DICOM. Para la ejecución de esta investigación se sigue una estrategia explicativa y se emplearon los siguientes métodos:
Análisis y Síntesis: análisis sobre los principales algoritmos existentes para realizar mediciones sobre imágenes médicas de resonancia magnética. Además, se estudió y analizó el estándar DICOM para poder manipular y procesar las imágenes.
Comparativo: Mediante este método se establece una comparación de los algoritmos analizados en cuanto a su funcionalidad, aplicación en la solución informática y la exactitud de los mismos sobre la imagen.
Estándar DICOM
El estándar DICOM define protocolos de comunicación de red, almacenamiento e intercambio de datos fuera de línea, protocolos semánticos para el intercambio de datos a niveles de conformidad. Estos estándares especifican protocolos de comunicación para dispositivos que reclaman conformidad. Además, una sintaxis y semántica de la información que se puede intercambiar en cada uno de sus protocolos. (Twa y Johnson, 2022)
Para introducirse en el complejo entorno de la medicina, DICOM usa un lenguaje especializado, basado en un modelo del mundo real. Se interpreta como todos los datos físicos o descriptivos como por ejemplo el nombre del paciente, el tipo de estudio, el dispositivo médico, los parámetros de la adquisición, la imagen digital, entre otros que son vistos por DICOM como elementos con sus respectivos atributos y propiedades. De esta forma se establece una jerarquía entre los datos que permite realizar una clasificación según el contenido de la información por grupos, facilitando la identificación, el acceso a las variables y los parámetros de interés dentro de un mismo archivo.(PIEDRAHITA, SERNA y LEMUS, 2010)
Por lo general un archivo DICOM es reconocible por su extensión *.dcm, sin embargo, esto no es una exigencia del Estándar, por lo que la forma de diferenciarlo es por medio de su encabezado (HEADER) que consta de 128 bytes de archivos de preámbulo y 4 bytes de prefijo “DICM”(Larobina, 2023). El preámbulo puede estar en blanco o contener información sobre la aplicación principal con la que debe ser ejecutado.(PIEDRAHITA, SERNA y LEMUS, 2010)
Cada una de las estructuras de estos archivos se pueden tratar de manera independiente mediante el apoyo de librerías como pydicom para el tratamiento de estos, enfocada principalmente a la visualización o modificación en cualquiera de las series (Clunie, 2021; Cvija y Severinski, 2021; Mantri, Taran y Sunder, 2020). Estas se pueden tratar de manera independiente lo cual propicia poder sincronizar dos estudios distintos de un mismo paciente en la misma serie, a este punto se realizan las observaciones previas y se replican en el resto de las imágenes. A partir de esta estructura resulta cómodo el trabajo con estos datos asegurando la accesibilidad a la información contenida en las series. Dado que el Centro de Neurociencias de Cuba y el sistema XAVIA PACS utiliza resonancias magnéticas y radiografías en el estándar DICOM se busca que exista la homogenización en el manejo de los mismos.
Algoritmos para la medición de imágenes médicas
Los autores consultaron bibliografía actual y variada en bases de datos referenciadas como PubMed, ISI, Frontiers, entre otras para identificar y obtener información asociada a los algoritmos necesarios para la realización de mediciones de distancia en imágenes médicas. Este análisis propició seleccionar el algoritmo más adecuado en la realización de mediciones sobre imágenes de resonancias magnéticas cerebrales en formato DICOM. Los algoritmos para mediciones de distancia en imágenes fueron la Transformada de Gough (Vázquez-Bautista, 2021), Detección de puntos de referencia (Eseng-Bilogo, 2016), Algoritmo de Canny (NIÑO NAVARRETE, 2019).
Para las siguientes comparaciones se utilizó el set de datos de imágenes medicas de resonancia magnética en formato DICOM proporcionado por CNEURO. Estas imágenes se sometieron a un tratamiento de anonimización a fin de mantener la integridad de los pacientes a los cuales pertenecen estos estudios. Las imágenes presentes en este set de datos fueron tomadas bajo las mismas medidas, condiciones y se calibraron debidamente por los expertos en radiología del Centro de Neurociencias de Cuba.
Algoritmo de detección de puntos de referencia
Este algoritmo se basa en la distancia que se encuentra entre dos puntos de una imagen. Estos puntos unidos por una recta que se encuentra en un plano de dos dimensiones, en este caso una imagen. Una imagen bidimensional se encuentra representada por un conjunto de píxeles formando una matriz de dos dimensiones. Cada una de estas dimensiones se representa en largo y ancho como los ejes de referencia X y Y en la geometría matemática.(Eseng-Bilogo, 2016). Para la realización de este cálculo de distancia en línea recta se utiliza la fórmula euclidiana.
 Fuente: Elaboración propia
Fuente: Elaboración propia Fig. 2 -Espacio Cartesiano para calcular la distancia de un vértice a otro.
Una vez despejada la ecuación se obtiene la distancia en píxeles desde un punto al otro de la imagen, pero se hace necesario conocer la distancia real fuera del eje de coordenadas. Por lo que es necesario multiplicar el resultado por la cantidad de filas de pixeles contenidas en la imagen o multiplicar por el valor de escala de la imagen.(Eseng-Bilogo, 2016)
Para las imágenes DICOM se utiliza el valor PixelSpacing contenido en los Tags que proporciona el propio archivo(Decourt y Duong, 2020; Florkow et al., 2022). Así se logra transformar la medición de unidades euclidianas a unidades métricas reales. Lo anterior se resume en la fórmula.
Al realizar el cálculo sustituyendo las variables por los valores se obtiene la distancia de un punto hasta el otro. La exactitud de esta medición depende de la manera en que se obtuvieron, trataron y calibraron los valores de la imagen.
Algoritmo de medición por la transformadora de Hough
Es un método propuesto por Hough (1962), para encontrar la ecuación de una línea que pase por un conjunto de puntos en el plano xy. Una línea que une una secuencia de puntos o pixeles puede expresarse con la ecuación de la recta:  siendo b la pendiente de esta recta. (Vázquez-Bautista, 2021).(Moncho Santonja et al., 2022)
siendo b la pendiente de esta recta. (Vázquez-Bautista, 2021).(Moncho Santonja et al., 2022)
Se puede describir de manera analítica un segmento de línea en varias formas. Sin embargo, una ecuación conveniente para describir un conjunto de líneas es la notación paramétrica o normal.
Donde ( es la longitud normal desde el origen hasta la línea y ( es el ángulo de ( con respecto al eje x.(Santini y Santini 2021)

Fig. 3 - Representación del espacio cartesiano y representación del espacio paramétrico. (Santini & Santini, 2020.)
En las figuras anteriores se muestran:
El espacio cartesiano que representa la línea físicamente visible.
El espacio paramétrico del algoritmo de Hough, en el cual la línea se comporta como una curva dependiendo del valor de los píxeles cercanos.
Al ser representadas estas líneas en forma de matriz se evidencia el camino seguido a partir de su pendiente correspondiente y el ángulo de cada uno de los pixeles seguidos. (Vázquez-Bautista, 2021). Finalizada la detección estas líneas rectas representadas en la imagen funcionan como guía para la realización de las mediciones a fin de dar mas exactitud en estas; dada la capacidad de mostrar las posibles estructuras en la imagen con estas formas geométricas(Pico-Velásquez et al., 2022).
Para llevar a la implementación el algoritmo anterior se utilizó la librería OpenCV perteneciente al lenguaje Python (Kritskiy et al., 2022; Gollapudi 2019; Sigut et al., 2020). En las figuras 4, 5, 6 y 7 se muestran cada una de las operaciones y los estados que va tomando la imagen procesada.
Aplicado el algoritmo sobre una imagen se resalta la existencia de estas líneas rectas contenidas en la matriz de píxeles(Sivkov et al., 2020). Al ser trazadas estas líneas apoyan la funcionalidad de poder trazar las mediciones sobre estas líneas estructurales a fin de mejorar la exactitud en el momento de la medición.
Algoritmo para la detección de bordes. Algoritmo de Canny
El algoritmo de detección de bordes o algoritmo de Canny se conoce como filtro de alto paso por su cualidad para detectar los límites entre dos regiones con un nivel de gris relativamente distinto de propiedades. En este los bordes se consideran son píxeles en los que la función de brillo cambia bruscamente, siendo los detectores de bordes una colección de métodos de procesamiento de imágenes locales muy importantes que se utilizan para localizar cambios nítidos en la función de intensidad.(Radhakrishnan, Panneerselvam y Nachimuthu, 2020; Peng y Zhao, 2019; Euclidiana, 2019; NIÑO NAVARRETE, 2019)
La ecuación para esta representación está argumentada por:
A partir de la fórmula del gradiente de color de cada pixel se establece el peso de los mismos y aplicando la fórmula para el ángulo de la evaluación de gradiente a fin de trazar el camino a seguir. Esto da como resultado que los bordes son discontinuidades repentinas en una imagen, que pueden surgir de la superficie normal, el color de la superficie, la profundidad, la iluminación u otras discontinuidades(Barrios y Rueda, 2012; NIÑO NAVARRETE, 2019; Bermejo, 2019). Estos bordes son importantes por dos razones fundamentales.
La mayor parte de la información semántica y de formas se puede deducir de ellos, por lo que se puede realizar el reconocimiento de objetos y analizar las perspectivas y la geometría de una imagen.
Es una representación más compacta que los píxeles. Se puede señalar dónde se producen los bordes desde el perfil de intensidad de una imagen a lo largo de una fila o columna de la imagen. Dondequiera que haya un cambio rápido en la función de intensidad indica un borde.
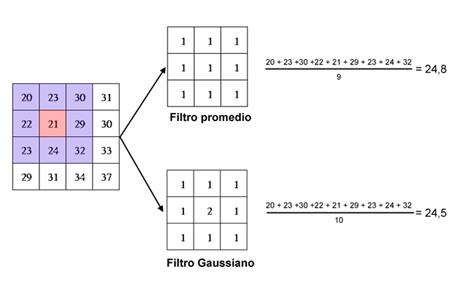
Como parte de su procesamiento típico se aplica el filtro de la siguiente manera visual.
Se le adjudica la prioridad al pixel central al cual se le hace el cálculo de gradiente para detectar si existe un cambio brusco de coloración de un pixel a otro. Utilizando la librería OpenCV se implementa y prueba el algoritmo de Canny en lenguaje python y realizar la prueba de detección en una imagen cualquiera a escalas de grises(Gollapudi, 2019; Kritskiy et al., 2022; Sigut et al., 2020). Una vez aplicados estos filtros se obtiene el siguiente resultado representado en las figuras 9, 10, 11 y 12.
Este Algoritmo realiza la detección y trazado de todos los contornos en la imagen cerebral, pero por consiguiente ignora la formación de algunas estructuras u otras formas tumorales que se asemejan a estructuras básicas del cerebro, por ende, ayuda con las mediciones, pero no tiene en cuenta la existencia de estas. Además, al existir ruido en la imagen, este no es capaz de detectar los cambios para trazar los contornos.
Comparación de los algoritmos aplicables a la solución informática
Para realizar las pruebas se seleccionaron al azar un grupo de cortes cerebrales de distintos estudios del dataset utilizado. Los cortes son de distintas áreas y regiones a fin de obtener resultados aplicables a cada zona cerebral. Todas las imágenes seleccionadas fueron sometidas a la aplicación de estos algoritmos.
Al realizar el análisis comparativo de los algoritmos utilizados para las mediciones en imágenes cerebrales los autores obtuvieron los siguientes resultados, significativos para la presente investigación.
Tabla 1 - Comparación de los algoritmos analizados. Fuente: Elaboración propia
| Detección de puntos de referencia | 10 | Permite realizar las mediciones a partir de los datos de matriz de la imagen y la variable de escala real (PixelSpacing) obtenida en los metadatos de la imagen. | 97.8% |
| Transformada de Hough | 10 | Permite la detección de estructuras lineales en las imágenes de médicas a fin de realizar mediciones más exactas. | 95.3% |
| Algoritmo de Canny | 10 | Permite detectar los contornos de cada una de las estructuras anatómicas del cerebro a partir de los bordes de cada estructura. | 85.5 % |
En la tabla anterior se aprecia la diferencia de precisión entre los algoritmos analizados. En los casos de prueba a los que se sometieron se utilizaron los mismos estudios y las mismas condiciones a fin de obtener resultados más justos. Se utilizó el software eFilmWorkstation para comparar y verificar la precisión de las mediciones realizadas con los algoritmos(«eFilm Solutions» 2023).
Resultados y discusión
Al ejecutar el análisis de los algoritmos de medición localizados en la literatura revisada se pudo determinar la precisión de cada uno de estos para la medición. Se analizó la utilidad de los mismos en cada caso de prueba aplicado. Se describe en los siguientes puntos.
La transformada de Hough es aplicable para resonancias magnéticas en distintas zonas del cuerpo pero no es aplicable de manera precisa para realizar mediciones en imágenes cerebrales. En los casos de prueba utilizados detecta muy pocas líneas en las imágenes del cerebro, por este motivo no es viable para estas mediciones.
El algoritmo de Canny, utilizado para la detección de contornos detecta áreas del cerebro en las cuales sus contornos son resaltantes, pero no detecta las demás en las cuales se difumina levemente el borde. No es aplicable pues no detecta todos los contornos al existir distintos degradados o zonas perfectamente resaltadas. Al realizar las mediciones sobre este y no detectar todos los contornos no muestra utilidad en las mediciones.
La detección de puntos de referencia para las mediciones mostró ser el algoritmo más preciso en las pruebas para las mediciones. Este utiliza para su estimación de distancia la fórmula euclidiana y la variable de escala PixelSpacing contenida en los tags de la imagen DICOM. En este caso las mediciones se realizan de manera manual y dependen de la exactitud en que se seleccionen los puntos para medir.
El algoritmo seleccionado para la solución informática es el algoritmo de detección de puntos de referencia. Al realizar las pruebas de mediciones mostró resultados similares en un 97.8 % respecto a las realizadas con el software eFilmWorkstation(«eFilm Solutions» 2023).
Conclusiones
El análisis de los algoritmos para mediciones en imágenes de resonancia magnética demostró que el mejor algoritmo aplicable es la detección de puntos de referencia basado en la formula euclidiana de distancia entre dos puntos del plano cartesiano. El data set utilizado permitió realizar pruebas con datos reales y perfectamente estandarizados del Centro de Neurociencias de Cuba, estos datos garantizaron la exactitud en las mediciones. El software eFilm fue una herramienta clave en estas comparaciones, la precisión de este software fue un criterio principal en el proceso.