INTRODUCCIÓN
A partir de diciembre del año 2019 comenzó a circular en la ciudad de Wuhan, provincia de Hubei en China el coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2). Esto marcó el inicio de la enfermedad producida por este virus (COVID-19) y el número de personas infectadas a nivel mundial aumentó de manera exponencial. Como resultado de número elevado de víctimas a causa del virus, en marzo del 2020 la Organización Mundial de la Salud (OMS) declaró el carácter pandémico de la COVID-19. 1
En Cuba los primeros casos fueron diagnosticados en marzo del 2020, a raíz de ello, se emplearon los protocolos de la OMS para la detección molecular del virus, mediante la técnica de reacción en cadena de la polimerasa en tiempo real (RT-PCR). Cabe destacar que los equipos e insumos importados para garantizar y sostener el diagnóstico del SARS-CoV-2 han sido costosos y el diagnóstico a nivel nacional de este virus ha sufrido afectaciones por la incertidumbre en las cadenas de suministros internacionales, a causa de la situación pandémica. En el primer semestre del 2020 se creó un comité de expertos con el propósito de lograr soberanía tecnológica nacional para la detección molecular del SARS-CoV-2, recurriendo a la innovación en la industria biofarmacéutica cubana. 2,3 Esta soberanía debía transitar desde la toma, colecta y procesamiento de las muestras, hasta la obtención del resultado final.
Un eslabón importante para el diagnóstico de la infección por SARS-CoV-2 han sido los medios de transporte que se han empleado para la colección, preservación y traslado de muestras de exudado nasofaríngeo (ENF) desde el sitio clínico hasta el laboratorio donde se realiza el análisis de las mismas. Varias casas comerciales han producido medios de transporte para virus, tales han sido los casos de CITOSWAB® VTM, UTM (Copan), Puritan® UniTranz-RT Universal Transport Medium (UTM) y MANTACC UTM. En el contexto de la COVID-19 y para reducir el riesgo de exposición al virus durante la manipulación y el procesamiento de los especímenes clínicos, las compañías biofarmacéuticas desarrollaron medios de transporte con capacidad virucida, basados en isotiocinato de guanidina como agente caotrópico, como el CITOSWAB® VTM-N y Copan eNAT™. (4
Para dar respuesta emergente a la contingencia de la pandemia de COVID-19 y como parte de la soberanía tecnológica, el Centro Nacional de Biopreparados de Cuba (BIOCEN) desarrolló en el 2020 un medio de transporte viral denominado BioTransVir-BTV (BTV) basado en medio esencial mínimo (MEM) y en contexto con las recomendaciones específicas de la OMS. Este fue validado por laboratorios destinados a la detección por RT-PCR del SARS-CoV-2, incluido el Laboratorio de Referencia para Virus Respiratorio del Instituto de Medicina Tropical Pedro Kouri (IPK). 3 Igualmente, se obtuvo el registro sanitario de la autoridad regulatoria nacional. Similar a otros productos comerciales de esta clase, el medio se diseñó para mantener la viabilidad viral y la estabilidad de moléculas de interés para el diagnóstico molecular en condiciones refrigeradas. Como dato adicional, en Montalvo et al. Se describió su uso en la detección de antígenos del SARS-CoV-2 con el estuche Elecsys SARS-CoV-2 Antigen de la Roche. 5 Con este mismo propósito, el medio BTV ha sido empleado en la prueba de UMELISA SARS-CoV-2 antígeno producida por el Centro Nacional de Inmunoensayo.
El BIOCEN desarrolló otros medios de transporte viral, a partir de materias primas de mayor disponibilidad comercial, denominados medio de transporte universal BTU y medio de transporte de ácidos nucleicos TAN. Este último con capacidad de inactivar virus envueltos, como el virus del Zika. 6
El objetivo de la presente investigación fue evaluar el desempeño de los medios BTU y TAN, para el diagnóstico por RT-PCR del SARS-CoV-2. Para realizar estas validaciones se tomó como referencia el medio BTV, referido con anterioridad.
MÉTODOS
Muestras
Desde junio a noviembre del 2021, un total de 113 muestras pareadas fueron colectadas con los medios en estudio y el medio de referencia (BTV). En la toma de ENF se emplearon los hisopos disponibles en la red nacional, como los producidos por CNEURO de BioCubaFarma, Copan de Huachenyang Technology y Mantacc de Miraclean Technology, ambos productos de China.
El número de especímenes según los medios en estudio fue 58 y 55, para BTU y TAN respectivamente. Al mismo tiempo, para el medio BTU se estimó en muestras positivas por RT-PCR para SARS-CoV-2 con ciclo umbral (Ct) ≤ 29 con el medio de referencia, la estabilidad del ARN viral a partir del tiempo 0 (T 0, en las primeras 12 h de colecta), a las 48 (T 48h) y 72 (T 72h) horas de colectada las muestras, todas fueron conservadas de 4-8 ºC.
En la evaluación del medio TAN fueron seleccionadas muestras positivas (Ct ≤ 29 con medio BTV) para evaluar la estabilidad del ARN viral al tiempo T 0, T 48 h, T 72 h y 168 h (T 168 h o 7 días). Para este ensayo de estabilidad las muestras en medio TAN se conservaron a temperatura ambiente (T.A) de 23 a 25 ºC. El tiempo de observación se prolongó hasta 7 días, para tratar de reproducir los hallazgos descritos por Richard Greenblatt et al. con el medio inactivante Copan eNAT™. 4) Todas las personas involucradas en el estudio fueron informadas de los objetivos de la investigación y dieron su consentimiento para que se les tomara muestras pareadas.
RT-PCR para la detección del SARS-CoV-2
El ARN de las muestras clínicas se extrajo con los estuches comerciales de QIAGEN (QIAGEN, Alemania) siguiendo estrictamente las indicaciones del fabricante y se empleó el equipo de extracción automática QIAcube.
Según la disponibilidad de pruebas de RT-PCR para la detección cualitativa del SARS-CoV-2 se utilizó la prueba STAT-NAT® COVID-19 MULTI (Sentinel Diagnostic, Italia) para evaluar el medio BTU. El estuche RIDA®GENE SARS-CoV-2 (R-Biopharm, Alemania) se empleó para evaluar el desempeño del medio TAN. Los equipos de RT-PCR Light Cycler 96 (Roche, Alemania) y BIOER (Hangzhou, China) se utilizaron para detectar la amplificación del genoma viral con STAT-NAT® COVID-19 MULTI y RIDA®GENE SARS-CoV-2 respectivamente, según las recomendaciones del fabricante. Las muestras con un valor de Ct < 37 fueron consideradas positivas para ambas pruebas comerciales.
Análisis estadístico
Se empleó el paquete estadístico GraphPad Prism V.6. para analizar los resultados. Se calculó el porcentaje de concordancia y el coeficiente Kappa de Cohen entre los resultados de la RT-PCR obtenidos con los medios en evaluación con respecto al de referencia. Según los valores de Kappa, las concordancias se definieron como pobre (0,21-0,40), moderada (0,41-0,60), sustancial (0,61-0,80) y perfecta o casi perfecta (0,81-1). 7 Para los resultados positivos se calculó la media de los valores de Ct, desviación estándar, medianas y los rangos intercuartílico (25 % y 75 % percentil). La prueba no paramétrica de rangos con signo de Wilcoxon se empleó para estimar la diferencia entre los datos pareados. Para evaluar la estabilidad de las muestras se realizó un análisis de regresión logística tomando como variable independiente el tiempo y como dependiente los valores de Ct. La significación estadística se consideró con valor de p < 0,05.
RESULTADOS Y DISCUSIÓN
Dependiendo del método diagnóstico utilizado para identificar las infecciones respiratorias por virus se emplean medios de transporte de muestras que preserven los antígenos virales o que garanticen la estabilidad del ARN o ADN viral, e incluso ambos, si se pretende cultivar el virus. Sin embargo, para realizar el diagnóstico molecular por RT-PCR se puede prescindir de la integridad de las proteínas virales. Por tanto, algunos medios de colecta de muestras que inactivan la infectividad viral han reducido el riesgo biológico y los requisitos para transportar las muestras clínicas. Además, el nivel de bioseguridad para trabajar estas muestras inactivadas ha disminuido, siendo este costoso mientras más elevado sea el riesgo.
En esta investigación el medio que mostró un mejor desempeño fue el medio TAN, seguido del medio BTU. La concordancia de los resultados obtenidos con el medio TAN con respecto al medio de referencia BTV (94,54 %) fue casi perfecta (Kappa = 0,89) para el diagnóstico molecular del SARS-CoV-2. Mientras que, el valor Kappa obtenido con el medio BTU indicó una concordancia moderada (0,58) con el medio BTV (tabla 1). Los resultados fueron divergentes con respecto al medio BTV, en un 5,46 % para el medio TAN y 20,69 % para el medio BTU.
Tabla 1 Características del desempeño de los medios transportadores BTU y TAN tomando como referencia el medio BTV
| Medios transportadores en estudio | N | Concordancia (%) | Coeficiente Kappa Cohen (interpretación) | Resultados correctamente identificados | Resultados incorrectamente identificados |
|---|---|---|---|---|---|
| BTU | 58 | 79,31 |
0,58 (moderado) |
46 | 12 |
| TAN | 55 | 94,54 |
0,89 (casi perfecto) |
52 | 3 |
No obstante, no se puede descartar que la discrepancia entre las pruebas empleadas para la detección del SARS-CoV-2 influyeran en la diferencia de la concordancia de estos medios en estudio. Con el medio BTU se empleó la prueba STAT-NAT® COVID-19 MULTI que detectó 3 fragmentos de genes virales que codificaron para la proteína de envoltura (E), de la nucleocápside (N) y la polimerasa de ARN dependiente de ARN (RdRp). Igloí et al. señaló que este ensayo no fue el más sensible al compararlo con otros para la detección del SARS-CoV-2, dentro de los cuales se incluyó el RIDA®GENE SARS-CoV-2 que detectó un fragmento del gen E y este se utilizó en la evaluación del medio TAN. 8
Aunque se identificaron diferencias en la concordancia entre los medios evaluados, las medianas de los valores de Ct de las muestras positivas correctamente identificadas por los medios evaluados BTU (19/26, 73,07 %) y TAN (27/28, 96,42 %) no mostraron diferencias significativas con respecto a las medianas de los Ct obtenidos con el medio de referencia (figura 1).
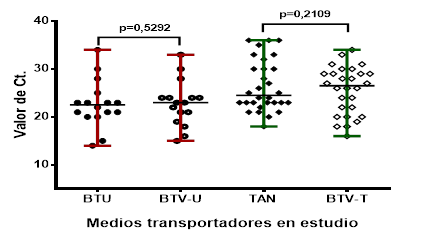
Fig. 1 Distribución de las medianas de los Ct obtenidos con el medio de referencia (BTV) y con los medios en estudio (BTU, TAN). Abreviaturas: BTV-U es la comparación con el medio BTU y BTV-T es la comparación con el medio TAN. El análisis se hizo con la prueba no paramétrica de rangos con signo de Wilcoxon para datos pareados y en la figura se muestran las medianas con los rangos intercuartílicos.
Como dato adicional, al analizar los valores de Ct obtenidos por los medios en estudio y el de referencia se demostró la funcionabilidad del método de extracción de ARN y las pruebas de RT-PCR para SARS-CoV-2 con los medios BTU y TAN. No obstante, se pudieron validar otros métodos de extracción de ARN viral a los medios evaluados, lo que pudiera incrementar las prestaciones de los mismos. Como valor añadido para el medio TAN se identificó la ausencia de signos de contaminación visual (turbidez, cambio de coloración, presencia de gas) que generalmente pueden aparecer en las muestras clínicas conservada a temperatura ambiente, debido al crecimiento de bacterias u otros microorganismos presentes en la microbiota oral. Dichos contaminantes biológicos, que usualmente se contienen con la adición de antibióticos al medio, pueden inutilizar la muestra para el diagnóstico molecular por RT-PCR.
En cuanto a la estabilidad de la positividad de las muestras colectadas con el medio BTU y conservadas de 4 a 8 ºC fue de 100 % (14/14) a las 48 h. A su vez, la positividad decreció a 85,71 % (12/14) a las 72 h lo que pudiera estar justificado por un incremento en las muestras clínicas de enzimas que degradan el ARN. 9) Para el medio TAN, la estabilidad de las muestras conservadas a temperatura ambiente se comportó al 100 % (16/16) en todos los tiempos evaluados (48 h, 72 h, 168 h) al comparar con el T 0. En un estudio similar realizado por Blow et al. los autores describieron que las muestras respiratorias estabilizadas en AVL (QIAGEN) conservaron su estabilidad por 35 días a temperaturas entre −20 a 4 °C; sin embargo, se observó degradación del ARN viral después 7 días a temperatura ambiente. 10
El análisis de regresión lineal arrojó que la tendencia a la variación de los valores de Ct en el tiempo no fue estadísticamente significativa, tanto para el medio BTU (p = 0,3382) como para el medio TAN (p = 0,4191). Además, no se constató diferencia significativa al comparar la tendencia de los Ct del medio de referencia BTV con el medio BTU (p = 0,4328) y con el medio TAN (p = 0,3482) respectivamente (figura 2).

Fig. 2 Análisis de regresión logística de los medios evaluados. En la figura se muestra el promedio y la desviación estándar en cada tiempo evaluado. El color azul corresponde al medio de referencia (BTV) y el color rojo representa el medio en evaluación. A) Se muestran los Ct del medio BTU evaluados en 3 tiempos. B) Aparecen los Ct del medio TAN evaluados en 4 tiempos. Los medios en estudio y el de referencia se evaluaron justamente en los tiempos que se muestran en la figura. En ambos paneles se obtuvieron valores de p estadísticamente no significativos (p > 0,05) al evaluar la tendencia de los valores de Ct para cada medio (BTU y TAN) y con relación al medio de referencia.
Los medios inactivantes como el medio TAN han permitido preservar las muestras clínicas varios días sin cadena de frio, lo cual ha contribuido a la fiabilidad del diagnóstico. El medio de transporte CyMol basado en alcohol con capacidad de inactivar virus ha mantenido la integridad de los especímenes colectados de muestras respiratorias hasta 21 días a temperatura ambiente. 11 En el contexto cubano, un producto de este tipo tendría ventajas, particularmente, para el traslado de muestras de sitios alejados hasta las cabeceras provinciales, donde se encuentran los laboratorios de biología molecular. Igualmente, entre las provincias en caso de interrupciones no planificadas en el trabajo de estos laboratorios.
Conclusiones
De esta manera, en este estudio se avala que ambos medios de transporte (BTU y TAN) desarrollados por BIOCEN pueden emplearse para la detección molecular (RT-PCR) del SARS-CoV-2. Esto permite contar con diagnosticadores de producción nacional, imprescindibles en la colecta de muestras clínicas para el diagnóstico virológico de la COVID-19. Se recomienda extender la evaluación del desempeño de estos medios a otros microorganismos, para ampliar las prestaciones en el diagnóstico microbiológico.














