INTRODUCCIÓN
A pesar de los avances logrados en la incorporación de nuevos antirretrovirales (ARV), así como en el acceso a los mismos, la epidemia de VIH ha continuado afectando a un elevado número de personas a nivel global. 1 Se ha propuesto que el tropismo o fenotipo viral (R5, X4, R5X4) se relacione con la patogénesis, la replicación del VIH-1, además que ha sido un elemento importante para la elección del tratamiento. 2
El VIH-1 es altamente variable y hasta el presente se han reconocido 4 grupos genéticos (M, N, O y P). Dentro del grupo M existen 9 subtipos y más de 100 formas recombinantes. 3) A pesar de la baja prevalencia de la infección por VIH-1 en Cuba circulan gran variedad de subtipos y formas recombinantes. 4
El CRF19_cpx es una forma recombinante de los subtipos D, A1 y G del VIH-1. Se detectó por primera vez en Cuba en 1999, con un origen africano. 5 Sin embargo, mientras el CRF19_cpx ha mantenido una prevalencia muy baja en el continente africano, el virus se ha propagado en Cuba, colocándolo entre los subtipos circulantes más prevalentes. 6
Un estudio anterior reveló que el CRF19_cpx causaba una progresión rápida al sida, asociado fenotipo X4, con altos niveles de RANTES y alta carga viral (CV) se sugirió por primera vez a nivel global que el CRF19_cpx podría ser un virus con mayor patogenicidad. 7
La presente propuesta ha tenido como objetivo profundizar en el origen, evolución y patogenia del CRF19_cpx.
MÉTODOS
El trabajo se estructuró en 4 estudios fundamentales, uno relacionado con las variantes de VIH que circularon en pacientes cubanos en el período 2014-2019. Otros 2 concernientes a la relación de las variantes y el uso del correceptor o fenotipo viral y uno relacionado con el origen, la filogeografía y la diseminación del CRF19_cpx.
Se determinó el subtipo viral de VIH-1 en 701 muestras de pacientes tratados, seropositivos al VIH-1 del período 2014-2019 mediante secuenciación parcial del gen pol.8
Para determinar la relación entre los subtipos virales y el uso del correceptor se estudiaron 176 plasmas de pacientes infectados con VIH-1. De ellos, 107 correspondían al período 2014-2016 y 69 al período 2017-2019. Se les determinó el subtipo viral y la predicción del fenotipo viral (R5, R5X4 o X4). Se secuenciaron los genes env (región V2C2V3) y pol (reverso transcriptasa y proteasa). 8,9 La predicción del fenotipo se realizó con Geno2pheno. 10
Para el estudio del origen y evolución del CRF19_cpx se analizaron un conjunto de datos que comprendieron 350 secuencias parciales de los genes pol y env de muestras de pacientes cubanos infectados con VIH durante los últimos 15 años y 350 obtenidas de la base de datos de Los Álamos. Este conjunto de datos contenía secuencias del recombinante CRF19_cpx (315) y secuencias de los subtipos A1, D y G. Se realizaron análisis utilizando enfoques de máxima verosimilitud, que incluyeron la reconstrucción de filogenia, el análisis espacio-temporal de la propagación del virus y la reconstrucción del carácter ancestral, así como el modo de transmisión y las mutaciones de resistencia a fármacos (DRM). (11-13
RESULTADOS Y DISCUSIÓN
Subtipos y formas recombinantes del VIH-1 en pacientes cubanos tratados
En el período 2014-2019 el análisis de las secuencias de VIH-1 reveló que los subtipos detectados con mayor frecuencia fueron el subtipo B (29,5 %), el CRF19_cpx (25,9 %), los CRF_BG (20, 23, 24) (28,1 %), el CRF18_cpx (11,3 %) y las formas recombinantes únicas (7,3 %). Aunque hasta el momento se han detectado otros subtipos (G, C, H, A1 y F1), la frecuencia actual de los mismos se encuentra por debajo del 5 % cada una. Es importante destacar que las formas recombinantes del VIH-1 en su conjunto, fueron mayoría (72,6 %).
Predicción del tropi smo en variantes de VIH-1 que circulan en Cuba
El análisis de las 107 secuencias que abarcaron el período 2014-2016 reveló una asociación entre el CRF19_cpx y los virus con tropismo X4 o R5X4. Por el contrario, la mayoría de los CRF_BG mostraron virus con tropismo R5 (tabla 1).
Tabla 1 Uso del correceptor entre los diferentes subtipos en 107 secuencias de VIH-1 cubanas del período 2014-2016
| Subtipo en la región env | |||||
|---|---|---|---|---|---|
| Número de muestras (%) | Todos los subtipos | Subtipo B | CRF19_cpx | CRF18_cpx | CRF_BG (20-23-24) |
| 107 (100 %) | 35 (100 %) | 29 (100 %) | 9 (100 %) | 34 (100 %) | |
| R5 (%) | 66 (61,7 %) | 22 (62,9 %) | 10 (34,5 %) | 8 (88,9 %) |
26 (76,5 %) p = 0,032; OR: 2,68; IC: 1,07-6,70 |
| R5X4 (%) | 24 (22,4 %) | 6 (17,1 %) |
11 (37,9 %) p = 0,019; OR:3,05; IC:1,17-7,96 |
1 (11,1 %) | 6 (17,6 %) |
| X4 (%) | 17 (15,9 %) | 7 (20 %) |
8 (27,6 %) p = 0,044; OR: 2,92; IC:1.00-8.51 |
0 (0,0 %) | 2 (5,9 %) |
| R5X4/X4 (%) | 41 (38,3 %) | 13 (37,1 %) |
19 (65,5 %) p = 0,0004;OR:4.83; IC:1,95-12,02 |
1 (11,1 %) | 8 (23,5 %) |
OR: Razones de desigualdad (del inglés, odd ratio), IC: intervalo de confianza del 95 %
De las muestras correspondientes a los años 2017-2019, 33 pertenecían a pacientes tratados y 36 a no tratados, de ellas, la mayoría mostró el fenotipo R5. Sin embargo, los subtipos C y CRF19_cpx mostraron asociación significativa con el empleo del correceptor CXCR4 (tabla 2).
Tabla 2 Comparación de la predicción del uso del correceptor entre los subtipos. Análisis del gen env y los subtipos globales de VIH en muestras del período 2017-2019
| Correceptor | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Muestras N (%) | Muestras N (%) | ||||||||||||
| Total | B | C | CRF_BG | CRF19_cpx | CRF18_cpx | Total | B | C | CRF_BG | CRF19_cpx | CRF18_cpx | URF | |
| 69 (100%) | 26 (100 %) | 5 (100 %) | 17 (100 %) | 18 (100 %) | 3 (100 %) | 58 (100 %) | 18 (100 %) | 5 (100 %) | 10 (100 %) | 12 (100 %) | 1 (100 %) | 12 (100 %) | |
| R5 | 44 (63,77 %) | 19 (73,08 %) | 2 (40 %) | 12 (70,58 %) | 8 (44,44 %) | 3 (100 %) | 36 (62,06 %) | 14 (73,91 %) | 2 (40 %) | 8 (71,43 %) | 4 (42,86 %) | 1 (100 %) | 7 (54,55 %) |
| Valor p R5 |
P = 0,0487 OR: 0,343 CI:0,11-0,99 |
P = 0,0234 OR: 0,236 CI:0,64-0,86 |
|||||||||||
| R5X4 | 14 (20,29 %) | 4 (15,38 %) | 0 (0 %) | 4 (23,53 %) | 6 (33,33 %) | 0 (0 %) | 12 (20,68 %) | 1 (13,04 %) | 0 (0 %) | 2 (21,43 %) | 6 (42,86 %) | 0 (0 %) | 3 (27 %) |
| Valor p R5X4 |
P = 0,0051 OR: 6,230 CI:1,58-24,43 |
||||||||||||
| X4 | 11 (15,94 %) | 3 (11,54 %) | 3 (60 %) | 1 (5,88 %) | 4 (22,22 %) | 0 (0) | 10 (17,24 %) | 3 (13,04 %) | 3 (60 %) | 0 (0 %) | 2 (14,29 %) | 0 (0) | 2 (18,18 %) |
| Valor p X4 |
P = 0,0260 OR: 9,305 CI:1,57-54,96 |
P = 0,0318 OR: 9,680 CI:1,43-52,34 |
|||||||||||
| R5X4/X4* | 25 (36,23 %) | 7 (26,92 %) | 3 (60 %) | 5 (29,41 %) | 10 (55,56 %) | 0 (0 %) | 22 (37,93 %) | 4 (26,09 %) | 3 (60 %) | 2 (28,57 %) | 8 (57,4 %) | 0 (0 %) | 5 (45,45 %) |
| Valor p R5X4/X4* |
P = 0,0487 OR:2,90 CI:1,00-8,42 |
P = 0,0234 OR: 4,233 CI:1,15-15,52 |
|||||||||||
OR: Razones de desigualdad (del inglés, odd ratio), IC: intervalo de confianza del 95 %
Al estratificar el uso de correceptor entre pacientes tratados y no tratados se revelaron algunas diferencias entre los subtipos. Los virus del subtipo C fueron predominantemente X4 en los pacientes tratados (3/4; 75 %; p = 0,0178; OR: 14,77; IC: 1,71-127,58) y los virus CRF19_cpx de tipo R5X4 (3/7; 42,86 %; p=0,0375; OR: 5,88; IC: 1,09-32,74). (4/7 pacientes; 36,36 %; p = 0,0081; OR: 27,00; IC: 1,29-56,39) a pesar de que el 81,82 % tenían diagnóstico reciente. Al comparar los valores de CV entre los diferentes subtipos virales teniendo en cuenta la variante detectada al secuenciar el gen env, pol y en el análisis del subtipo global (datos no mostrados) se observaron diferencias en los valores de CV entre los subtipos, demostrándose cifras de CV significativamente superiores para los virus CRF19_cpx en comparación con los otros virus (p ≤ 0,05).
Origen e historia evolutiva del recombinante de VIH-1 CRF19_cpx
Para el desarrollo de la investigación se reconstruyeron árboles filogenéticos y se encontró que todas las secuencias de CRF19_cpx formaban un grupo único (figura 1). Las fechas obtenidas para el ancestro común más cercano (MRCA, del inglés most recent common ancester) del clúster CRF19_cpx se estimó en la década de 1970 y para sus virus parentales (es decir, D, A1 y G) en 1969, 1959 y1958 respectivamente (figura 1 y tabla 3).

Fig. 1 Árboles a escala temporal reconstruidos para los 3 conjuntos de datos: A1 + CRF19 (arriba a la izquierda), G + CRF19 (arriba a la derecha) y D + CRF19 (abajo al centro). Los nodos y las ramas están coloreados por ubicación (ACR realizado por PastML). Cuba está coloreada de rojo, África en marrón, excepto el Congo (violeta), los países europeos en color verdes, los países asiáticos en azul, los países de América (Brasil y EE. UU.) en naranja, Australia en amarillo. Las tiras de colores alrededor de los árboles muestran los subtipos: A1 (azul claro), D (amarillo), G (naranja), CRF19 (rojo) y A1-A3 recombinante (azul, tenga en cuenta que esta secuencia se usó en el A1 + El conjunto de datos CRF19 como parte que se alinea con la parte A1 de CRF19 es completamente A1). Se muestran las fechas (con IC, reconstruido por LSD2) y países (con probabilidades marginales, reconstruido por PastML) de los MRCA del clúster CRF19 y sus nodos parentales no CRF19 (es decir, A1, D o G). También se muestran las fechas de los clústeres cubanos no recombinantes.
Tabla 3 Fechas del ancestro común más cercano (MRCA) del CRF19_cpx y sus parentales no CRF19_cpx, en los diferentes grupos de datos analizados
| Datos | CRF19. Fecha del MRCA | No CRF19. Fecha del MRCA |
|---|---|---|
| D + CRF19 |
1974 (1970-1977) (LSD2) 1972 (1963-1980) (BEAST) |
1959 (1954-1962) |
| A1 + CRF19 | 1978 (1975-1980) | 1969 (1966-1972) |
| G + CRF19 | 1975 (1970-1980) | 1958 (1950-1963) |
El análisis de la filogeografía a nivel provincial demostró que la epidemia se introdujo muy temprano en Cuba, probablemente por la provincia de Villa Clara (probabilidad marginal de 0,92; fecha de 1977; CI: 1973-1979) antes del descubrimiento del VIH en la década de 1980. Luego CRF19_cpx dio lugar a múltiples introducciones de Villa Clara a La Habana en las décadas de 1980 y 1990 y luego se extendió a otras provincias (figura 2).
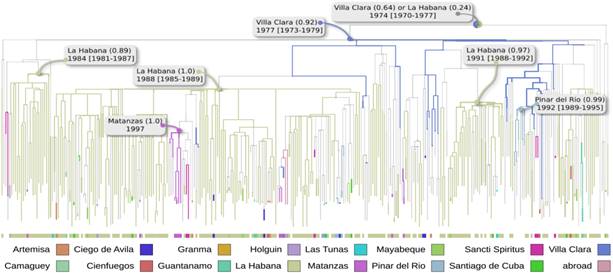
Fig. 2 Filogeografía (nivel provincial) del CRF19_cpx. La ACR y la visualización se realizaron con PastML [18] (MPPA + F81). Las ramas están coloreadas por provincias (el código de color se explica a continuación). Las ramas grises más delgadas corresponden a un cambio de provincia. La raíz está sin resolver entre Villa Clara y La Habana. Se indicó el nodo resuelto más antiguo y su fecha, correspondiente a Villa Clara (probabilidad marginal 0,92). Igualmente se marcaron las fechas y probabilidades marginales de las principales introducciones de Villa Clara a La Habana.
El subtipo B de VIH-1 se reconoció como el más prevalente en la región de las Américas, Caribe, Europa y Oceanía, 14,15 en contraste a lo reportado en Cuba en estudios anteriores y en la presente investigación, donde se ha observado la gradual disminución de dicho subtipo a través de los años. Esto último se verificó en estudios cubanos realizados en varios períodos. En pacientes del año 1999 se identificó un 48 % del subtipo B, del 2003 un 41,2 %, en el período 2009 a 2014 la frecuencia se redujo al 30,9 % y en el presente estudio se mostró ligeramente inferior (29,5 %). 4,16,17,18
El incremento en la circulación de los recombinantes ha sido notorio en los últimos años a nivel mundial. 19 Los resultados antes descritos han ratificado la considerable diversidad genética del VIH-1 en Cuba, lo que se ha atribuido al origen de la epidemia cubana a partir de contactos con la epidemia de VIH de África Central. 4,5,8
El tropismo o fenotipo viral observado para el CRF19_cpx con preferencia por el uso del correceptor CXCR4 (virus X4 o R5X4) ha sido un hallazgo interesante. Además, en los no tratados, aquellos infectados con CRF19_cpx fueron mayoritariamente del tipo X4. Esto ha apoyado la hipótesis de que el tropismo CXCR4 observado para el CRF19_cpx no está relacionado con la evolución de la historia natural de la infección con VIH, sino con características del virus. 7) Sin embargo, como no tenemos certeza de la fecha de infección de estos pacientes, no se puede descartar que sean diagnósticos tardíos. Por el contrario, el subtipo C (también asociado en este estudio a fenotipo X4) se encontró principalmente en pacientes tratados, con 5 o más años de diagnosticados, coincidiendo con lo establecido clásicamente para el cambio de tropismo de R5 a X4. 20
Se ha conocido que el cambio en el tropismo viral de R5 a R5X4 ó X4 ha estado asociado con un deterioro acelerado del sistema inmune y un pronóstico clínico desfavorable. 2,20) Es probable que algunos subtipos de VIH-1, como el CRF19_cpx tengan mayor probabilidad de evolucionar hacia variantes X4 y R5X4 durante el curso de la infección.
La causa de un uso del correceptor dependiente del subtipo por parte del virus podría explicarse por las propiedades virológicas intrínsecas de cada subtipo (es decir, mayor capacidad replicativa, diversidad antigénica) o por el efecto diferencial de los virus sobre el sistema inmune del hospedero, lo cual puede llevar eventualmente al aumento de la población de virus X4. 20,21 Los resultados de la presente investigación 22,23,24 ha estado en concordancia con lo antes reportado para el CRF19_cpx y han reforzado la teoría de que este virus podría tener una alta capacidad replicativa y mayor patogenicidad.
Considerando el análisis filogenético del origen y evolución del CRF19_cpx hemos sugerido que el virus se recombinó entre la comunidad cubana presente en África subsahariana desde la década de 1960, muy probablemente en la República Democrática del Congo, como consecuencia de la cocirculación de los subtipos A, D y G. 3,4 Se introdujo en Cuba muy temprano, en la década de 1970 y comenzó a extenderse a partir de un solo evento de transmisión, primero en Villa Clara y luego en la década de 1980 en La Habana. Esta temprana introducción a Cuba podría explicar el éxito local del CRF19_cpx que no tuvo éxito en otras partes del mundo. 25,26
El éxito local ha resultado de un efecto fundador en lugar de alguna ventaja de transmisión de CRF19_cpx respaldada por la incidencia relativamente estable de CRF19_cpx a lo largo del tiempo, con 18,3 % en 2007 (muestreado en 2003), 27,5 % en 2012 (diagnosticado entre 2009-2010) y 24,1 % en 2019 (muestreado en 2017).
Conclusiones
El tropismo preferencial por el correceptor CXCR4 detectado en el CRF19_cpx, acompañado de una mayor replicación viral y sin relación con el tiempo de diagnóstico de los pacientes ha reforzado la hipótesis de que esta variante viral pudiera tener mayor patogenicidad. El análisis filogenético mostró una introducción muy temprana de CRF19_cpx en Cuba, lo que podría explicar su éxito epidemiológico.














