Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Pediatría
versión On-line ISSN 1561-3119
Rev Cubana Pediatr v.82 n.2 Ciudad de la Habana abr.-jun. 2010
Mecanismos biológicos implicados en el comportamiento epidemiológico y patogénico de la influenza
Biological mechanisms involved in epidemiological and pathogenic behavior of influenza
Amaury Noda Albelo,I Lázaro Arturo Vidal Tallet,II Manuel Araña Rosaínz,III Andrea Xiomara Casal MenéndezIV
IEspecialista de I Grado en Inmunología Clínica. Máster en Infectología y Enfermedades Tropicales. Máster en Atención Integral al Niño. Asistente. Hospital Pediátrico «Eliseo Noel Caamaño». Matanzas, Cuba.
IIEspecialista de II Grado en Pediatría. Máster en Enfermedades Infecciosas. Máster en Educación Superior. Profesor Consultante. Hospital Pediátrico «Eliseo Noel Caamaño». Matanzas, Cuba.
IIIDoctor en Ciencias Médicas. Especialista de II Grado en Inmunología Clínica. Instituto Nacional de Endocrinología. La Habana, Cuba.
IVEspecialista de II Grado en Microbiología. Máster en Enfermedades Infecciosas. Asistente. Educación Superior. Profesor Consultante. Hospital Pediátrico «Eliseo Noel Caamaño». Matanzas, Cuba.
RESUMEN
La influenza estacional y pandémica continúa siendo un desafío para los investigadores y clínicos. Se realiza una revisión de los mecanismos biológicos implicados en el comportamiento epidemiológico y patogénico de la influenza, así como de algunos aspectos generales de sus manifestaciones clínicas y la terapéutica antiviral.
Palabras clave: Mecanismos biológicos, comportamiento epidemiológico, comportamiento patogénico, influenza.
ABSTRACT
The seasonal and pandemic influenza remains as a challenge for researchers and clinicians. Authors carry out a review of biological mechanisms involved in epidemiological and pathogenic of influenza, as well as of some general features of its clinical manifestations and the antiviral therapy.
Key words: Biological mechanisms, epidemiological behavior, pathogenic behavior, influenza.
INTRODUCCIÓN
Entre 1740 y 1758 el Papa Benedicto XIV acuña el término influenza para referirse a la enfermedad causada por influencia del frío o de los astros.1 La Organización Mundial de la Salud estima que durante las epidemias anuales de influenza se afecta entre el 5 y el 15 % de la población, con 3 a 5 millones de casos graves y 250 000 a 500 000 muertes en el mundo.2 En Estados Unidos de América es motivo de más de 200 000 hospitalizaciones y 40 000 defunciones,3 e implica un costo por concepto de atención médica y reducción de la productividad de 87 millones de dólares.4
La influenza es la enfermedad febril aguda usualmente autolimitada que resulta de la infección por el virus de la influenza A o B; existe un virus C, pero es de escasa importancia clínica o epidemiológica, ya que produce cuadros respiratorios altos banales de manera esporádica. El impacto de las epidemias de influenza varía de año en año en dependencia de la virulencia de la cepa de virus circulante y de la presencia o ausencia de inmunidad poblacional al virus epidémico.
BIOLOGÍA DEL VIRUS Y EPIDEMIOLOGÍA
Los virus influenza pertenecen a la familia Orthomyxoviridae, son virus ARN.5 Los virus A y B se diferencian en cuanto al comportamiento epidémico y el tropismo de hospederos, pero en el orden clínico es difícil diferenciarlos. El influenza B solo afecta a seres humanos y es incapaz de provocar pandemias,6 mientras que el virus A tiene una amplia gama de hospederos (seres humanos, cerdos, aves y mamíferos marinos) y puede generar pandemias.7 En el cuadro se resumen estas características.
Cuadro. Diferencias entre los virus influenza A, B, y C (Orthomyxoviridae, ARN)
| Parámetros | Influenza A | Influenza B | Influenza C |
| Genética | 8 segmentos génicos | 8 segmentos génicos | 7 segmentos génicos |
| Estructura | 10 proteínas virales | 11 proteínas virales | 9 proteínas virales |
| Hospederos | Seres humanos, cerdo, equinos, aves, mamíferos marinos | Seres humanos | Seres humanos y cerdos |
| Epidemiología | Deriva antigénica y cambio antigénico | Solo deriva antigénica; más de una variante puede cocircular | Solo deriva antigénica; múltiples variantes |
| Características clínicas | Puede causar pandemias con alta mortalidad en jóvenes | Enfermedad grave, limitada a ancianos y personas con alto riesgo; no pandemias | Enfermedad ligera, esporádica |
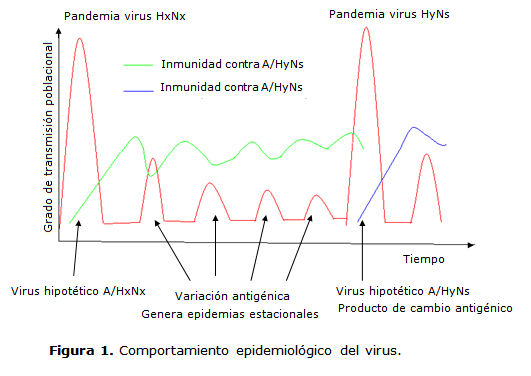
El virus de la influenza tiene un comportamiento excepcional desde el punto de vista epidemiológico, se caracteriza por la generación de pandemias cíclicas entre las cuales ocurren brotes epidémicos estacionales.8 Esta epidemiología especial depende de dos factores: el estado de inmunidad poblacional, que impide la propagación del virus, y la facilidad del virus para evadir esta defensa cambiando sus características antigénicas (figura 1).
Las pandemias resultan de la introducción de un nuevo virus (características antigénicas muy diferentes a sus antecesores), con un nivel de inmunidad poblacional muy bajo. En el período interpandémico ocurren epidemias estacionales, por pequeñas variaciones antigénicas que logran burlar, de manera limitada, la inmunidad poblacional. Los fenómenos que explican los grandes cambios antigénicos (que generan pandemias) y las pequeñas variaciones antigénicas (responsables de la epidemias estacionales) se sustentan desde el orden molecular en dos características del virus: el carácter segmentario del genoma y la ineficiencia de la ARN polimerasa del virus.
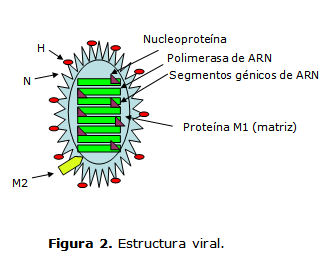
El genoma constituido por ARN está formado por segmentos independientes (8 segmentos) que codifican 10 proteínas (figura 2) y PB1-F2, que potencia la actividad proapoptótica inducida por el virus, pero no está siempre presente (inconstante):9,10
1. Hemaglutinina (H): es el ligando del receptor viral. Define el tropismo de especie y el área del árbol respiratorio que infecta. Está implicada en la eficacia de transmisión. Su ligando es un residuo de ácido siálico en la membrana celular del hospedero.
2. Neuraminidasa (N): cliva el ácido siálico y libera el virión durante el ciclo vital del virus. Es la diana de intervención terapéutica sobre la que actúa el oseltamivir y el zanamivir (derivados sintéticos similares al ácido siálico).
3. M1: proteína de la matriz.
4. Proteína M2: controla el pH en el interior del virus; es un canal iónico que permite que el genoma viral se libere durante el proceso de replicación. Es la diana de intervención terapéutica de la amantadina y la rimantadina, que bloquean su acción.
5. Proteína NS1: inhibe la respuesta inmune de hospedero.
6. Proteína NS2: transporta las ribonucleoproteínas (RNP) del núcleo al citoplasma.
7. Nucleoproteína NP: se une al genoma viral.
8. PB1, PB2, PA: actividad ARN polimerasa.
La nomenclatura del virus para el caso de influenza A incluye el subtipo de dos de estas proteínas, la hemaglutinina y la neuraminidasa. De modo general se nombran de la manera siguiente: tipo de virus/especie del hospedero (no humano)/lugar donde fue aislado/numero de la cepa/año de aislamiento/subtipo de hemaglutinina y neuraminidasa (esto último para influenza A). Ejemplo:
- Influenza A/California/04/2009 (H1N1).
- Influenza A/California/7/2004 (H3N2).
- Influenza B/Hong Kong/20/2003.
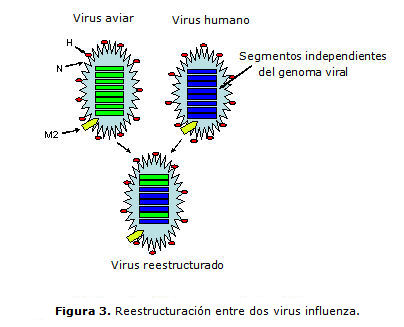
La característica segmentaría del genoma brinda la posibilidad de la reestructuración azarosa entre dos virus o más, que coincidan en un hospedero dado, generando un virus nuevo en el orden antigénico y también en cuanto a tropismo de hospedero y a eficacia de transmisibilidad (figura 3).
La otra característica importante es la ineficiencia de la ARN polimerasa vírica. Esta enzima es incapaz de corregir errores de replicación, por lo que en el proceso de replicación del genoma viral, de manera constante, se introducen mutaciones, que implican la presencia de virus con diferencias de mayor o menor envergadura en el orden antigénico. Cuando el cambio antigénico viral es lo suficientemente importante para evadir la inmunidad poblacional y generar pandemias, se denomina al fenómeno cambio antigénico, y los cambios pequeños que generan epidemias estacionales se conocen como deriva antigénica.11
Es de señalar que además de las variaciones antigénicas mencionadas, estas mutaciones también alteran el tropismo de especie y la posibilidad de transmisión, por lo que un virus de una especie puede eventualmente adaptarse a otra.12
Transmisión y replicación del virus
La transmisión de influenza A ocurre a través de microgotas de secreciones respiratorias que son muy pesadas para mantenerse suspendidas por distancias superiores a 1 m. Las condiciones climáticas, relacionadas con la humedad relativa y la temperatura, repercuten en la eficiencia de esta transmisión, de tal manera que en el hemisferio norte la estación de influenza se extiende entre los meses de noviembre a marzo, mientras que en el sur de mayo a septiembre.13 Las manos contaminadas con el virus al contactar las mucosas son otra vía de infección.14 El germen penetra en el árbol respiratorio superior donde se inicia la replicación viral a nivel de las células epiteliales.
Es importante señalar que el receptor reconocido por el virus, evento que realiza a través de la hemaglutinina (H), es un residuo de ácido siálico que se encuentra en la membrana de las células epiteliales.15 Este residuo está unido a una galactosa por un enlace que puede ser de tipo alfa 2,3 o alfa 2,6; en los humanos el receptor predominante en vías aéreas superiores es alfa 2,6 y en aves alfa 2,3.16
Es interesante conocer que en el cerdo existe la presencia de ambos tipos de receptores en vías respiratorias superiores. Los virus aviares reconocen alfa 2,3 y los humanos alfa 2,6, y el cambio de preferencia de la H vírica de alfa 2,3 por alfa 2,6 favorece que un virus aviar se adapte a humanos.17 El cerdo puede ser afectado por virus de origen humano, aviar y los propios del cerdo, constituyendo el reservorio ideal para que ocurran reestructuraciones víricas. Se han descrito 16 H y 9 neuraminidasas (N);18 todas se encuentran presentes en virus de influenza que colonizan el intestino de aves marinas silvestres, por lo que estos animales son el reservorio natural de todos los virus de influenza.19
Una vez que ocurre la unión entre la H y el ácido siálico, el virus penetra en la célula diana en una vesícula endosómica, el cambio de pH modifica la H y libera al virus de la unión a la membrana. La proteína M2, cuya función es actuar como canal iónico, induce cambios de pH intraviral y permite, durante el ciclo replicativo, que el genoma viral se desprenda de la matriz y se libere al citoplasma celular y posteriormente al núcleo. La maquinaria celular sintetiza las nuevas proteínas víricas y se ensambla un nuevo virión que eclosiona de la célula, pero se mantiene unido a la membrana y necesita ser liberado por la N, que degrada el ácido siálico.8 Este es un momento crítico de ciclo replicativo viral susceptible de intervención terapéutica; los agentes antivirales oseltamivir y zanamivir son estructuralmente semejantes el ácido siálico, se unen e inhiben la N, provocan aglomeración viral en la membrana celular e interrumpen el ciclo de replicación.
Después de ocurrida la infección por el virus de la influenza pueden transcurrir entre 18 y 72 h antes del comienzo de la sintomatología clínica; sin embargo, la excreción viral ocurre desde antes del comienzo de los síntomas (0-24 h) y continúa por 5-10 días, la excreción viral es más prolongada en niños pequeños e inmunocomprometidos.
Pandemias conocidas
En el siglo XX ocurrieron 3 pandemias de influenza: en 1918 por un virus aviar adaptado al humano A/H1N1, en 1957 por un virus reestructurado aviar-humano A/H2N2, y en 1968 por un virus reestructurado aviar-humano A/H3N2.20
Un virus pandémico puede producirse por reestructuración entre dos o más virus o por adaptación de un virus a una nueva especie, justificadas ambas circunstancias por los fenómenos de deriva antigénica y cambio antigénico antes descritos.
La primera pandemia de influenza del siglo XXI ocurre por un virus reestructurado A/H1N1 aviar-cerdo-humano; de los 8 fragmentos génicos que constituyen su genoma 1 es humano, 2 son aviares y 5 son de cerdo.21
La influenza A difiriendo de otras pandemias que amenazan la humanidad, tiene potencial para infectar al 30 % de la población mundial, y si se asume un estimado conservador de letalidad del 2 % resultaría en 135 millones de defunciones en el primer año de la nueva pandemia; esto significa 4 veces la mortalidad total atribuible al VIH-1 en los últimos 30 años.22
PATOGENIA
El virus se replica, de manera local, provocando principalmente una traqueobronquitis transitoria, sin generar viremia, por lo que las manifestaciones clínicas dependen fundamentalmente de la liberación sistémica de mediadores con actividad proinflamatoria producido localmente, como la interleucina-6 (IL-6), el factor de necrosis tumoral-alfa (TNF-α) y la IL-1.23,24 De esta manera la exuberante respuesta inflamatoria en el parénquima pulmonar es responsable de la letalidad asociada al virus de la influenza. Las cepas virales más letales se caracterizan por una capacidad excepcional de inducción de una respuesta de producción de citocinas proinflamatorias por los macrófagos.25 La actividad de la óxido nítrico sintasa inducible también contribuye al daño durante la infección.26
Uno de los mecanismos defensivos cruciales del hospedero frente a la infección lo constituyen los interferones tipo I alfa y beta.27 La eficiencia de la proteína viral NS1 bloquea este mecanismo permitiendo que el virus evada la respuesta inmune.
En la enfermedad complicada la replicación viral se extiende a las porciones inferiores del árbol respiratorio afectando bronquiolos y alvéolos, y provoca broquitis necrotizante y bronquiolitis, con inducción de apoptosis de las células epiteliales, trombosis capilar y de los pequeños vasos y daño de los neumocitos tipo I y tipo II. Estos fenómenos generan edema intersticial e infiltrado inflamatorio, se forma membrana hialina en el alvéolo y el ductus alveolar, y se compromete el intercambio de gases.28 A nivel de bronquiolo y alvéolo, es más frecuente la presencia de residuos de ácido siálico unidos a galactosa por enlaces alfa 2,3.
Como antes se comentó los virus aviares tienen mayor afinidad por estos receptores, lo que en parte puede explicar la mayor frecuencia de complicación y letalidad del virus A/H5N1 que afecta con mayor intensidad las porciones bajas del árbol respiratorio.29
MANIFESTACIONES CLÍNICAS
Las manifestaciones clínicas de la influenza son dependientes de la edad del paciente:
• Menores de 2 meses: puede simular sepsis bacteriana; en el neonato es común la apnea.30
• Lactantes: se solapan los síndromes producidos por influenza y otros virus, en especial, virus sincicial respiratorio (SRV), adenovirus y parainfluenza, principalmente manifestaciones respiratorias inespecíficas, bronquiolitis, etc.
• Menores de 5 años: principalmente fiebre y síntomas respiratorios altos, incluido laringotraqueobronquitis, aunque es importante señalar que entre el 10 y el 50 % de los pacientes se describe sintomatología baja como bronconeumonía generalmente de evolución benigna.
• Mayores de 5 años y adultos: se caracteriza por comienzo abrupto de fiebre acompañado de cefalea, faringe enrojecida, mialgias, anorexia y tos seca.31 Es de señalar que la combinación de tos y fiebre es el mejor predictor de influenza en adultos, con un valor predictivo positivo del 79 % en etapas de circulación de influenza.32
La enfermedad respiratoria alta se caracteriza por tos seca que empeora para el tercer o cuarto día y persiste por una o dos semanas, después que el resto de los síntomas desaparecen. La faringitis es frecuente y no se acompaña de exudados. Así como la rinorea, el lagrimeo y la fotofobia, cuando aparece crup es más grave que el asociado a virus parainfluenza y SRV. La fiebre típicamente alta en las primeras 72 h. El compromiso de las vías aéreas, en ocasiones severo, se asocia a superinfección bacteriana. Puede aparecer otitis media aguda entre el 3 el 5 % de los pacientes, de manera habitual, 3 a 4 días después del comienzo de los síntomas respiratorios. Los síntomas gastrointestinales (vómitos, dolor abdominal y diarrea) son más comunes en niños que en adultos.33
TRATAMIENTO
El virus pandémico A/H1N1 es resistente a los inhibidores de la proteína M2 (amantadina y rimantadina) pero es sensible a los inhibidores de la N (oseltamivir y zanamivir). El tratamiento con estos inhibidores de N se debe iniciar en las primeras 48 h del comienzo de los síntomas, y en las dosis siguientes de acuerdo con la edad del paciente
- Menores de 1 año:
- < 3 meses: 12 mg, 2 veces al día.
- 3-5 meses: 20 mg, 2 veces al día.
- 6-11 meses: 25 mg, 2 veces al día.
- Mayores de 1 año: 4 mg/kg
- Adultos: 75 mg 2 veces al día.
En todos los casos el tratamiento es por 5 días. En casos graves se puede duplicar la dosis y prolongarla, aunque no existen evidencias de que esto sea más eficaz. En la profilaxis se utiliza la mitad de la dosis por 10 días.
La dosis del zanamivir por vía inhalatoria es 10 mg (2 aplicaciones), 2 veces al día, durante 5 días de tratamiento. En la profilaxis se utiliza igualmente la mitad de la dosis por 10 días. En el niño menor de 6 años no está indicado.
Peramivir
Peramivir es un nuevo inhibidor de N, es de uso endovenoso su presentación en el mercado es en viales de 200 mg/20 mL (10 mg/mL).34
Dosis (una vez al día por 5 a 10 días):
• < 30 días: 6 mg/kg
• 31-90 días: 8 mg/kg
• 91-180 días: 10 mg/kg
• 181 días -5 años: 12 mg/kg
• 6 años-17 años: 10 mg/kg
Dosis máxima diaria: 600 mg endovenoso. Se debe reajustar dosis cuando exista insuficiencia renal.
REFERENCIAS BIBLIOGRÁFICAS
1. Diarte-Arellano. La Influenza. Parte I: Aspectos históricos. AS Sin. 2009;3(2):21-5.2.
2. Maltezou HC. Nosocomial influenza: new concepts and practice. Current Opinion in Infectious Diseases. 2008;21(4):337-43.
3. Dushoff J, Plotkin JB, Viboud C, Earn DJ, Simonsen L. Mortality due to influenza in the United States-an annualized regression approach using multiple-cause mortality data. Am J Epidemiol. 2006;163(2):181-7.
4. Molinari NA. The annual impact of seasonal influenza in the US: measuring disease burden and costs, Vaccine. 2007;25(27):5086-96.
5. Jindal N, Chander Y, Abin M. Amplification of four genes of influenza A viruses using a degenerate primer set in a one step RT-PCR method, Journal of Virological Methods. 2009;160:163-6.
6. Wright PF, Webster RG. Orthomyxoviruses. In: Knipe, D.M., Howley, P.M. (Eds.), Fields virology, fourth ed. Philadelphia: Lippincott Williams &Wilkins; 2001. Pp. 1533-79.
7. Beigel J, Bray M. Current and future antiviral therapy of severe seasonal and avian influenza. Antiviral Research. 2008;78:91-102.
8. Salomon R, Webster RG. The influenza virus enigma. Cell. 2009;136:402-10.
9. Dugan VG, Chen R, Spiro DJ. The evolutionary genetics and emergence of avian influenza viruses in wild birds. PLoS Pathog. 2008;4(5): e1000076. Disponible en: http://www.plospathogens.org/article/info:doi/10.1371/journal.ppat.1000076
10. Obenaue JC, Denson J, Mehta PK. Large-scale sequence analysis of avian influenza isolates. Science 311. 2006;1576-80.
11. Grebe KM, Yewdell JW, Bennink JR. Heterosubtypic immunity to influenza A virus: where do we stand. Microbes and Infection. 2008;10:1024-9.
12. Webster RG. Wet marketsa continuing source of severe acute respiratory syndrome and influenza? Lancet.. 2004;363:234-6.
13. Zuk T, Rakowski F, Radomski JP. A model of influenza virus spread as a function of temperature and humidity. Computational Biology and Chemistry. 2009;33:176-80.
14. Macias AE, de la Torre S, Moreno-Espinosa A. Controlling the novel A (H1N1) influenza virus: don't touch your face!, Journal of Hospital Infection. 2009;73:280-91.
15. Krug RM, Aramini JM. Emerging antiviral targets for influenza A virus. Trends in Pharmacological Sciences. 2009;30(6): 269-277 .
16. Shriver Z, Raman R, Viswanathan K. Context-specific target definition in influenza a virus hemagglutinin-glycan receptor interactions. Chemistry & Biology. 2009;16:803-14.
17. Bewley CA. Illuminating the switch in influenza viruses. Nat. Biotechnol. 2008;26:60-2.
18. Skehel J. An overview of influenza haemagglutinin and neuraminidase Biologicals. 2009;37:177-8.
19. Naffakh N, van der Werf S. April 2009: an outbreak of swine-origin influenza A(H1N1) virus with evidence for human-to-human transmission. Microbes and Infection. 2009;11:725-8.
20. Zimmer SM, Burke DS. Historical perspective-emergence of influenza A (H1N1) viruses. N Engl J Med. 2009;361:279-85.
21. Wang TT, Palese P. Unraveling the mystery of swine influenza virus. Cell. 2009;137: 883-5.
22. Gatherer D. The 2009 H1N1 influenza outbreak in its historical context. Journal of Clinical Virology. 2009;45:174-8.
23. de Jong, MD, Simmons CP, Thanh TT, Hien VM, Smith GJ, Chau TN, et al. Fatal outcome of human influenza A (H5N1) is associated with high viral load and hypercytokinemia. Nat Med. 2006;12:1203-7.
24. Kash JC, Goodman AG, Korth MJ, Katze MG. Hijacking of the host-cell response and translational control during influenza virus infection. Virus Res. 2006;119:111-20.
25. Snelgrove RJ, Edwards L, Rae AJ, Hussell T. An absence of reactive oxygen species improves the resolution of lung influenza infection. Eur J Immunol. 2006;36(6):1364-3.
26. McCarty MF. Practical strategies for targeting NF-kappaB and NADPH oxidase may improve survival during lethal influenza epidemics. Med Hypotheses. 2009; 74(1):18-20
27. Tumpey TM, Szretter KJ, Van Hoeven. The Mx1 gene protects mice against the pandemic 1918 and highly lethal human H5N1 influenza viruses. J Virol. 2007;81:10818-21.
28. Barnard DL. Animal models for the study of influenza pathogenesis and therapy antiviral. Research. 2009;82:A11022.
29. van Riel, D, Munster VJ, de Wit E, Rimmelzwaan GF, Fouchier RAM, Osterhaus ADME, Kuiken T. H5N1 Virus attachment to lower respiratory tract. Science. 2006;312(5772):399.
30. Meibalane R, Sedmak GV, Sasidharan P. Outbreak of influenza in a neonatal intensive care unit. J Pediatr. 1977;91:974-6.
31. Taubenberger JK, Morens, DM, The pathology of influenza virus infections. Ann Rev Pathol. 2008;3:499-522.
32. Monto AS, Gravenstein S, Elliott M. Clinical signs and symptoms predicting influenza infection. Arch Intern Med. 2000;160:3243-7.
33. Wright PF, Bryant JD, Karzon DT: Comparison of influenza B/Hong Kong virus infections among infants, children, and young adults. J Infect Dis. 1980;141:430-5.
34. Hamburg M. Peramivir letter for emergency use authorization. October 23, 2009. Available at: http://www.www.fda.gov/downloads/Drugs/DrugSafety /PostmarketDrugSafetyInformationforPatientsandProviders/UCM187800.pdf Accessed November 4. 2009).
Recibido: 9 de diciembre de 2009.
Aprobado: 16 de marzo de 2010.
Amaury Noda Albelo. Hospital Pediátrico «Eliseo "Noel" Caamaño». Calle 67 entre 300 y 302, Matanzas, Cuba.
Correo electrónico: amauryn.mtz@infomed.sld.cu