Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Obstetricia y Ginecología
versión On-line ISSN 1561-3062
Rev Cubana Obstet Ginecol v.25 n.2 Ciudad de la Habana Mayo-ago. 1999
Instituto de Neurología y Neurocirugía
Atrofia muscular espinal infantil. Introducción del diagnóstico molecular en Cuba
Lic. Ana María Acevedo López,1 Dra. Tatiana Zaldívar Vaillant,1 Dr. Felipe Moreno2y Dra. Concepción Hernández-Chico2
Resumen: La atrofia muscular espinal (AME) es un trastorno neurodegenerativo hereditario causado por la afectación selectiva de las motoneuronas del asta anterior de la médula espinal. Las atrofias musculares espinales son un importante grupo de enfermedades paralizantes que pueden afectar a los individuos de todas las edades y sexo. Forman un amplio espectro clínico genético que las distingue en 3 grupos de acuerdo con la edad de comienzo de los signos clínicos y la severidad de la enfermedad. Las 3 formas de AME autosómica recesiva fueron mapeadas en la región 5q 11.2-13.3, región que contiene múltiples copias de genes marcadores. Muchos fueron los estudios genéticos moleculares realizados para aislar el gen o genes que provocan la enfermedad, entre ellos, el análisis de ligamiento con marcadores polimórficos de la región, han permitido el estudio en familias afectadas. Precisamente este trabajo tuvo el objetivo de introducir en Cuba el diagnóstico molecular de la AME, para lo cual se evaluaron los marcadores del locus de la enfermedad: L407, M4, D5S125, D5S112, D5S127, D5S610, D5S1416, D5S107, D5S1356 y D5S557. Para el análisis de segregación alética se aplica la técnica molecular de reacción en cadena de la polimerasa (PCR), que permite identificar los cromosomas afectados a través de la obtención del genotipo en las familias diagnosticadas con los tipos clásicos I, II, III. En una familia donde el primero de 3 hijos es enfermo con el tipo III, se determinó que su hermanita de 4 años es portadora y su hermanito de 5 años es sano. Se realizó consejo genético a la familia.
Descriptores DeCS: ENFERMEDAD DE WERDNIG-HOFFMANN/genética; ENFERMEDAD DE WERDNIG-HOFFMANN/diagnóstico; CUBA.
Dentro de las enfermedades neuromusculares, los trastornos en el nivel medular suelen desarrollar un cuadro clínico donde los elementos predominantes son: debilidad muscular, atrofias musculares por denervación, disminución o pérdida de los reflejos musculares, hipotonía y en muchos casos fasciculaciones de los músculos de la lengua.1 En este grupo las AMEs son trastornos neurodegenerativos de causa genética hereditaria, producidos por la afectación selectiva de las neuronas motoras del asta anterior de la médula espinal, que pueden aparecer en edades muy tempranas de la vida y resultar fatales.
La atrofia muscular espinal de la infancia (AMEI) es considerada la segunda enfermedad autosómica recesiva fatal después de la fibrosis quística,2 con una incidencia estimada de 1/10 000 nacidos vivos, con una frecuencia de portadores3 que oscila para el tipo I entre 1/40 a 1/60. La heterogeneidad clínica de la AMEI fue reconocida lo que hizo que las interpretaciones de los resultados llevaran a varias clasificaciones. La considerable variación en la edad de comienzo y el curso clínico en diferentes pacientes, permitió la clasificación en 3 tipos de AMEI.4
La forma infantil más severa, tipo I, o enfermedad de Werdnig-Hoffmann, presenta un curso clínico más o menos homogéneo, que puede comenzar con manifestaciones antes del nacimiento o antes de los 6 primeros meses de vida, con deterioro progresivo que culmina con la muerte entre el primer y segundo años de vida.5 El tipo II, forma intermedia, con un comienzo antes de los 2 años y el tipo III o enfermedad de Kugelber-Welander, son formas clínicamente heterogéneas.6,7 En esta categoría no se incluyen los casos que comienzan entre los 17 y 55 años, llamados atrofias musculares del adulto y aquéllas con un modo de herencia ligada al cromosoma X llamadas enfermedad de Kennedy.8 Las 3 formas fueron localizadas9-12 en el mismo locus en el cromosoma 5q11.3-13.3.
Numerosos han sido los estudios genéticos moleculares realizados para localizar el gen o genes responsables de la AME, dentro de ellos el estudio de ligamiento con marcadores microsatélites polimórficos constituye un método empleado en el consejo genético en familias con AME autosómica recesiva. Muchas de estas familias deseaban tener un diagnóstico prenatal en el siguiente embarazo, que no era posible hasta el momento por no contar con alguna prueba útil para el diagnóstico. Ahora con el empleo de marcadores genéticos de la región del cromosoma 5q es posible brindar un diagnóstico a familias con al menos un niño previo afectado.
Métodos
Fueron estudiados 32 miembros de 14 familias con al menos un niño afectado, diagnosticados clínicamente como AME, confirmados por electromiografía y biopsia de músculo. Los pacientes fueron informados sobre el tipo de estudio que se realizaría que sirve de herramienta complementaria al diagnóstico clínico-genético. Se tomaron 10 mL de sangre periférica para la obtención de ADN de linfocitos utilizando el método de salting-out.13 En la caracterización de los cromosomas afectados, fueron usados 10 marcadores polimórficos del tipo CA(n) microsátelites en el genotipaje de la familia a través del análisis de segregación.14-17 Las muestras de ADN obtenidas fueron amplificadas utilizando el método de reacción en cadena de la polimerasa (PCR),18 empleando un termociclador Minicicler MJ Reseach, INC., versión 2.0, bajo condiciones específicas para cada marcador. Los resultados de los productos PCR fueron interpretados en gel de acrilamida específico para cada marcador y visualizados por tinción con plata. Las características de los marcadores utilizados en el estudio y las condiciones de PCR se presentan en las tablas 1 y 2.
| Marcadores | Locus | No. de alelos | Tamaño (pb) | PIC reportados | PIC obtenido |
| 38,3 | | | | | |
| 2ae 9.1 | | | | | |
| ET (TG/AG)n | | | | | |
| YN (CT)n | | | | | |
| Afm 114ye7 | | | | | |
| 116-CA1 | | | | | |
| 236B1-CA1 | | | | | |
| JK53-CA? | | | | | |
| Mfa27 | | | | | |
Origen: Referencias 10,14,15,16 y 17. Resultados de la investigación.
Tabla 2. Condiciones de reacción de amplificación (PCR) para los marcadores utilizados en el estudio
| Locus | Temp. | MgC12 | Tiempo de |
| annealing | (mmol/L) | corrida (min) | |
| (° C) | |||
| D5S610 | | | |
| D5S557 | | | |
| D5S125 | | | |
| D5S465 | | | |
| D5S127 | | | |
| D5S1416 | | | |
| D5S1356 | | | |
| D5S112 | | | |
| D5S107 | | | |
Resultados
Un total de 32 parientes y 14 niños afectados con AMEI identificados entre los 3 tipos clásicos I, II y III, fueron genotipados con 10 marcadores de la región del cromosoma 5q. En las figuras 1 y 2 se representan los árboles genealógicos de las familias estudiadas y los alelos obtenidos en cada marcador empleado. La figura 3 representa un gel de acrilamida donde se visualizan después de la tinción con plata, las bandas obtenidas de los resultados del producto del PCR en el sistema microsatélite D5S557.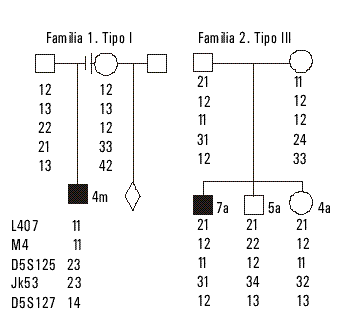
Fig. 1. Árboles genealógicos y haplotipos de las familias 1 y 2 descritas en el texto (ver resultados).
Familia 1. Forma severa tipo I (Werdnig-Hoffmann) (figura 1).
El caso índice fue diagnosticado a los 4 meses con signos clínicos característicos del tipo I: hipotonía extrema, fasciculaciones de la lengua y biopsia de músculo y electromiografía neurogénica. La evaluación con 5 marcadores polimórficos de la región del locus de la AME mostró el genotipo en la familia y permitió identificar la herencia en el niño afectado. La madre se embarazó, pero de un nuevo cónyuge no consanguíneo, por lo que el estudio molecular en el feto deja de ser útil. El niño nació y es sano.Familia 2. Forma crónica tipo III (figura 1).
El propósito presentó signos clínicos comparables con la AME tipo III, debilidad muscular y dificultades para caminar alrededor de los 7 años. La biopsia y la electromiografía fueron neurogénicas. Dos hermanos fueron también genotipados al tener riesgo de portar la enfermedad y pudo determinarse que el hermano de 5 años es sano y la hermanita de 4 años es portadora del cromosoma materno afectado.
Familias 3 y 4. Forma intermedia tipo II (figura 2).
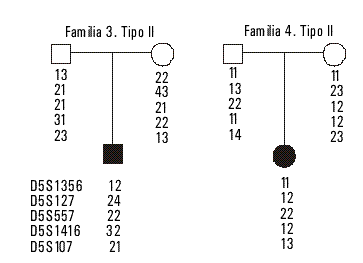
Fig. 2. Árboles genealógicos de las familias 3 y 4 descritas en el texto (ver resultados).
El caso índice en la familia 3 evaluado a los 2 años, presentó signos de hipotonía, debilidad para caminar comparable con la AME tipo II, al igual que el caso de la familia 4 diagnosticado a los 10 meses. Estas familias tienen solo un hijo y enfermo por lo que fue realizado el consejo genético.
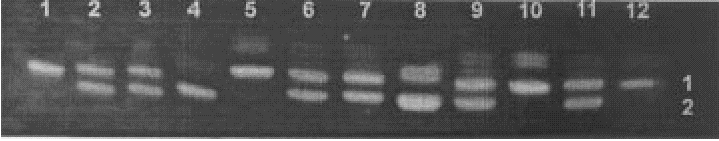
Fig. 3. Análisis de segregación en 4 familias afectadas con el marcador microsatélite D5S557 (ver texto en resultados).
De izquierda a derecha las 4 primeras carrileras corresponden a una familia donde el padre (1) y la madre (4) son homocigóticos para el alelo 1 y 2 respectivamente. El caso índice (2) y el (3), su hermano, aparentemente sano, son heterocigóticos. De 5 a 8 es otra familia que presenta un patrón de bandas similar al obtenido en la familia 1. Por el resultado que se muestra en estas familias este marcador es poco informativo ya que los padres son homocigóticos y los hermanos tienen la misma herencia alélica, pero no se pudo determinar cuál es el alelo afectado. En la tercera familia, carrileras de la 9 a la 12, resultó un patrón donde el padre (9) es heterocigótico, la madre (12) y el niño enfermo (10) son homocigóticos para el alelo 1, el segundo hijo (11) es heterocigótico y portador de la mutación. En esta familia el alelo 1 es el afectado.
Discusión
El estudio en 14 familias permitió valorar la informatividad de los marcadores en la población determinando los valores del PIC comparados con los reportados en la literatura (tabla 2). Los valores obtenidos de PIC para los 9 marcadores coinciden prácticamente con los reportados, lo que significa que en general, son informativos en nuestra población. Los marcadores microsatélites probados, proximales al gen fueron D5S125 y D5S435 y los marcadores distales al gen fueron D5S557, D5S112 y D5S127. El marcador D5S557 de 4 alelos fue el menos informativo, aunque en la poblacion cubana los más frecuentes son los alelos 1 y 2.Muchas familias portadoras de AME deseaban el estudio genético en un nuevo embarazo, que sólo se realizaba a través del consejo genético donde el riesgo de recurrencia es de 1 en 4, es decir, 25 % en cada embarazo por ser una enfermedad autosómica recesiva. Con el empleo del análisis de ligamiento utilizando marcadores polimórficos de la región del gen de la AME se brinda la posibilidad de realizar el diagnóstico molecular en las familias afectadas.
Esta experiencia en la aplicación del diagnóstico molecular de las formas de AME es una clara ventaja en la complementación del estudio genético molecular de la enfermedad.
Sin embargo, la exactitud del diagnóstico prenatal depende de la distancia genética entre los marcadores y el locus de la enfermedad y de la estructura de la familia, que hace necesario contar con una serie de marcadores óptimos cercanos al locus, teniendo en cuenta su informatividad dentro de cada familia. De esta forma es importante seguir un criterio diagnóstico estricto, basado en los estudios clínicos, electromiografías y biopsias de músculo positivas antes de realizar el consejo genético.
Cuando en las familias portadoras el niño ha fallecido, no existe sangre, músculo u otros tejidos que permitan la obtención de ADN, el estudio indirecto deja de ser útil, por ello es de gran importancia la captación de las familias con niños que presentan signos de la enfermedad desde los primeros meses de vida, fundamentalmente en los tipo I, donde la espectativa de vida es tan reducida. De esta forma resulta importante determinar el genotipo en la familia utilizando marcadores moleculares. Sin embargo, la predicción de un feto enfermo utilizando el método directo de identificación de los genes candidatos19?21 resulta un diagnóstico más rápido y exacto para el diagnóstico y brinda la posibilidad de realizar un consejo genético adecuado.
Summary: The spinal muscular atrophy (SMA) is a hereditary neurodegenerative disorder caused by selective affection of motor neurones in the anterior of the spinal cord. The spinal muscular atrophics are an important group of paralyzing diseases that may affect any individual regardless of age or sex and they make up a wide clinical genetic range classified into 3 groups interms of age of onset of clinical signs and disease severity. The 3 forms of recessive autosomal SMA were mapped in 5q 11.2-13.3 region which contains several copies of marker genes. Many molecular genetic studies such as linkage analysis with polymorphic markers of the region carried out to isolate that gene or those genes causing the disease have allowed to study the affected families. Hence this paper is aimed at introducing the SMA molecular diagnosis in Cuba for which disease locus markers are evaluated i.e, L407;M4, D5S125, D5S127, D5S610, D5S1416, D5S107, D5S1356 and D5S57. The molecular polymerase chain reaction technique is used for analyzing allele segregation, which allows to identify affected chromosomes by obtaining genotypes in families diagnosed with types 1, 2 and 3 SMA. It was determined that in a family whose eldest son had type 3 SMA, his 4 years-old sister was a SMA gene carrier whereas his 5 years-old brother was helathy. genetic counselling was given to this family.
Subject headings: ENFERMEDAD DE WERDNING-HOFFMAN/genetics;WERDNING HOFFMANN DISEASE/diagnosis; CUBA.
Referencias Bibliográficas
- Temas de neurología para la práctica médica. La Habana Editorial Ciencias Médicas, 1988;1:113-34.
- Pearn J. Classification of spinal muscular atrophies. Lancet 1980;1:919-22.
- Emery AEH. The nosology of the spinal muscular atrophies. J Med Gent 1971;8:481-95.
- Brooke MH. Disease of the motor neurons: a clinical view of neuromuscular diseases. 2 ed. London. Williams and Wilkins 1985:36-80.
- Dubowitz V. Infantile muscular atrophy: a progressive study with particular reference to a slowly progressive variety. Brain 1964;87:707-18.
- Pearn JH, Carter CO, Wilson J. The genetic identity of acute infantile spinal muscular atrophy. Brain 1973;96:463-70.
- Wohlfart G, Fex J, Eliason WS. Hereditary proximal spinal muscular atrophy: a clinical entity simulating progressive muscular dystrophy. Acta Psychiatr Neurol Scand 1955;30:395-406.
- Kennedy WB, Alter M, Sung JG. Report of an X-linked form of spinal muscular atrophy. Neurology 1968;18:671-80.
- Brzustiwicz LM, Lehner T, Castilla LH, Penchaszadeh GK, Wil helmsen KC, Daniels R. Genetic mapping of chronic childhood-onset spinal muscular atrophy locus in a region flanked by MAPIB and D5S6. Genomics 1990;13:991-8.
- Francis MJ, Morrison KE, Campell L, Grewal PK, Christodoulou Z, Daniels RJ, et al. A conting of nonchimaeric YACs containig the spinal muscular atrophy gene in 5q13. Hum Mol Genet 1993;2:1161-7.
- Soares VN, Brzustowicz LM, Kleyn PW, Knowles JA, Palmer PA, Azokan S. et al. Refinement of the spinal muscular atrophy locus to the interval between D5S435 and MAPIB. Genomic 1993;15:365-71.
- Clermont O, Burlet P, Burglen L, Lefebrve S, Pascal F, Mc Pherson J, et al. Use of genetic and physical mapping to locate the spinal muscular atrophy locus between two new highly polymorphic DNA markers. Am J Hum Genet 1994;54:687-94.
- Miller SA, Dykes DD, Polenky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res 1988;3:1215.
- Daniels RJ, Suthers GK, Morrison KE, Thomas NH, Francis MJ, Mathew CG, et al. Prenatal prediction of spinal muscular atrophy. J Med Genet 1992;29:165-70.
- Thompson TG, Morrison KE, Kleyn P, Bengtsson U, Gilliam TC, Davies KE, et al. High resolution physical map of the region surrounding the spinal muscular atrophy gene. Hum Mol Genet 1993;2:1169-76.
- Velasco E, Valero C, García E, Puente A. de la, Cruces J, San Millán JL, et al. Isolation of microsatellites from the spinal muscular atrophy (SMA) region cadidate region on chromosome 5q and linkage analysis in Spanish SMA families. Eur J Hum Genet 1995;3:96- -101.
- Melki J, Burlet P, Clemont O, Pascal F, Abdelhak S, et al. Refined linkage map of chromosome 5 in the region of the spinal muscular atrophy gene. Genomics 1993;15:521- 4.
- Saiki RK, Gelfand DH, Stoffe S, Scharf SJ, Higuchi R, Horn GT, et al. Primer-directed enzimatic amplification of DNA with a thermostable DNA polymerase. Science 1988:239-487.
- Lefebvre S, Büurglen L, Rebaullet S, Clemont, Burlet P, Viollet L, et al. Identification and characterization of SMA determining gene. Cell 1995;80:155-65.
- Roy N, Mahaderan MS, Mc Lean M, Shutler G, Yaraghi Z, Farahani R, et al. The gene for neuronal apoptosis inhibidor protein (NAIP): a novel protein with homology to baculoviral inhibitors of apoptosis is partially deleted in individual with types 1,2 and 3 spinal muscular atrophy (SMA).Cell 1995;80:167-78.
- Rodrigues NR, Oween N, Talbot K, Patel S, Muntoni F, Ignatius J, et al. Gene deletions in spinal muscular atrophy. J Med Genet 1996;33:93-6.
Lic. Ana María Acevedo López. Instituto de Neurología y Neurocirugía. Ciudad de La Habana, Cuba.
1 Laboratorio de Genética. Instituto de Neurología y Neurocirugía, Ciudad de La Habana.
2 Unidad de Genética Molecular. Hospital Ramón y Cajal, Madrid, España.














