INTRODUCCIÓN
Desde los inicios de la pandemia de la COVID-19, la vigilancia genómica del SARS-CoV-2 ha conllevado a la obtención de más de 15 millones de aislados de virus que han sido sometidos a secuenciación parcial o completa del genoma.1 Este panorama cambió durante los últimos meses de 2020 con los primeros reportes de variantes emergentes de SARS-CoV-2 asociadas con mayor transmisibilidad, gravedad de la enfermedad y escape a la inmunidad humoral.2 Aunque el genoma del SARS-CoV-2 parece relativamente estable, este tipo de virus ARN de cadena simple acumula una tasa de mutaciones estimada en torno a 10−6-10−4 por ciclo replicativo. Dichas mutaciones pueden afectar al gen que codifica el antígeno espicular (S), que interactúa con el receptor específico de la célula huésped, y seleccionan variantes mutantes con alteraciones en su capacidad infectiva, potencial patogenicidad y resistencia a los anticuerpos neutralizantes.1,2
A raíz de ello, la Organización Mundial de la Salud (OMS)3) propuso una denominación para nombrar a las variantes emergentes de SARS-CoV-2, con el empleo de letras del alfabeto griego y la nomenclatura científica según los diferentes linajes. Desde la descripción de los primeros casos de la COVID-19 en el mundo, se han descrito más de 20 variantes de SARS-CoV-2, con múltiples linajes; con predominio de una variante viral en cada pico pandémico. Los estudios4,5,6 de vigilancia genómica realizados en Cuba han detectado la presencia de múltiples variantes del SARS-CoV-2, con predominio de la cepa viral D614G, y las variantes de preocupación (VOC, siglas del inglés) Beta, Delta y Ómicron, durante las diferentes oleadas pandémicas descritas en el país.
La aparición y presencia de diversas variantes genéticas del SARS-CoV-2 ha constituido un reto para la industria biofarmacéutica cubana, encargada desde los inicios de la pandemia de la COVID-19, del desarrollo de agentes terapéuticos y vacunas. A partir del aislamiento del SARS-CoV-2 en las instalaciones de nivel de seguridad biológica 3 (NSB3) del Centro de Investigaciones Científicas de la Defensa Civil (CICDC),7 la evaluación in vitro e in vivo de la efectividad de los productos desarrollados por BioCubafarma, para enfrentar la COVID-19, frente a las diferentes variantes del SARS-CoV-2 detectadas en Cuba, constituyó un requisito durante el proceso de su aprobación y registro. Esto conllevó a la necesidad de caracterizar genéticamente los aislamientos de SARS-CoV-2, para su posterior empleo en ensayos preclínicos y clínicos de productos biofarmacéuticos.
El objetivo del presente estudio es caracterizar genéticamente los aislamientos de SARS-CoV-2 cubanos, en el periodo comprendido entre junio de 2020 y diciembre de 2022.
MÉTODOS
Se realizó un estudio de corte transversal. Se procesaron 27 cultivos virales del SARS-CoV-2 obtenidos según el procedimiento descrito por Noa y otros,7 en las instalaciones del laboratorio de NSB3 del CICDC, durante el período de abril de 2020 a diciembre de 2022.
Los cultivos virales se obtuvieron a partir del exudado nasofaríngeo de pacientes diagnosticados de la COVID-19, con un valor umbral de ciclo (Ct) de la reacción en cadena de la polimerasa en tiempo real (PCR), en el momento del diagnóstico, menor de 25.
Se realizó la extracción del ARN viral a partir del sobrenadante de los cultivos virales del SARS-CoV-2, el cual se empleó para la amplificación y secuenciación de un fragmento de 1836 pares de bases del gen S (posiciones 21976-23812), siguiendo las instrucciones descritas en el protocolo del Centro de Control y Prevención de Enfermedades (CDC).8
Las mutaciones se identificaron mediante el algoritmo de interpretación CoVsurver: Mutation Analysis of hCoV-19 (https://www.gisaid.org/epiflu-applications/covsurver-mutations-app).
La designación de las variantes de los aislamientos estudiados se asignó de acuerdo con el perfil mutacional descrito previamente, para cada variante conocida según la herramienta Audicity Instant (https://gisaid.org/database-features/audacity-instant-app/).
Se realizó un análisis filogenético mediante el empleo de las herramientas disponibles en el paquete de programas MEGA v 5.09 y se construyó un árbol filogenético según el modelo de sustitución nucleotídica T92+G+I.
Los procedimientos éticos se llevaron a cabo según las exigencias o normas del Ministerio de Salud Pública de la República de Cuba y el Ministerio de Ciencia Tecnología y Medio Ambiente (CITMA), que contempla los principios enunciados en la declaración de Helsinki.
RESULTADOS
En el estudio las variantes de SARS-CoV-2 detectadas fueron: Ómicron con sus diferentes linajes (33,3 %), Beta B 1.351 (29,6 %), otros linajes de SARS-CoV-2 (25,9 %), Alfa B 1.1.7 (7,4 %) y Delta B 1.575 (3,7 %) (tabla 1).
Tabla 1 Variantes del SARS-CoV-2 detectada en los 27 aislamientos obtenidos durante el período de junio de 2020 y diciembre de 2022
| Código de la Cepa | Código GISAID | Variante | Linaje | Clado | Año de aislamiento |
|---|---|---|---|---|---|
| DC01 | EPI_ISL_7495115 | - | B.1 | GH | 2020 |
| DC02 | EPI_ISL_7495120 | - | B.1.517 | GH | 2020 |
| DC03 | EPI_ISL_7495130 | Alfa | B.1.1.7 | GRY | 2020 |
| DC04 | EPI_ISL_7495135 | Beta | B.1.351 | GH | 2021 |
| DC05 | EPI_ISL_7495138 | Delta | B.1.575 | GK | 2021 |
| DC06 | EPI_ISL_7495141 | Beta | B.1.351 | GH | 2021 |
| DC07 | EPI_ISL_7495144 | Beta | B.1.351 | G | 2021 |
| DC08 | EPI_ISL_7495152 | Beta | B.1.351 | GH | 2021 |
| DC09 | EPI_ISL_7495159 | - | B.1.1.222 | GR | 2020 |
| DC10 | EPI_ISL_7495166 | - | B.1.1777 | GV | 2020 |
| DC11 | EPI_ISL_7495172 | - | B.1575 | G | 2021 |
| DC12 | EPI_ISL_7495177 | - | B.1.2 | GH | 2020 |
| DC13 | EPI_ISL_7495188 | Beta | B.1.351 | GH | 2021 |
| DC14 | EPI_ISL_7495197 | Beta | B.1.351 | GH | 2021 |
| DC15 | EPI_ISL_7495206 | Alfa | B.1.1.7 | GRY | 2021 |
| DC16 | EPI_ISL_7495215 | Beta | B.1.351 | GH | 2021 |
| DC17 | EPI_ISL_7495221 | - | B.1.258 | G | 2020 |
| DC18 | EPI_ISL_7495226 | Beta | B.1.351 | GH | 2021 |
| 2202 | EPI_ISL_12691753 | Ómicron | BA1.2 21K | GRA | 2022 |
| 2301 (8) | EPI_ISL_17788608 | Ómicron | BA.5.2 | GRA | 2022 |
| 2302 (12) | EPI_ISL_17788609 | Ómicron | BQ.1.13 | GRA | 2022 |
| 2303 (25) | EPI_ISL_17788610 | Ómicron | BQ.1.1 | GRA | 2022 |
| 2304 (83) | EPI_ISL_17788611 | Ómicron | BQ.1.1.23 | GRA | 2022 |
| 2305 (88) | EPI_ISL_17789272 | Ómicron | BQ.1.1.32 | GRA | 2022 |
| 2306 (C15) | EPI_ISL_17789346 | Ómicron | BQ.1.1 | GRA | 2022 |
| 2307 (C16) | EPI_ISL_17789347 | Ómicron | XBB6.1 | GRA | 2022 |
| 2308 (C17) | EPI_ISL_17789349 | Ómicron | XBB6.1 | GRA | 2022 |
Durante el período comprendido entre mayo de 2020 y enero de 2021 se aisló la variante del SARS-CoV-2, perteneciente al linaje B.1 (conocida como variante D614G), seguido de las variantes Alfa B 1.1.7 y Beta B 1.351. Entre los meses de mayo y noviembre de 2021, se aislaron las variantes Delta B.1.617.2 y otra parte de Beta B.1.351. En este tiempo se detectaron, además, virus cercanos filogenéticamente a las variantes B 1.429+B1.427 y la C36.3 (Fig. 1). Ya en diciembre de 2021 y hasta diciembre de 2022, se aislaron diversos linajes pertenecientes a la variante Ómicron (B.A 1.2.21K, BA.5.2, BQ.1.1; BQ 1.13, BQ 1.1.23, BQ1.1.32, XXB6.1).
La mutación D614G se detectó en todos los aislamientos de SARS-CoV-2 secuenciados. La variante Ómicron presentó mayor número de mutaciones en la región del genoma que codifica para la proteína S (más de 30), seguido de la variante Delta con 11 mutaciones y las variantes Alfa y Beta con 10, respectivamente.
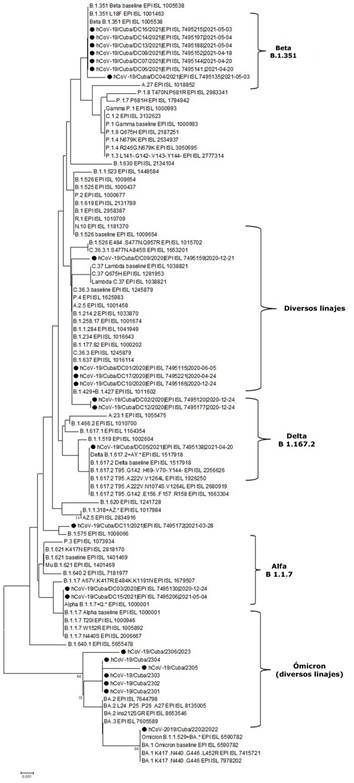
Fig. 1 Árbol filogenético de las secuencias del gen SARS-CoV-2 S de los 27 aislamientos analizados. Los aislamientos de SARS-CoV-2 cubanos se indican con el símbolo ●. El árbol se construyó por el método de máxima verosimilitud y la distancia genética se estimó según el modelo T92+G+I. Los números cerca de los nodos representan valores de bootstrap (1000 repeticiones).
DISCUSIÓN
Desde el diagnóstico de los primeros casos de la COVID-19 en Cuba, una de las principales misiones del CICDC fue la obtención de aislamientos virales de SARS-CoV-2 a partir del exudado nasofaríngeo de personas positivas a la infección y su posterior caracterización genética para la designación de la variante viral. Esto permitiría su posterior empleo en la evaluación de la efectividad de los agentes terapéuticos y vacunas contra este coronavirus, desarrollados por la industria biofarmacéutica cubana.
El primer aislamiento de SARS-CoV-2 en Cuba, obtenido en las instalaciones del laboratorio NSB3 del CICDC, correspondió a la cepa viral D614G, cuya circulación se había detectado en los estudios de vigilancia genómica realizados por el Laboratorio Nacional de Referencia de Influenza y otros Virus Respiratorios del Instituto de Medicina Tropical Pedro Kourí (IPK) durante el año 2020.4 La D614G es una mutación no sinónima implicada en la unión del ligando al receptor celular ACE-2 y se ha detectado en las variantes y linajes surgidos posteriormente, como parte de la dinámica evolutiva del SARS-CoV-2. El análisis estructural sugiere que D614G altera la conformación de unión del receptor, lo que hace más probable la unión y fusión con el ACE-2, lo que se ha asociado con una mayor virulencia, transmisibilidad y capacidad de supervivencia viral.10
Estudios de vigilancia genómica realizados en Cuba en los primeros 5 meses de 2021,2,10,11,12 detectaron la circulación de 16 variantes de SARS-CoV-2, con predominio de las variantes Alfa B.1.1.7 y Beta B.1.351, lo que coincide con los aislamientos obtenidos en ese período pandémico. Las mutaciones descritas en Alfa B.1.1.7 se han relacionado con un aumento en la transmisibilidad. Varios estudios5,13,14,15 han asociado la variante Beta B 1.351 con un aumento en la incidencia, disminución de la respuesta a los anticuerpos neutralizantes ante diferentes formatos de vacunas, así como a un posible riesgo de hospitalización, gravedad clínica y mortalidad.
El tercer período de transmisión en Cuba se produjo entre julio y septiembre de 2021, con un predominio de la variante Delta (B.1.575). Esta variante es más transmisible con estimaciones superiores al 60 % en comparación con otras descritas anteriormente. Según GISAID, el 90 % de las secuencias globales de junio a septiembre de 2021 fueron Delta. Estos valores se reflejan en los casos confirmados16 de la COVID-19 en Cuba en ese período, en el cual se detectaron 688 532 casos (6155 casos ( 100 000 habitantes), con un aumento de 57,11 veces respecto al total de casos notificados en 2020.
La variante Ómicron irrumpió en Cuba en un escenario donde Delta era la variante circulante predominante y la desplazó rápidamente, sustentada en su mayor tasa de transmisión, infectividad y escape vacunal.17,18 Las causas de dicho comportamiento se deben a que Ómicron se diferencian de otras variantes, por la presencia de numerosas mutaciones en su genoma. Hay más de 30 mutaciones en la proteína S,19 que es la encargada de reconocer las células del huésped y es el principal objetivo de las respuestas inmunitarias del organismo, y por tanto es fundamental en la infección del virus y blanco antigénico de la mayoría de las vacunas. Sin embargo, a pesar de ser más transmisible, su efecto no fue mayor, debido a la amplia cobertura de vacunación en el país con las vacunas Soberana Abdala, y a la rápida implementación de las dosis de refuerzo.6
La caracterización genética de los aislamientos virales cubanos de SARS-CoV-2 obtenidos en el laboratorio NSB3 del CICDC muestra una elevada diversidad genética, lo que permite la confección de un cepario de este coronavirus, para su empleo en la evaluación in vitro e in vivo de agentes terapéuticos y candidatos vacunales desarrollados por la industria biofarmacéutica cubana.
La caracterización molecular de los aislamientos cubanos de SARS-CoV-2 tiene una elevada diversidad genética. Posibilita evaluar in vitro e in vivo los candidatos vacunales y agentes terapéuticos desarrollados por la industria biofarmacéutica cubana.














