Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Salud Animal
versión On-line ISSN 2224-4700
Rev Salud Anim. vol.37 no.1 La Habana ene.-abr. 2015
ARTÍCULO ORIGINAL
Identificación de los genes que codifican para las proteínas VirB9, VirB10, proteína conjugal de transferencia y el factor de elongación -Tu de un aislamiento cubano de Anaplasma marginale
Identification of genes codifying for VirB9, VirB10, conjugal transfer protein and elongation factor Tu of an Anaplasma marginale Cuban isolate
Belkis Corona GonzalezI, Siomara Martínez MarreroI, Carlos Luiz MassardII, Adivaldo Henrique Da FonsecaII, Marcus Sandes PiresII, Maristela Peckle PeixotoII, Claudia Bezerra Da SilvaII, Dasiel Obregón RodríguezIII, Adrian Díaz SánchezI, Huarrisson Azevedo SantosII
ICentro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, Mayabeque, Cuba. Correo electrónico: bcorona@censa.edu.cu.
IIUniversidad Federal Rural de Rio de Janeiro, RJ, Brasil.
IIIUniversidad Agraria de la Habana (UNAH), Mayabeque, Cuba.
RESUMEN
A partir de la secuenciación del genoma completo de aislamientos de Anaplasma marginale, se ha logrado la identificación de genes que codifican para proteínas de membrana de este hemoparásito y la identificación de nuevos inmunógenos, como posibles candidatos vacunales. Sin embargo, se conoce poco acerca de la variabilidad genética de genes que codifican para proteínas de membrana de aislamientos de Anaplasma marginale. El objetivo del presente estudio fue determinar el grado de conservación en la secuencia nucleotídica de los genes que codifican para las proteínas VirB9, VirB10, la proteína conjugal de transferencia (CTP) y el factor de elongación -Tu (Ef-Tu) de un aislamiento cubano de Anaplasma marginale, así como de la secuencia deducida de aminoácidos, y compararlas con las descritas para los aislamientos Florida y Saint Maries de Anaplasma marginale y el aislamiento Israel de Anaplasma (centrale) marginale. La secuencia de los genes y la secuencia deducida de aminoácidos mostraron alto grado de identidad con las secuencias descritas para otros aislamientos de Anaplasma marginale.
Palabras clave: Anaplasma marginale, VirB9, VirB10, CTP, Ef-Tu.
ABSTRACT
From the sequencing of the entire genome of Anaplasma marginale isolates, genes encoding membrane proteins of this hemoparasite have been identified, as well as novel immunogens as potential vaccine candidates. However, little is known about the genetic variability of genes encoding membrane proteins of Anaplasma marginale isolates. The aim of this study was to determine the degree of conservation of the genes encoding the VirB9, VirB10 proteins, conjugal transfer protein (CTP) and the elongation factor ( Ef- Tu) in the nucleotide sequence of a Cuban Anaplasma marginale isolate and from the deduced amino acid sequence and compare them with those described for Florida and Saint Maries Anaplasma marginale isolates and Anaplasma (centrale) marginale from Israel . The gene and deduced amino acid sequences showed high degree of identity with the sequences described for other isolates of Anaplasma marginale.
Key words: Anaplasma marginale, VirB9, VirB10, CTP, Ef-Tu.
INTRODUCCIÓN
Anaplasma marginale es una bacteria Gram-negativa intraeritrocítica obligada que afecta al ganado bovino. La infección por este hemoparásito resulta en rickettsemia ascendente, a partir de las cuatro o cinco semanas postinfección, con fiebre, anemia, y en ocasiones la muerte. Los animales que controlan la infección permanecen persistentemente infectados durante toda su vida, y sirven como reservorio de la infección para el resto de los animales (1).
Este microorganismo presenta múltiple variabilidad antigénica, de morfología, virulencia, transmisibilidad por garrapatas y habilidad para inducir protección cruzada contra aislamientos heterólogos, con polimorfismo a nivel de genoma entre los distintos aislamientos de A. marginale. De estos, la variabilidad antigénica es uno de los factores a ser considerados en la evaluación de candidatos para el desarrollo de vacunas (2).
La inmunización de bovinos con proteínas de membrana puede inducir protección completa contra la enfermedad y la infección (3). Sin embargo, las proteínas principales de superficie, inmunodominantes, 1 a 5 (MSP1 a MSP5), las cuales incluyen las proteínas antigénicamente variables MSP2 y MSP3, no inducen protección equivalente contra la infección por Anaplasma marginale (4, 5). Debido a que estas proteínas principales de superficie no son protectivas, varios trabajos han estado dirigidos a identificar proteínas de superficie subdominantes y conservadas, incluso aquellas asociadas dentro de la membrana que pudieran ser importantes componentes de una vacuna con proteínas de membrana externa (6).
Entre los antígenos subdominantes identificados en la membrana externa están las proteínas del Sistema de Secreción Tipo 4, del inglés Type four secretion system (TFSS) (7). Varias de estas proteínas inducen respuesta de células TCD4+, como la producción de interferón gamma (IFN-g), y han sido reconocidas por IgG2 en ganado inmunizado con vacunas de membrana externa (7, 8, 9).
La estructura y función de las TFSS de rickettsias patógenas, incluyendo la familia Anaplasmataceae, aún no está bien definida. Sin embargo, la importancia de estas proteínas para estos patógenos, como un posible factor de virulencia, se infiere a partir de la retención y conservación de las proteínas entre las diferentes especies de rickettsias que han sufrido una evolución reductiva (10).
El presente trabajo tiene como objetivo amplificar y secuenciar los genes que codifican para las proteínas VirB9, VirB10, CTP y Ef-Tu de un aislamiento cubano de Anaplasma marginale.
MATERIALES Y MÉTODOS
Aislamiento de Anaplasma marginale: la extracción del ADN de A. marginale se realizó a partir de sangre de un animal esplenectomizado, experimentalmente infectado con el aislamiento Habana de Anaplasma marginale. La sangre fue lavada con PBS y conservada en 30% de glicerol a -20°C hasta su uso. El ADN se purificó utilizando el Kit Blood and Tissue DNA Purification Puregene Genta System (QIAGEN).
Genes amplificados por PCR y análisis de las secuencias: El ADN del aislamiento Habana de A. marginale se utilizó para la amplificación de los genes virB9, virB10, CTP y Ef-Tu, mediante la reacción en cadena de la polimerasa (PCR). La secuencia de los cebadores utilizados aparece en la Tabla 1.
Las reacciones de amplificación por PCR se realizaron en un volumen final de 50ml, conteniendo 50ng de ADN, 0.4mM de cada cebador y 1X GoTaq® Green Master Mix (Promega®, Madison, USA). Las condiciones de amplificación fueron las siguientes: 94°C durante 5 min, seguido por 30 ciclos de 94°C por 1 min, 55°C por 1 min, 68°C por 1 min y una extensión final de 72°C durante 10 min, en un Thermal Cycler Gene Amp PCR System 9700 (Perkin Elmer).
Los resultados del PCR se aplicaron en geles de agarosa (Sigma) al 1%, en tampón TBE 0.5 X; se corrieron a 100 Volts y 50mA, en cámara de electroforesis «Maxiphor 2012» (LKB Bromma) durante 35 minutos en tampón de corrida (TBE 0.5X) que contenía Bromuro de Etidio (0.5mg/ml). Los resultados se visualizaron en un transluminador de luz ultravioleta «Macro Vue» (Pharmacia) y se fotografiaron con cámara digital (Olympus) de 10 megapixel. Se utilizó un marcador de peso molecular de 1kb (DNA Ladder, Promega).
Los productos amplificados se purificaron con el Kit QIAquick PCR Purification Kit (QIAGEN) y se cuantificaron utilizando el Thermo Scientific NanoDrop 200 Spectophotometer.
Secuenciación de los fragmnetos amplificados: Los fragmentos amplificados fueron secuenciados utilizando el Kit BigDye Terminator (Applied Biosystems), en el equipo ABI 3130, Applied Biosystems. Para cada gen se realizaron tres reacciones de secuenciación con los mismos cebadores que se utilizaron para la amplificación de los genes. Las secuencias se ensamblaron y se generó la secuencia consenso utilizando el programa Chromaspro 1.5 y la secuencia deducida de aminoácido se obtuvo mediante el programa MEGA 4 (12). Las secuencias se depositaron en el GenBank.
Posteriormente, se compararon con las secuencias de nucleótidos y aminoacídicas disponibles en el GenBank, utilizando el programa Blastn (http://blast.ncbi.nlm.nih.gov/blastn). El alineamiento múltiple entre las secuencias se realizó en el programa CLUSTALW (http://ebi.ac.uk/Tools/clustalw2/index.html).
RESULTADOS Y DISCUSIÓN
A partir de la amplificación de los genes del aislamiento Habana de Anaplasma marginale, se obtuvieron amplicones de 840pb, 1338pb, 812pb, 1118pb, correspondientes a virB9, virB10, CTP y Ef-Tu, respectivamente (Fig. 1), lo que coincide con las tallas descritas por Vidotto et al. (13) para el gen virB9, y por Lopez et al. (7) para el resto de los genes.
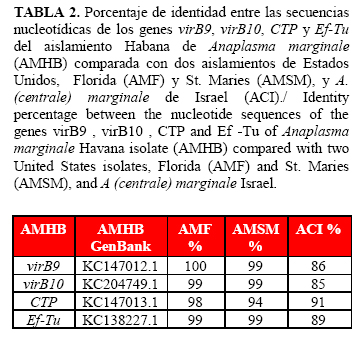
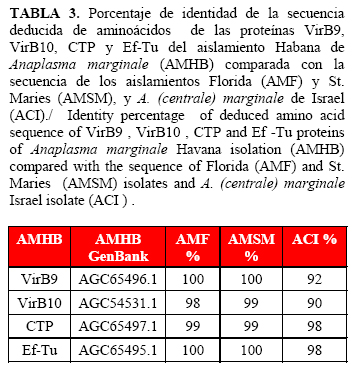
Las secuencias de los genes virB9, virB10, CTP y Ef-Tu se depositaron en el GenBank con número de Acceso: virB9: KC147012, virB10: KC204749, CTP: KC147013 y Ef-Tu: KC138227. El análisis de la secuencia nucleotídica de estos genes mostró alta identidad con la secuencia descrita para otros aislamientos de Anaplasma marginale y valores de identidad menores para la secuencias de estos genes de un aislamiento de Anaplasma (centrale) marginale (Tabla 2).
La comparación de la secuencia deducida de aminoácidos de estas proteínas del aislamiento cubano, con las secuencias de las cepas Florida y St. Maries, mostró valores entre 100 y 98% de identidad y con el aislamiento de A. (centrale) marginale mostró un 98% para las proteínas CTP y Ef-Tu y valores menores entre 90 y 92% para VirB9 y VirB10 (Tabla 3). Estos resultados coinciden con los obtenidos por Guedes-Junior et al. (11), cuando estudiaron algunas de estas proteínas.
Lopez et al. (7) compararon las secuencias deducidas de aminoácidos de VirB9, VirB10 y CTP de A. phagocytophilum, E. chaffeensis, y E. canis con las secuencias de estas proteínas del aislamiento St. Maries de A. marginale, y obtuvieron 55%, 53%, y 54% de identidad, respectivamente para VirB9; 60%, 51%, y 51% de identidad para VirB10, y 74%, 66%, y 66% de identidad para CTP. Como era de esperar, la comparación de estas proteínas entre A. marginale y A. phagocytophilum mostró el más alto grado de relación.
La secuencia deducida de aminoácidos de la proteína VirB9 del aislamiento Habana mostró 100% de identidad con esta proteína de los aislamientos Florida y St. Maries. Esto coincide con los resultados obtenidos por Lopez et al. (7), quienes encontraron 100% de identidad de VirB9 para los aislamientos Florida y St. Marie, y plantearon que el algoritmo de predicción de la topología calculado para VirB9 es similar al de una proteína de membrana externa o una proteína asociada a la superficie. Estas predicciones coinciden con la localización en la superficie de otras proteínas pertenecientes al TFSS en otras bacterias, según lo planteado por Christie et al. (14).
La conservación genética y antigénica de una proteína protectiva es importante para su eficacia como vacuna. Las proteínas VirB9, VirB10 y CTP son componentes de la fracción de membrana externa de A. marginale y son altamente conservadas, por lo que pueden ser candidatos más apropiados para vacunas que MSP2 y MSP3. Lopez et al. (7) mostraron que en bovinos inmunizados con membrana externa, estas tres proteínas produjeron significante proliferación de linfocitos TCD4+, segregación de IFN-g y producción de IgG2, respuesta inmune asociada con inmunidad protectiva (3).
Por su parte, Araújo et al. (15) y Vidotto et al. (13) sugieren la conservación de VirB9 y VirB10 entre diferentes aislamientos de A. marginale y demuestran que estas proteínas son reconocidas por anticuerpos de bovinos infectados de forma natural y experimental.
La proteína EF-Tu fue previamente clasificada como una proteína citoplasmática, pero el análisis in silico, mediante el programa TMHMM, la sugiere como una proteína anclada a la membrana externa y expuesta al ambiente extracelular (8). Esta proteína es una enzima perteneciente a la familia de las hidrolasas, involucrada en la síntesis de proteínas que promueve la elongación de la cadena durante la síntesis polipedtídica en el ribosoma.
Araújo et al. (15) y Lopez et al. (8) expresaron EF-Tu en E. coli y la proteína recombinante fue capaz de inducir una respuesta proliferativa de linfocitos T y títulos de IgG2 superiores a IgG1, demostrando su capacidad para la inmunoestimulación.
La secuencia deducida de aminoácidos de EF-Tu de un aislamiento de Brasil mostró 100% de identidad con la secuencia de aislamientos americanos y 97% de identidad con un aislamiento de A. (centrale) marginale (11). La secuencia inducida de aminoácido del aislamiento Habana mostró 100% de identidad con los aislamientos Florida y St. Maries y 98% con el aislamiento de A (centrale) marginale. Según Guedes-Junior et al. (11), esta proteína es importante como un posible antígeno candidato vacunal, ya que muestra un alto porcentaje de homología en la secuencia deducida de aminoácidos, entre diferentes aislamientos estudiados y el gen es considerado como altamente conservado. Esto corrobora los resultados obtenidos por Araújo et al. (15), quienes publicaron la conservación de este gen; además, es capaz de estimular la proliferación de linfocitos T, según Lopez et al. (8).
Las proteínas del TFSS transportan macromoléculas a través de la membrana de manera ATP dependiente y están relacionadas con el sistema de conjugación en las bacterias Gram-negativas (16). Debido a su localización en la superficie celular, a ser altamente conservadas y requeridas para la supervivencia intracelular, las proteínas del TFSS son blancos importantes de la investigación inmunológica en las bacterias Gram negativas (7). Su función en A. marginale no se ha determinado, pero en un patógeno que se encuentra bajo evolución reductiva, la retención de estos genes indica su requerimiento para la invasión y la supervivencia dentro del eritrocito y/o las células de garrapatas (10), por lo que recomendamos se continúen los estudios de evaluación de estas proteínas como posibles candidatos vacunales para A. marginale.
AGRADECIMIENTOS
Agradecemos al Proyecto CAPES/MES Cuba 089110 por el financiamiento para esta investigación.
REFERENCIAS
1. Eric LS, Norimine J, Beare PA, Heinzen RA, Lopez JE, et al. Anaplasma marginale Type IV Secretion System Proteins VirB2, VirB7, VirB11, and VirD4 Are Immunogenic Components of a Protective Bacterial Membrane Vaccine. Infect Immun. 2010;78(3):1314-1325.
2. Palmer GH, Brown WC, Rurangirwa FR. Antigenic variation in the persistence and transmission of the ehrlichia Anaplasma marginale. Microbes Infect. 2000;2:1-10.
3. Brown WC, Shkap V, Zhu D, McGuire TC, Tuo W, et al. CD4+ T-lymphocyte and immunoglobulin G2 responses in calves immunized with Anaplasma marginale outer membranes and protected against homologous challenge. Infect Immun. 1998;66:5406-5413.
4. Abbott JR, Palmer GH, Kegerreis KH, Hetrick PF, Howard CJ, et al. Rapid and long-term disappearance of CD4+ T lymphocyte responses specific for Anaplasma marginale major surface protein-2 (MSP2) in MSP2 vaccinates following challenge with live A. marginale. J Immunol. 2005;174:6702- 6715.
5. Palmer GH, Barbet AF, Davis WC, McGuire YC. Immunization with an isolate-common surface protein protects cattle against anaplasmosis. Science. 1986;231:1299-1302.
6. Kaitlyn M, Norimine J, Palmer GH, Sutten EL, Baszler TV, Brown WC. Association and evidence for linked recognition of type IV secretion system proteins VirB9-1, VirB9-2, and VirB10 in Anaplasma marginale. Infect Immun. 2001.
7. Lopez JE, Palmer GH, Brayton KA, Dark MJ, Leach SE, Brown WC. Immunogenicity of Anaplasma marginale type IV secretion system proteins in a protective outer membrane vaccine. Infect Immun. 2007;75:2333-2342.
8. Lopez JE, Siems WF, Palmer GH, Brayton KA, McGuire TC, et al. Identification of novel antigenic proteins in a complex Anaplasma marginale outer membrane immunogen by mass spectrometry and genomic mapping. Infect Immun. 2005;73:8109-8118.
9. Lopez JE, Beare PA, Heinzen RA, Norimine J, Lahmers KK, et al. High-throughput identification of T-lymphocyte antigens from Anaplasma marginale expressed using in vitro transcription and translation. J Immunol Methods. 2008;332:129-141.
10.Gillespie JJ, Ammerman NC, Dreher-Lesnick SM, Sayeedur Rahman M, Worley MJ, et al. An anomalous type IV secretion system in Rickettsia is evolutionarily conserved. PLoS One. 2009;4:e4833.
11.Guedes-Junior D, Araújo FR, Junior N, Adi SS, Cheung LM, Fragoso PS, et al. Analysis of membrane protein genes in a Brazilian isolate of Anaplasma marginale. Mem Inst Oswaldo Cruz, Rio de Janeiro, 2010;105(7):843-849.
12.Tamura K, Dudley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol. 2007;24:1596-1599.
13.Vidotto MC, Venancio EJ, Vidotto O. Cloning, sequencing, and antigenic characterization of rVirB9 of Anaplasma marginale isolated from Parana State, Brazil. Gen Mol Res. 2008;7:460-466.
14.Christie PJ, Atmakuri K, Krishnamoorthy V, Jakubowski S, Cascales E. Biogenesis, architecture, and function of bacterial type IV secretion systems. Annu Rev Microbiol. 2005;59:451-485.
15.Araújo FR, Costa CM, Ramos CAN, Farias TA, Souza IIF, Melo ESP, et al. IgG and IgG2 antibodies from cattle naturally infected with Anaplasma marginale recognize the recombinant vaccine candidate antigens VirB9, VirB10 and elongation factor-Tu. Mem Inst Oswaldo Cruz, 2008;103:186-190.
16.Niu H, Rikihisa Y, Yamaguchi M, Ohashi N. Differential expression of VirB9 and VirB6 during the life cycle of Anaplasma phagocytophilum in human leukocytes is associated with differential binding and avoidance of lysosome pathway. Cell Microbiol. 2006;8:523-534.
Recibido: 18-12-2014.
Aceptado: 9-2-2015.