Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760
Rev Cubana Med Trop vol.64 no.3 Ciudad de la Habana jul.-sep. 2012
ARTÍCULO ORIGINAL
Reacción en cadena de la polimerasa en tiempo real para la cuantificación del ADN del virus de la hepatitis B
Real-time polymerase chain reaction assay for Hepatitis B virus DNA quantification
Dra. Licel de los Ángeles Rodríguez Lay,I Dra. María Caridad Montalvo Villalba,I MSc. Susel Sariego Frómeta,I MSc. Marité Bello Corredor,I Lic. Elin Mora Laguna,II Dra. Vivian Kourí Cardellá,I MSc. Pedro Ariel Martínez Rodríguez,I Téc. Meilin Sánchez Wong,I Téc. Bárbara MarreroI
I Instituto de Medicina Tropical "Pedro Kourí". La Habana, Cuba.
II Centro Provincial de Higiene, Epidemiología y Microbiología. La Habana, Cuba.
RESUMEN
Introducción: los niveles de ADN viral en muestras de suero son un marcador útil para monitorear la progresión de la enfermedad y la respuesta al tratamiento en pacientes con hepatitis B crónica; de ahí que se comercialicen estuches diagnósticos para esta función, con la desventaja de ser costosos.
Objetivos: desarrollar y evaluar el desempeño analítico de un sistema de reacción en cadena de la polimerasa en tiempo real para la cuantificación del ADN del virus de la hepatitis B.
Métodos: se utilizaron cebadores que amplifican un fragmento del gen C y sonda de hidrólisis en el equipo LightCycler 1.5. Se construyó una curva estándar y se evaluó su eficiencia. Se utilizaron 272 muestras de suero para ensayos de especificidad analítica y clínica, especificidad y exactitud genotípica, coeficientes de variación intraensayo e interensayo, comparación con un estuche comercial y con la reacción en cadena de la polimerasa cualitativa para el virus de la hepatitis B.
Resultados: la curva estándar mostró excelente correlación lineal (r= -1) y valores muy bajos de error a lo largo de varias magnitudes de concentración de ADN diana. La especificidad analítica y clínica fue de 100 %, en tanto que al evaluar la especificidad y exactitud genotípica, se obtuvo que las diferencias entre los Log10 del valor obtenido y el de referencia eran inferiores a 0,5 Log10. El límite de detección por análisis de Probit se estimó en 16,41 UI/µL con un rango dinámico de cuantificación de hasta 108 UI/mL. El sistema mostró bajos coeficientes de variación intraensayo (0,16 a 1,45 %) e interensayo (0,9 a 2,62 %). La comparación con el estuche comercial artus HBV LC PCR kit mostró una correlación de r= 0,964 y r2= 0,929; con la reacción en cadena de la polimerasa cualitativa se confirmó la mayor sensibilidad y ventajas de la reacción en cadena de la polimerasa en tiempo real.
Conclusiones: el ensayo cumple con los requisitos para la cuantificación del ADN del virus de la hepatitis B, que demuestra ser específico, sensible y reproducible. Su aplicación permitirá un mejor diagnóstico y seguimiento de los pacientes con hepatitis B crónica en Cuba.
Palabras clave: reacción en cadena de la polimerasa en tiempo real, hepatitis B, cuantificación del ADN del virus de la hepatitis B, estandarización de sistemas diagnósticos.
ABSTRACT
Introduction: viral DNA levels in serum samples are a useful marker to monitor the disease progression and the treatment response in patients with chronic hepatitis B. Commercial kits for this purpose are available, but they are considerably expensive.
Objectives: to evaluate the analytical performance of a real-time polymerase chain reaction (RT-PCR) assay for Hepatitis B virus DNA quantification.
Methods: specific primers to the gene C and TaqMan chemistry in a LightCycler 1.5 equipment was used. A standard curve was made and evaluated. Two hundred and seventy-two serum samples were used to assess the clinical and analytical specificity, the genotypic accuracy and specificity, the intra-assay and interassay coefficients of variation and the comparison with a commercial assay and with the qualitative PCR.
Results: the standard curve showed a strong linear correlation (r= -1) and low error values in the tested target DNA concentration. Analytical and clinical specificities were 100 %. Genotype accuracy and specificity showed that the differences between the results obtained by RT-PCR assay and those of the reference assay were less than 0.5 Log10. The 95% HBV DNA detection end-point assessed by Probit analysis was 16.41 IU/µL with a dynamic range of quantification of 108 IU/mL. Intra-assay and interassay coefficients of variation ranged from 0.16 to 1.45 % and 0.9 to 2.62 % respectively. The RT-PCR assay correlated well with those from a commercial assay (r= 0.964 and r2= 0.929) and with the HBV qualitative PCR, thus confirming its better sensitivity and advantages.
Conclusions: the RT-PCR assay is well suited to monitoring HBV DNA levels showing to be sensitive, specific and reproducible. Its application in the clinical practice ensures a better diagnosis and management of patients with chronic hepatitis B in Cuba.
Key words: real-time polymerase chain reaction, hepatitis B, hepatitis B virus DNA quantification, standardization of diagnostic reagents.
INTRODUCCIÓN
A pesar de los logros alcanzados en el control y la prevención de la hepatitis B, todavía la infección crónica por el virus de la hepatitis B (VHB) impacta de forma significativa en la población mundial. Desde el punto de vista clínico, la hepatitis B aguda posee una definición de caso muy bien descrita y su diagnóstico se basa en la detección de proteínas virales y anticuerpos contra el virus que son producidos por el huésped, siendo de poco uso el diagnóstico molecular.1 Sin embargo, en la infección crónica, que ocurre aproximadamente en 10 % de los adultos, en 25 a 30 % de niños menores de 5 años que se infectan horizontalmente y en 90 % de los neonatos que adquieren tempranamente la infección, el diagnóstico molecular es una herramienta imprescindible para comprender y manejar el complejo clínico que representa esta infección crónica.2
En el estudio de las fases de la infección crónica por el VHB: inmunotolerancia, aclaramiento inmune, portador inactivo del antígeno de superficie del VHB (HBsAg) y hepatitis crónica con antígeno e negativo (HBeAg-); se precisan una sucesión de marcadores serológicos, bioquímicos, histológicos y sobre todo moleculares. En este sentido, se han empleado numerosas técnicas como la carga viral, el genotipaje, el análisis de la resistencia a las drogas antivirales y los análisis de las mutaciones, especialmente del gen C, en el diagnóstico y tratamiento de la hepatitis crónica por VHB.3
La carga viral es imprescindible en la evaluación inicial del paciente y durante el seguimiento. existe un consenso del umbral de 20 000 UI/mL (equivalente a 100 000 copias/mL) para distinguir a los portadores inactivos de las otras fases con replicación viral y en consecuencia para identificar a los no respondedores de la terapéutica.3,4
Existen varios métodos de cuantificación del genoma del VHB, en particular la reacción en cadena de la polimerasa en tiempo real (RCP-TR) últimamente ha cobrado mayor interés por el buen desempeño analítico, los bajos límites de detección, el amplio rango lineal de cuantificación y la excelente precisión. A estas ventajas se unen la rapidez, amplia variedad de plataformas de amplificación y mínimo riesgo de contaminación.3
Están disponibles diversos ensayos comerciales para cuantificar el ADN del VHB, además varios laboratorios de diagnóstico virológico han estandarizado ensayos caseros diseñando sus propios cebadores, sondas y metodologías que permiten disminuir los costos.5-9 Una alternativa ha sido la normalización de estos ensayos utilizando estándares internacionales reconocidos, que fueron creados precisamente en respuesta a la necesidad de pautar la cuantificación del genoma del VHB.4,7 En este trabajo se presenta el desarrollo y la evaluación de un sistema de RCP-TR para la detección y cuantificación del ADN del VHB.
MÉTODOS
Muestras de estudio
Se colectaron 272 muestras de suero clasificadas de la forma siguiente: 6 muestras de suero de pacientes crónicamente infectados con genoma de ADN del VHB detectable en el rango de 106 y 107 UI/mL analizados en triplicado por el estuche comercial artus HBV LC PCR kit (Qiagen, Alemania) y volumen suficiente para preparar un estándar secundario (ES); 16 muestras de suero negativas al HBsAg, anti-VHC y títulos de anti-HBs ³ 50 UI/L, para ensayos de especificidad analítica; 8 muestras de suero positivas a diferentes agentes virales que pudieran presentar reactividad cruzada o co-infectan con el VHB: virus de la hepatitis A, VHA (n= 2); virus de la hepatitis C, VHC (n= 2);virus de la inmunodeficiencia humana, VIH (n=2); y citomegalovirus, CMV (n=2), para ensayos de especificidad clínica; panel de 20 muestras de suero cuantificadas por el estuche comercial artus HBV LC PCR kit y genotipadas por secuenciación nucleotídica, de las cuales 10 correspondían al genotipo A2 y 10 al genotipo D [D1, (n=1), D3, (n=2) y D4, (n=7)] para ensayos de especificidad y exactitud genotípica; panel de 40 muestras de suero de pacientes crónicamente infectados por el VHB para el ensayo de comparación con un estuche comercial y 182 muestras de suero de pacientes HBsAg positivos que fueron enviados para el diagnóstico molecular del VHB (RCP cualitativa diagnóstica) o se encontraban en seguimiento de la terapia antiviral.
Las muestras se recibieron en el Laboratorio Nacional de Referencia de Hepatitis Virales del Instituto de Medicina Tropical "Pedro Kourí", entre 2008 y 2011, que se encontraban conservadas a - 80 ºC hasta su utilización.
Consideraciones éticas
Se utilizaron muestras de suero enviadas al laboratorio para diagnóstico molecular del VHB. No se utilizaron con otros fines y los resultados se informaron a los médicos de asistencia para la evaluación de la conducta a seguir, aplicar el tratamiento adecuado y mejorar así la calidad de vida de los pacientes estudiados.
Ensayos realizados
Reacción en cadena de la polimerasa para la detección cualitativa del ADN del virus de la hepatitis B (RCP-VHB)
Para la extracción de ADN del VHB, se utilizó el método de Proteinasa K a partir de 80 µL de suero añadidos a una mezcla de 20 µL de tampón de lisis (10 mM Tris-HCl (pH 8.0), 100 mM NaCl, 1 mM EDTA, 0,5 % SDS) y 1 µL de la enzima (20 µg/µL).10 Para la RCP se utilizaron los cebadores M3 CTGGGAGGAGTTGGGGGAGGAGATT (posición 1739 del genoma del VHB) y 3C CTAACATTGAGATTCCCGAGA (2452) que amplifican un segmento de la región preC/C. Se tomaron 10 µL del ADN previamente extraído y diluido (1:10 con agua libre de ARNasas/ADNasas), para ser dispensados en una mezcla de RCP que contenía tampón Taq 10X, MgCl2 (1,5 mM), dNTPs (0,2 mM), cebadores M3-3C (0,3 µM) enzima Taq Polimerasa (2 u) y agua libre de ARNasas/ADNasas hasta completar un volumen de 40 µL. Los parámetros del ciclaje fueron los siguientes: 94 °C por 10 min y 40 ciclos compuestos por 94 °C durante 1 min, 53 °C por 1 min, 72 °C durante 2 min y 72 °C durante 10 min. Una RCP positiva para el VHB debe mostrar una banda de 722 nucleótidos.11
Reacción en cadena de la polimerasa en tiempo real (estuche comercial artus HBV LC PCR kit, Qiagen, Alemania) para la cuantificación del genoma del virus de la hepatitis B
Para la extracción de ADN del VHB, se utilizó el método de columnas del estuche QIAamp® DNA Mini Kit (Qiagen, Alemania) a partir de 200 µL de suero siguiendo las instrucciones del fabricante.
El estuche comercial artus HBV LC PCR Kit constituye una prueba rápida para la detección del ADN del VHB usando la RCP-TR en un equipo LightCycler. La mezcla contiene reactivos y enzimas para la amplificación específica de una región del genoma del VHB de 134 pb. Este estuche posee un segundo sistema heterólogo de amplificación para identificar posible inhibición de la RCP. Con las concentraciones previamente conocidas de los estándares del estuche y sus Ct (ciclo umbral, por sus siglas en ingles threshold cycle) correspondientes se construyó una curva patrón; se interpolaron en ella los valores de los Ct de cada muestra problema y se calcularon los valores de la carga viral en UI/mL utilizando una fórmula contenida en el manual de operaciones.
Reacción en cadena de de la polimerasa en tiempo real (RCP-TR) para la cuantificación del genoma del virus de la hepatitis B
Construcción de la curva estándar: se realizaron diluciones seriadas del ES en base 10 en H2O libre de ARNasas/ADNasas desde 106 hasta 1 UI/mL, 3 réplicas de cada una. La curva estándar se construyó al relacionar el valor del Ct en el eje y, versus el logaritmo del número inicial de copias (eje x) con un intervalo de confianza de 95 %, por medio de un análisis de regresión lineal.
Para la RCP-TR a evaluar se siguió la metodología propuesta por Chen y otros en 2001, con algunas modificaciones.8 La sonda y cebadores usados están diseñados para amplificar una región altamente conservada del genoma del VHB (120 nt de la región del gen C), representada en los genotipos A-F: Cebador 1 AGTGTGGATTCGCACTCCT (posición 2269-2287), Cebador 2 GAGTTCTTCTTCTAGGGGACCTG (2387-2365) y Sonda TaqMan 6-FAM-CCAAATGCCCCTATCTTATCAACACTTCC-MGB (2303-2331) (FAM: 6-carboxy-fluoresceina, MGB: Minor Groove Binder).
Para la amplificación y detección se preparó la mezcla de reacción utilizando el juego de reactivos LightCycler Taqman Master (mezcla universal de RCP, Roche) que contiene reactivos y enzimas listos para usar en el equipo LightCycler usando sondas de hidrólisis (Taqman). Se probaron diferentes concentraciones de los cebadores y sonda (desde 0,1 hasta 0,9 µM). El programa empleado para el ciclaje fue: 95 °C por 10 min y 45 ciclos compuestos por 95 °C durante 15 s, 58 °C por 1 min y 72 °C durante 1 s. La emisión de fluorescencia se realizó en cada ciclo al final de la fase de extensión con el canal de detección de fluorescencia fijado en F1.
Límite de detección: se hicieron diluciones seriadas en base 10 del ES con agua libre de ARNasas/ADNasas hasta obtener una concentración de 1 UI/mL. Se realizaron 3 réplicas de cada una de las diluciones. Para determinar el límite de detección del ensayo al 95 percentil se tomó del total de réplicas, en cuántas de ellas se logró detectar el ADN viral.
Precisión: reproducibilidad y repetibilidad. Se determinó la media, la desviación estándar (DE) y el coeficiente de variación (CV) para cada concentración evaluada dentro de un mismo ensayo y en diferentes ensayos. El CV= (DE/Promedio) x 100.
Análisis estadístico
Para evaluar la curva patrón se tuvieron en cuenta los parámetros siguientes que son calculados automáticamente por el equipo LightCycler 1.5:
Slope (inclinación o pendiente, en español): se utiliza para evaluar la eficiencia de la reacción (para lograr una eficiencia de la curva estándar entre 1,5 y 2,2 este debe estar entre - 5,7 y - 2,9). La eficiencia de la reacción puede ser calculada por la ecuación siguiente: Eff=10(-1/slope) -1.
Error: señala las variaciones entre capilar y capilar, un valor de 0,6 corresponde a una desviación del valor x (concentración) de hasta 50 %.
Intercept (intercepción, en español): se utiliza para evaluar la sensibilidad de la RCP-TR, mientras menor sea el valor del Ct en la ecuación de regresión, mayor será la sensibilidad del sistema.
r: coeficiente de regresión, ofrece un control de la adecuada distribución lineal de la curva.
En los experimentos de validación del sistema RCP-TR se determinó el CV, coeficiente de correlación r y r2, así como la curva de regresión lineal. Se utilizó además el programa GraphPad_Prism 5 para el análisis de los resultados y confección de los gráficos. Para el cálculo del límite de detección se usó el paquete estadístico MINITAB Release 14 y el análisis probabilístico Probit, se asumió la distribución smallest extreme value.
RESULTADOS
Estandarización del sistema utilizando la mezcla universal de reacción en cadena de la polimerasa (Roche) y construcción de la curva estándar
La concentración de los cebadores sentido y antisentido se estimaron en 0,7 µM y la sonda en 0,15 µM, mientras que la mezcla universal de RCP y el agua se utilizaron en un volumen de 4 µL y 8 µL, respectivamente. Se usaron 5 µL de ADN diana para un volumen final de 20 µL en cada capilar. El tiempo estimado para obtener los resultados, una vez que se colocan los capilares en el equipo LightCycler 1.5, fue de 70 min.
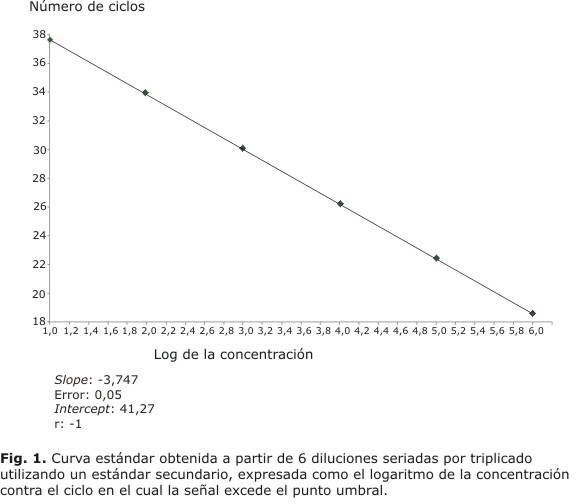
El ES utilizado fue aquel suero humano cuyo resultado obtuvo la mejor reproducibilidad en las 3 corridas realizadas con el estuche comercial, luego se procedió a la construcción de la curva estándar. Los valores medios de Ct de cada una de las réplicas se emplearon para la creación de la curva (Fig. 1).
Una vez exportada y archivada la curva estándar en el equipo, se procedió a diluir el ES en pequeñas alícuotas, tomando la dilución 103 como punto de referencia de la curva para posteriores corridas. La dilución fue conservada a - 80 ºC, con el propósito de descongelarla solo una vez antes de usarse y descartar el resto. Se estimó el rango dinámico de cuantificación de hasta 108 UI/mL.
Evaluación del sistema de reacción en cadena de la polimerasa en tiempo real
Especificidad analítica: en los 16 sueros estudiados, negativos a infección por el VHB, no se detectó presencia del ADN, por lo que la especificidad fue de 100 %.
Especificidad clínica: no se evidenció reactividad cruzada con ninguna de las 8 muestras de suero positivo a los patógenos evaluados que pudieran presentar cuadros clínicos similares al VHB (VHA y VHC), otros agentes virales con genoma ADN (CMV) y otros virus que pueden coinfectar con el VHB (VIH).
Especificidad y exactitud genotípica
De las 20 muestras de suero caracterizadas genéticamente, se pudo cuantificar el ADN del VHB en 100 % de las muestras analizadas, independientemente del genotipo del VHB. Las diferencias entre los Log10 del valor obtenido y el de referencia resultaron inferiores a 0,5 Log10 (0,215 para el genotipo A2, 0,032 para D3 y 0,202 para D4).
Sensibilidad o límite de detección de 95 %
Con el análisis de Probit se obtuvo el límite de detección al 95 percentil del ensayo en 16,41 UI/µL.
Precisión: reproducibilidad y repetibilidad
Se determinó la media, la DE y el CV para cada concentración de ADN del VHB (baja, media y alta) evaluada dentro de una misma corrida. El CV intraensayo sobre la base de los valores del Ct varió desde 0,16 a 1,45 % (tabla 1).
El CV interensayo en tres muestras de suero de altas concentraciones de ADN del VHB utilizando los resultados cuantitativos (Log UI/mL) varió entre 0,18 y 2,62 %, mientras que usando los valores medios de Ct, el CV varió entre 0,9 y 2,15 % (tabla 2).
Comparación con el estuche comercial artus HBV LC PCR kit
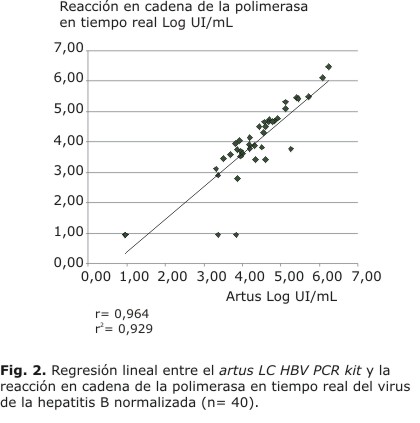
Se evaluaron 40 muestras por el estuche comercial artus HBV LC PCR kit y por la RCP-TR, una muestra (2,5 %) tuvo idéntico valor al obtenido con la referencia, 30 muestras (75 %) mostraron diferencias entre los Log10 del valor obtenido y el de referencia inferiores a 0,5 Log10, 3 (7,5 %) entre 0,5 y 1 Log10 y 6 (15 %) mayor de 1 Log10. Se construyó la curva de regresión lineal con los resultados obtenidos, con r= 0,964 y r2 = 0,929 (Fig. 2).
Comparación de la reacción en cadena de la polimerasa en tiempo real con la reacción en cadena de la polimerasa cualitativa diagnóstica
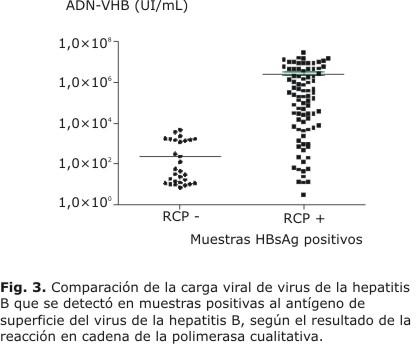
De los 182 sueros de pacientes con infección crónica por el VHB (HBsAg positivos) recibidos en el laboratorio, 65 (35,7 %) resultaron no detectables (ND) por la RCP cualitativa y no cuantificables por la RCP-TR; 29 (15,9 %) no detectables por la RCP y con niveles bajos de cuantificación (menos de 4 Log UI/mL) por la RCP-TR; 87 (47,8 %) positivos por la RCP y cuantificables por la RCP-TR; y 1 muestra (0,5 %) resultó positiva por la RCP y no cuantificable por la RCP-TR. Se confirma la mayor sensibilidad y ventajas de la RCP-TR.
En la figura 3 se exponen los niveles de ADN-VHB, en las muestras positivas al HBsAg. Las muestras se dividieron en dos grupos según los resultados de la RCP cualitativa diagnóstica.
DISCUSIÓN
La carga viral y la detección de las mutaciones relacionadas con la resistencia son dos de los aspectos más importantes a tener en cuenta para el tratamiento adecuado de la hepatitis B crónica.3 La RCP-TR se considera el estándar de oro para la cuantificación de ácidos nucleicos y dentro de ellos, la plataforma LightCycler ha demostrado resultados confiables y precisos.12,13 La RCP-TR normalizada en las condiciones del laboratorio, basándose en el protocolo propuesto por Chen y otros mantuvo las características ideales para emplearse en la práctica, con los parámetros válidos de la curva estándar construida y similares a los obtenidos por el autor: slope: -3,747 vs. -3,338, intercept: 41,27 vs. 47,07 y r: -1 vs. 0,999.8 Otros autores, con el mismo equipamiento y sondas de hibridación, obtuvieron parámetros similares en la construcción de la curva estándar (r= 1, y= 3,533, x= 42,73).14
Numerosos ensayos tipo RCP-TR normalizadas en los laboratorios diagnósticos, utilizan estándares externos para la generación de resultados cuantitativos, su exactitud es directamente proporcional a la del estándar.12,15 A su vez, la validez de un estándar se determina a través del valor obtenido con los diferentes parámetros derivados de la curva construida.16
Para evaluar la eficiencia de las RCP-TR, se utiliza el valor del slope de la curva estándar, se señala que el valor ideal de eficiencia se logra cuando el parámetro es igual a - 3,33.16,17 La sensibilidad de la RCP-TR puede calcularse también a través del valor del intercept de la curva, se recomienda que en la medida en que este sea menor, la sensibilidad es mayor.
En este estudio se obtuvo una excelente correlación lineal con un valor de error muy bajo en las seis magnitudes de concentración de ADN utilizado. En muchas aplicaciones de las RCP-TR, se postula que la eficiencia del estándar usado para construir la curva de calibración, es la misma que la eficiencia asociada con las muestras en evaluación, por lo tanto, el coeficiente de regresión es un paso importante para asegurar la calidad de los resultados.16 Por todo lo anterior consideramos que la curva estándar obtenida es confiable para ser usada en el sistema de RCP-TR propuesto.
Se ha cuestionado la utilización de curvas estándar externas, debido a que no se pueden monitorear ni controlar las variaciones inter-capilares, lo cual se minimiza con el uso de buenas prácticas de laboratorio.15 Sin embargo, otros autores han demostrado la existencia de pocas variaciones en los parámetros evaluados.7 La experiencia en el uso de equipos LightCycler ha demostrado la factibilidad de importar curvas estándares con diferentes corridas.12
Para la cuantificación del genoma del VHB existe una variedad de ensayos comerciales disponibles y de uso rutinario en los laboratorios de diagnóstico virológico. Sin embargo, algunos de ellos poseen un rango limitado de cuantificación y otros son muy costosos. Las metodologías más empleadas son las sondas de hibridación,14,18 sondas de hidrólisis o TaqMan,8,19 SYBR Green como señal de detección20 y molecular beacons.21
En estudios de especificidad de la mayoría de las técnicas de cuantificación del ADN del VHB, se han obtenido excelentes resultados, con más de 97 %, utilizando muestras seronegativas al VHB.3 El genotipo es una variable que puede influenciar la cuantificación del VHB por diferentes métodos, se ha reportado que el genotipo F no se cuantifica adecuadamente con algunos ensayos,6 sin embargo, para los sistemas de RCP-TR, no se ha observado dicho fenómeno.22 Los autores del protocolo utilizado en el presente estudio confirmaron que los cebadores y la sonda diseñados por ellos detectan los genotipos de la A a la F.8
Dependiendo de la fase clínica en que se encuentre el paciente en estudio, los niveles de ADN del VHB pueden variar, de ahí que se prefieren ensayos con un rango de cuantificación amplio, evitando realizar diluciones y re-análisis de las muestras.12,23 Una ventaja de la RCP-TR es que ofrece un mejoramiento significativo de la cuantificación de la carga viral, debido a su enorme rango dinámico que puede determinar al menos 8 log10 de ADN diana; esto es posible porque los datos son analizados de la fase linear de la curva construida, donde las condiciones son óptimas.14
El límite de detección obtenido en este estudio es de particular significado, porque permite asegurar la no existencia o supresión de la replicación viral en pacientes en tratamiento y detectar recaídas o emergencia de resistencia.
Los resultados satisfactorios en cuanto a especificidad y sensibilidad obtenidos en esta evaluación, se pueden deber además al uso de la sonda Taqman con marcaje MGB, la cual por su pequeño tamaño, unión más estable y precisión en el registro de la señal, aumenta la especificidad de las RCP-TR.14,24
Lograr una buena reproducibilidad es esencial en los ensayos de cuantificación de ácidos nucleicos. Se ha planteado que en los ensayos de RCP-TR se puede lograr un CV de 1 a 3 % para el valor del Ct.13 El sistema propuesto logró un excelente CV intraensayo e interensayo, cercanos a los obtenidos por otros autores empleando metodologías similares.8,18
La variabilidad de la cuantificación entre diferentes ensayos puede ocurrir,25 por tanto, la obtención de los resultados en UI/mL permite obtener resultados comparables y lograr un seguimiento adecuado del paciente, independientemente del ensayo utilizado para cuantificar el ADN del VHB; aunque es deseable, siempre que sea posible, que los pacientes sean monitoreados con un mismo ensayo.3 La comparación de la RCP-TR con el estuche de referencia mostró una correlación excelente y similar a las obtenidas por otros autores en la validación de ensayos con igual metodología.14,18,19,20
La RCP cualitativa diagnóstica posee una sensibilidad de 103, que muestra resultados comparables con la RCP-TR, aunque esta última resultó más sensible. Unido a esto, el ensayo propuesto permitió la obtención de resultados en menor tiempo, se pudo completar el proceso de amplificación y detección en pocos minutos y minimizar el riesgo de contaminación cruzada entre muestras. Adicionalmente, esta RCP-TR se puede utilizar en la detección de hepatitis B oculta, donde los niveles de ADN son usualmente bajos.
La RCP-TR combinada o no con los nuevos sistemas automatizados de preparación de mezclas de reacción y extracción de ácidos nucleicos de las muestras, ofrece una plataforma ideal para el desarrollo de protocolos bien optimizados, sencillos, reproducibles y rápidos para que puedan ser implementados en laboratorios de diagnóstico virológico, lo que unido a un menor costo que el de un estuche comercial, lo hace atrayente para su amplio uso.
REFERENCIAS BIBLIOGRÁFICAS
1. Horvat RT, Tegtmeier GE. Hepatitis B and D viruses. In: Murray PR, Baron EJ, Jorgensen JH, Landry ML, Pfaller MA, editors. Manual of Clinical Microbiology. 9th ed. vol. 2. Washington, DC: ASM Press; 2007. p. 1641-59.
2. Yim HJ, Lok AS. Natural history of chronic hepatitis B virus infection: what we knew in 1981 and what we know in 2005. Hepatology. 2006;43:S173-81.
3. Valsamakis A. Molecular testing in the diagnosis and management of chronic hepatitis B. Clin Microbiol Rev. 2007;20(3):426-39.
4. Saldanha J, Gerlich W, Lelie N, Dawson P, Heermann K, Heath A. An international collaborative study to establish a World Health Organization international standard for hepatitis B virus DNA nucleic acid amplification techniques. Vox Sanguinis. 2001;80:63-71.
5. Chevaliez S, Bouvier-Alias M, Laperche S, Hezode C, Pawlotsky JM. Performance of Version 2.0 of the Cobas AmpliPrep/Cobas TaqMan Real-Time PCR Assay for Hepatitis B Virus DNA Quantification. J Clin Microbiol. 2010;48(10):3641-7.
6. Laperche S, Thibault V, Bouchardeau F, Alain S, Castelain S, Gassin M, et al. Expertise of Laboratories in viral load quantification, genotyping, and precore mutant determination for hepatitis B virus in a multicenter study. J Clin Microbiol. 2006;44(10):3600-7.
7. Niesters HGM. Quantitation of viral load using real-time amplification techniques. Methods. 2001;25:419-29.
8. Chen RW, Piiparinen H, Seppanen M, Koskela P, Sarna S, Lappalainen M. Real-Time PCR for detection and quantitation of hepatitis B virus DNA. J Med Virology. 2001;65:250-6.
9. Sun S, Meng S, Zhang R, Zhang K, Wang L, Li J. Development of a new duplex real-time polymerase chain reaction assay for hepatitis B viral DNA detection. Virology J. 2011;8:227.
10. Maniatis T, Fritsch EF, Sambrook J. Molecular cloning: A laboratory manual. New York: Cold Spring Harbor Laboratory; 1982. p. 191-5.
11. Carman WF, Jacyna MR, Hadziyannis S, Karayiannis P, McGarvey MJ, Makris A, et al. Mutation preventing formation of hepatitis B e antigen in patients with chronic hepatitis B infection. Lancet. 1989;2:588-91.
12. Donald CR, Qureshi F, Malcolm J, Burns MJ, Holden MJ, Blasic JR, et al. An inter-platform repeatability study investigating real-time amplification of plasmid DNA. BMC Biotechnology. 2005;5:15.
13. Ding C, Cantor CR. Quantitative analysis of nucleic acids-the last few years of progress. J Biochem Mol Biol. 2004;37(1):1-10.
14. Ho SKN, Yam WC, Leung ETK, Wong LP, Leung JKH, Lai KN, et al. Rapid quantification of hepatitis B virus DNA by real-time PCR using fluorescent hybridization probes. J Med Microbiol. 2003;52:397-402.
15. Mackay IM, Arden KE, Mitsche A. Real-time PCR in virology. Nucleic Acids Res. 2002;30(6):1292-305.
16. Burns MJ, Nixon GJ, Foy CA, Harris N. Standardization of data from real-time quantitative PCR methods-evaluation of outliers and comparison of calibration curves. BMC Biotechnology. 2005;5:31.
17. Gulley ML, Fan H, Elmore SH. Validation of Roche LightCycler Epstein-Barr virus quantification reagents in a clinical laboratory setting. J Mol Diagn. 2008;8(5):589-97.
18. Jardi R, Rodriguez F, Buti M, Costa X, Cotrina M, Valdes A, et al. Quantitative detection of hepatitis B virus DNA in serum by a new rapid real-time fluorescence PCR assay. J Viral Hepatitis. 2001;8:465-71.
19. Sitnik R, Paes A, Mangueira CP, Pinho JRR. A real-time quantitative assay for hepatitis B DNA virus (HBV) developed to detect all HBV genotypes. Rev Inst Med Trop Sao Paulo. 2010;52(3):119-24.
20. Mendy ME, Kaye S, van der Sande M, Rayco-Solon P, Waight PA, Shipton D, et al. Application of real-time PCR to quantify hepatitis B virus DNA in chronic carriers in The Gambia. Virology J. 2006;3:23.
21. Paraskevis D, Beloukas A, Haida C, Katsoulidou A, Moschidis Z, Hatzitheodorou H, et al. Development of a new ultra sensitive real-time PCR assay (ultra sensitive RTQ-PCR) for the quantification of HBV-DNA. Virology. 2010;7:57.
22. Hochberger S, Althof D, Gallegos de Schrott R, Nachbaur N, Rock H, Leying H. Fully automated quantitation of hepatitis B virus (HBV) DNA in human plasma by the COBAS AmpliPrep/COBAS TaqMan system. J Clin Virol. 2006;35:373-80.
23. Poljak M, Marin IJ, Seme K, Brinovec V, Maticic M, Meglic- Volkar J, et al. Second-generation Hybrid capture test and Amplicor monitor test generate highly correlated hepatitis B virus DNA levels. J Virol Methods. 2001;97:165-9.
24. Watzinger F, Ebner K, Lion T. Detection and monitoring of virus infection by Real-Time PCR. Molec Aspects Med. 2006;27(2-3):254-98.
25. Germer JJ, Qutub MO, Mandrekar JN, Mitchell PS, Yao JD. Quantification of hepatitis B virus (HBV) DNA with a TaqMan HBV analyte-specific reagent following sample processing with the MagNA Pure LC instrument. J Clin Microbiol. 2006;44:1490-4.
Recibido: 27 de abril de 2012.
Aprobado: 21 de junio de 2012.
Licel de los Ángeles Rodríguez Lay. Instituto de Medicina Tropical "Pedro Kourí", Autopista Novia del Mediodía. Km 6 ½. AP 601. Marianao 13, La Habana, Cuba. Teléf.: 2553546, Telefax: 2046051. Correo electrónico: licel@ipk.sld.cu