Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760
Rev Cubana Med Trop vol.65 no.2 Ciudad de la Habana abr.-jun. 2013
ARTÍCULO ORIGINAL
Detección de ADN de leptospiras en tejidos frescos de fallecidos en Cuba, 2008-2011
Detection of leptospiral DNA in fresh tissues from dead people in Cuba, 2008-2011
MSc. Yaindrys Rodríguez Olivera, Dr. C. Islay Rodríguez González, MSc. Yarelys Zamora Martínez, Téc. José Enrique Rodríguez Silveira, Téc. Yanais Valdés Labrador, Téc. Eduardo Echevarria Pérez, Dra. C. Ana Margarita Obregón Fuentes, Dr. C. Denis Verdasquera Corcho, Dra. C. Carmen Fernández Molina
Instituto de Medicina Tropical "Pedro Kourí". La Habana, Cuba.
RESUMEN
Introducción: la leptospirosis como importante zoonosis reemergente a nivel mundial presenta una mortalidad asociada entre 15 y 25 %. El diagnóstico postmortem de esta enfermedad es laborioso. Diferentes métodos moleculares se utilizan en su confirmación, entre ellos la reacción en cadena de la polimerasa. En Cuba, desde 2008 se realiza el diagnóstico molecular de los fallecidos con sospecha de leptospirosis en el Laboratorio Nacional de Referencia de Leptospiras del Instituto de Medicina Tropical "Pedro Kourí" (LNRL-IPK).
Objetivo: diagnosticar leptospirosis a partir de tejidos frescos de fallecidos por un método molecular.
Métodos: de 50 fallecidos: 3 de 2008, 7 de 2009, 15 de 2010 y 25 de 2011, se estudiaron 171 muestras de tejidos frescos de diferentes órganos, mediante la reacción en cadena de la polimerasa simple que amplifica un fragmento del gen lipl32. De los fallecidos, 9 disponían de un suero, utilizado para los estudios serológicos.
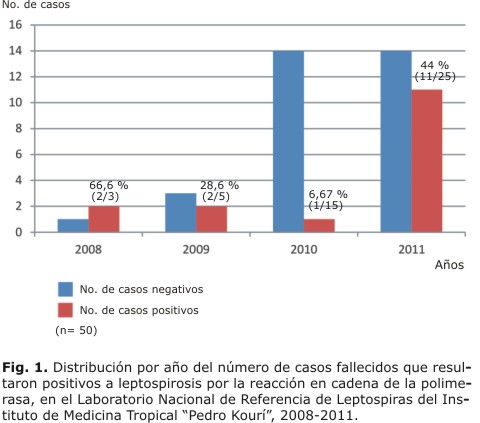
Resultados: se confirmó 32 % de los casos (16/50), correspondiendo 2 (66,7 %) a 2008, 2 (28,6 %) a 2009, 1 (6,7 %) a 2010 y 11 (44 %) a 2011. El pulmón mostró 25 % de positividad (12/48); el hígado 25,6 % (11/43); el riñón 22,5 % (9/40); el cerebro 6,3 % (1/16); el corazón 7,1 % (1/14); el bazo 12,5 %(1/8) y el músculo 50 % (1/2). Un caso resultó positivo por serología y negativo por la reacción en cadena de la polimerasa. Otros 3 casos dieron positivos por reacción en cadena de la polimerasa y negativos por serología. La concordancia global entre ambos métodos para el estudio de los sueros fue de 55,5 % (5/9).
Conclusiones: la reacción en cadena de la polimerasa, como herramienta para el diagnóstico postmortem contribuye a la confirmación en fallecidos con sospecha de leptospirosis. La serología puede usarse en estos casos para una mejor interpretación de los resultados, sin embargo, es una muestra que no se remite frecuentemente al laboratorio para su estudio.
Palabras clave: leptospirosis, diagnóstico molecular postmortem, reacción en cadena de la polimerasa, tejidos frescos.
ABSTRACT
Introduction: leptospirosis is an important re-emerging zoonosis worldwide, with an associated mortality ranging 15-25 %. Post-mortem diagnosis of the disease is painstaking. Different molecular methods for diagnosis of leptospirosis have been implemented. In Cuba, the National Reference Laboratory for Letospires of "Pedro Kourí" Institute of Tropical Medicine (LNRL-IPK) performs laboratory diagnosis of fatal cases suspected of leptospirosis.
Objective: to diagnose leptospirosis in fresh tissue samples from dead people by using a molecular method.
Methods: of 50 deceased individuals- 3 in 2008, 7 in 2009, 15 in 2010 and 25 of 2011- from different Cuban regions, 171 samples of fresh tissues of different organs were studied. A singleplex polymerase chain reaction that amplifies lipl32 gene fragment was used. Additionally, a serological diagnosis of leptospirosis was performed in 9 cases.
Results: the laboratory confirmed 32 % of cases as positive (16/50). Of these, 2 (66.7 %) in 2008, 2 (28.6 %) in 2009, 1 (6.7 %) in 2010 and 11 (44 %) in 2011. The positivity rate of the different sample was 25 % in the lung (12/48), 25.6 % in the liver (11/43), 22.5 % in the kidneys (9/40), 6.3 % in the brain (1/16), 7.1 % in the heart (1/14), 12.5 % in the spleen (1/8) and 50 % in the muscles (1/2). Of the nine cases that underwent serology, three were positive by PCR and negative by serological testing, whereas one case was found positive in the serology but negative in the PCR. The overall agreement coefficient between serology and PCR was 55.5 % (5/9).
Conclusions: polymerase chain reaction, as a molecular tool for diagnosis of leptospirosis at the LNRL-IPK, has allowed making post-mortem confirmation of that disease in Cuba. It is recommended to continue shipping serum samples from fatal cases to the LNRL-IPK for confirmation, which will allow a better interpretation of the results by using serology.
Key words: leptospirosis, post-mortem molecular diagnosis, polymerase chain reaction, fresh tissues.
INTRODUCCIÓN
Las enfermedades infecciosas zoonóticas han adquirido mayor importancia a escala global en los últimos años.1 Entre estas, la leptospirosis ha sido la más difundida, con un carácter reemergente principalmente en zonas tropicales y subtropicales. Aunque está considerada una enfermedad desatendida u olvidada, cada año se informa un número significativo de casos en diferentes regiones del planeta a causa de brotes epidémicos y casos esporádicos. Los países en vías de desarrollo aportan los mayores índices de morbilidad y mortalidad por presentar desfavorables condiciones socioeconómicas, factores culturales, carencias de estructuras sanitarias y una mayor exposición de las personas a ambientes contaminados.2,3 La mortalidad asociada se encuentra entre 15 y 25 %, principalmente debido a daño renal y hepático o severas afecciones respiratorias.
En Cuba, la leptospirosis humana tiene un carácter endemo-epidémico cíclico y estacional, enmarcado entre los meses de agosto a diciembre donde se registra la mayor morbilidad.4 Ha sido causante de brotes de importancia en provincias del centro y oriente del país,5 y se informan alrededor de 500 casos por año, con un ligero aumento en los últimos de la mortalidad y letalidad asociada a esta enfermedad.1,6
El diagnóstico de la leptospirosis resulta complejo, dado que esta enfermedad puede evolucionar con síntomas similares a los que se manifiestan en otras enfermedades infecciosas.7 Por esta razón, se requiere de datos clínicos, epidemiológicos y microbiológicos para definir un caso de leptospirosis, siendo estos últimos imprescindibles en la confirmación del caso en estudio.8
El diagnóstico microbiológico comprende la realización de métodos bacteriológicos, serológicos y moleculares, utilizados de manera conjunta para lograr un diagnóstico acertado de la infección.8,9 El cultivo y las técnicas histoquímicas permiten la detección directa de leptospiras; sin embargo, el éxito del cultivo se reduce de manera importante por ser laborioso y de muy fácil contaminación cuando se realiza a partir de fragmentos de vísceras.10,11 Por su parte, las pruebas histoquímicas constituyen herramientas a utilizar para el diagnóstico rápido postmortem, pero se consideran técnicas presuntivas.11 Los métodos serológicos por sí solos son poco efectivos en el diagnóstico de fallecidos porque generalmente no se cuenta con alguna muestra de suero de estos casos. De existir, ha sido recolectado con pocos días de evolución, lo que afecta la sensibilidad de la serología y, por lo tanto, los resultados son poco informativos.8 Por tales razones, el uso de métodos moleculares para el estudio de muestras de tejidos frescos en fallecidos con sospecha de la enfermedad, constituye una herramienta útil para la confirmación de estos casos. La técnica basada en la reacción en cadena de la polimerasa (PCR: polymerase chain reaction) ha sido una de las técnicas más ampliamente utilizadas.12,13 El Laboratorio Nacional de Referencia de Leptospiras (LNRL) del Instituto de Medicina Tropical "Pedro Kourí" (IPK), como parte de la vigilancia de la leptospirosis que se realiza en Cuba, emplea PCR simples para el estudio postmortem de los casos con sospecha de esta enfermedad infecciosa.
Este trabajo se propuso diagnosticar leptospirosis a partir de tejidos frescos de fallecidos por un método molecular, en la serie de casos de fallecidos con sospecha de esta enfermedad, recibida en el LNRL-IPK durante el período 2008-2011.
MÉTODOS
Marco de la investigación
El LNRL, perteneciente al Departamento de Bacteriología-Micología de la Vicedirección de Microbiología del IPK, sirvió de contexto al presente estudio. Este laboratorio tiene alcance internacional, con el equipamiento y personal calificado necesarios para los servicios especializados que brinda, y así enfrentar los eventos de leptospirosis humana que se presenten en Cuba.
Universo y muestras
Se estudió una serie de 50 fallecidos con sospecha de leptospirosis, de los cuales se recibieron 171 muestras de tejidos frescos de órganos durante el período 2008-2011. Las muestras procedían de los Centros Provinciales de Higiene, Epidemiología y Microbiología (CPHEM) y hospitales del país. La distribución de muestras y casos por año fue la siguiente: se estudiaron 14 muestras de 3 casos en 2008,18 muestras de 7 casos en 2009; 50 muestras de 15 casos en 2010; y 89 muestras de 25 casos en 2011 (tabla 1). Todos los fragmentos de tejidos frescos se transportaron al LNRL, en frascos de cristal de color ámbar, en solución salina 0,9 %.
Paralelamente, de 9 de los fallecidos se dispuso de un suero extraído entre el 5to. y 11no. día de evolución clínica para realizar el estudio serológico. Los sueros correspondieron a 1 caso de 2008, 2 casos de 2009, 3 casos de 2010, y 3 casos de 2011.
Métodos analíticos
Reacción en cadena de la polimerasa
Se cortaron tres fracciones pequeñas (aproximadamente de 10 mg) de cada fragmento de tejido fresco de órgano en estudio, y se les realizó extracción de ADN mediante el estuche comercial QIAamp® DNA MiniKit (Qiagen, Alemania). Se hizo una PCR simple de los extractos de ADN, usando el juego de iniciadores LipL32-270F (5´-CGCTGAAATGGGAGTTCGTATGATT-3´) y LipL32-692R (5´-CAACAGATGCAACGAAAGATCCTTT-3´) reportado por Levett y otros.14 El fragmento de interés de 423 pb se obtiene entre las posiciones 270 y 692 del gen que codifica para la proteína de membrana externa LipL32. Para la realización de la PCR se prepararon 20 µL de una mezcla de reacción que contenía: 1X- tampón PCR [10X: 2 mM- dNTP (A, T, C, G); 25 mM- MgCl2 x 6H20; 100 mM-Tris-HCl, pH 8,8; 2 mg x mL-1- BSA; 0.5 M- KCl; agua ultrapura]; 0,5 µM- solución de cada iniciador, 1,25 U-Taq Polimerasa (5 U x µL-1) (Promega, USA) y agua ultrapura (c.s.p.). A esta mezcla se le adicionó 5 µL de ADN muestra. El programa de amplificación consistió en un paso inicial a 95 ºC por 5 min, seguido de 32 ciclos de amplificación (94 ºC por 40 s, 67 ºC durante 30 s, 72 ºC por 60 s), y una extensión final a 72 ºC durante 10 min.
Como control positivo de la PCR se incluyó una muestra de ADN de la cepa de referencia Castellón 3 de L. borgpetersenii serogrupo Ballum serovar Castellonis. Como control negativo se utilizó una muestra de agua de calidad molecular.
Técnicas serológicas
Las 9 muestras de sueros se estudiaron por la técnica de microaglutinación (MAT: microagglutination test), utilizando las cepas de referencia de los siguientes serogrupos (serovares): Icterohaemorrhagiae (Copenhageni), Canicola (Canicola), Pomona (Pomona), Ballum (Castellonis), Sejroe (Hardjo), Pyrogenes (Pyrogenes), Australis (Australis), Hebdomadis (Hebdomadis) como patógenas; y Semaranga (Patoc) como no patógena. Además se empleó la técnica de hemaglutinación pasiva (HAT: hemagglutination test) con el antígeno sensibilizante de eritrocitos (AgESS) de producción nacional. Igualmente se utilizaron métodos de pesquisa de anticuerpos IgM e IgG como: el sistema comercial de aglutinación en partículas de látex LeptoTek Dri-Dot (Organon Tecknika LTD., Holanda-KIT) y un sistema de látex cubano de producción casera,7 ambos para la detección de anticuerpos totales; así como el sistema inmunocromatográfico SD Leptospira IgM-IgG (standard diagnostics, Korea) para la detección de anticuerpos IgM, IgG, o ambos.11,15
Criterios de positividad y confirmación de caso
Muestra de tejido positiva: toda réplica de tejido donde aparezca el fragmento de ADN de 423 pb, que coincida con el control positivo.
Suero positivo: suero que cumpliera al menos dos de los criterios siguientes: título por MAT mayor o igual que 160, título de mayor o igual que 80 por HAT, positivo por sistema látex y positivo o débil positivo por el sistema SD Leptospira IgM.
Caso confirmado: aquel que cumpliera con alguno de los criterios siguientes: caso con muestra de tejido positiva; caso con suero positivo; o caso con muestra de tejido positiva y suero positivo.11
Análisis estadístico
Para el análisis de los resultados se calcularon frecuencias absolutas y porcentajes. Para determinar las muestras de tejidos más útiles para el diagnóstico, se realizó una comparación de proporciones y se consideraron significativos los valores de p< 0,05, con un intervalo de confianza de 95 %. Se determinó la concordancia entre la PCR y las pruebas serológicas, utilizando el paquete estadístico Epidat 3.1.
RESULTADOS
A partir de la incorporación de métodos moleculares para el diagnóstico de la leptospirosis en el LNRL-IPK, se observó un aumento progresivo del envío de muestras de tejidos frescos de fallecidos con sospechas de la enfermedad, durante el período en estudio. En la figura 1 se muestran los porcentajes de positividad por la PCR, encontrados según el número de casos por año, donde el valor global fue de 32 % (16/50).
En la figura 2 se observan los porcentajes de muestras positivas por la PCR en los diferentes tejidos de órgano. Tomando en consideración el número de muestras estudiadas, la positividad fue mayor en los fragmentos de tejidos de pulmón, hígado y riñón; sin embargo, no se encontraron diferencias significativas en el comportamiento de la positividad entre los diferentes tipos de órgano analizados.
El 18 % (9/50) de los fallecidos contó con una muestra de suero para su estudio por técnicas serológicas, colectada alrededor de 5 a 11 días a partir de la fecha de inicio de los síntomas. De los 9 casos, 2 resultaron positivos por serología con sistema de látex cubano positivo, títulos de anticuerpos de 40 y 160 por HA, y título de anticuerpos de 2 560 por MAT frente a los serogrupos Icterohaemorrhagiae y Canicola, respectivamente.
En la tabla 2 se muestran de forma combinada, los resultados por la PCR en los tejidos de órgano y el estudio serológico en los sueros, en los 9 casos que contaron con ambos tipos de muestras. La concordancia entre estas técnicas fue de 55,5 % (5/9). El valor del índice de Kappa es de 0,0526; lo que indica una mala concordancia entre las pruebas. Tuvieron resultados serológicos negativos 3 casos positivos por PCR, mientras que un caso, cuyas muestras fueron negativas por PCR, se definió como positivo por los métodos serológicos.
DISCUSIÓN
La PCR ha sido uno de los métodos más utilizados para el diagnóstico de enfermedades infecciosas debido a su alta especificidad, entre ellas la leptospirosis. Esta técnica puede aplicarse en el diagnóstico temprano de la infección activa porque permite la detección de material genético del microorganismo en la muestra analizada, y no depende de la viabilidad de la célula de leptospira.8
La divulgación del fortalecimiento de las capacidades diagnósticas del LNRL-IPK pudo ser un factor condicionante para el aumento del número de muestras de fallecidos con sospecha de leptospirosis, recibidas para su estudio durante 2008-2011. El ligero aumento en la mortalidad y letalidad de la leptospirosis humana observado en los últimos años en Cuba, se ha relacionado al diagnóstico tardío de la enfermedad en investigaciones precedentes. Tales circunstancias imposibilitan el tratamiento antimicrobiano oportuno y contribuyen al aumento de complicaciones en estos casos fatales.1,16 Otros estudios destacan la demora del paciente en asistir a una entidad de salud, que en la mayoría de los casos media entre 2 y 6 días.4 Por otra parte, la gran similitud en la presentación clínica de la leptospirosis con otras enfermedades infecciosas que evolucionan como síndromes febriles agudos, tales como la fiebre del dengue, la influenza, el paludismo e incluso las hepatitis virales, provocan que el diagnóstico se confunda y pueda realizarse de manera incorrecta.13,15
La confirmación de los casos positivos a leptospirosis por la PCR contribuyó a la conclusión del diagnóstico postmortem de estos fallecidos. En los casos que las muestras resultaron negativas por la PCR, pudo estar cursando alguna de esas infecciones en las que habitualmente se piensa para el diagnóstico diferencial de la leptospirosis.
Por otra parte, la negatividad en las muestras de tejidos pudo deberse a resultados falsos negativos. El uso de un control positivo interno incluido en cada reacción de PCR es una herramienta necesaria para demostrar la ocurrencia o no de inhibición en la reacción.17 Sin embargo, no se utilizó durante el período que comprendió esta investigación. Asimismo, factores inherentes a la muestra como su inadecuada conservación, y específicamente en muestras de vísceras el empleo de fragmentos de tejidos donde no haya ADN bacteriano, también determinan el fallo del diagnóstico molecular. Por ello, la utilización de varios cortes del mismo fragmento de tejido en estudio contribuye a la detección de ADN diana en alguna de estas réplicas.
Pese a no encontrar diferencias significativas en la positividad hallada según el tipo de órgano estudiado, el mayor número de muestras se correspondió con órganos de marcada importancia en el desenlace fatal de los casos severos de leptospirosis.
Las muestras analizadas en esta investigación no se acompañaron en su mayoría de la información clínica necesaria, que permitiera asociar el resultado de laboratorio con los síntomas. Sin embargo, el previo conocimiento de la clínica del paciente por lo general condiciona el tipo de tejido a enviar. Las muestras de pulmón se colectan para el diagnóstico de los casos con afectaciones respiratorias y hemoptisis asociada a hemorragia pulmonar. En aquellos con ictericia, insuficiencia hepato-renal, hemorragias y complicaciones cardiovasculares, generalmente se envían tejidos de hígado, riñón y corazón. Las muestras de cerebro se colectan y remiten al laboratorio en fallecidos con complicaciones meníngeas. Estudios internacionales que incluyeron a nuestra región, reportan el fallo renal18 o hepatorrenal,16 así como la afectación pulmonar2,16,19 dentro de las principales causas de muerte de individuos con leptospirosis. Asimismo, el síndrome hemorrágico pulmonar severo se ha referido como causa de muertes asociadas a brotes epidémicos en países como Brasil.2,19
La identificación por MAT de los probables serogrupos infectantes en los dos casos positivos por serología, apunta a que algún serovar de estos serogrupos sea el causante de la infección en cada caso. Pese a los esfuerzos realizados no se lograron aislamientos de leptospiras que permitieran identificar y confirmar estos hallazgos.
Uno de los casos que resultó positivo por la PCR presentó anticuerpos frente al serogrupo Icterohaemorrhagiae, aunque ello no determina que este sea el serogrupo infectante.15 Además, entre los serogrupos de leptospiras se presenta el fenómeno de reactividad cruzada y se describe para Icterohaemorrhagiae con Canicola, Pyrogenes y Australis, todos circulantes en la región.11,20 El serovar Copenhageni/serogrupo Icterohaemorrhagiae ha sido causante de significativos brotes epidémicos en Brasil.2 En Cuba, en los últimos años se ha identificado el serovar Icterohaemorrhagiae con elevada frecuencia en los aislamientos tipificados (informes de LNRL-IPK) provenientes de la región oriental, de donde procedía este fallecido.
El hallazgo de las muestras negativas por la PCR en el otro caso confirmado por serología con elevados títulos de anticuerpos frente al serogrupo Canicola, pudo deberse a factores que pueden afectar la PCR, como el gen diana.
Respecto a la secuencia diana, son recomendables aquellas que se encuentren en muchas copias en el genoma para no comprometer la sensibilidad de la PCR.21 Sin embargo, la secuencia que se amplifica en esta PCR pertenece al gen de la lipoproteína estructural y funcional de membrana externa LipL32.22 Levett y otros en el 200514 reportaron que el gen lipl32 está en una sola copia en el cromosoma I de L. interrogans sensu lato. Pese a ello, la presencia de este gen solo en las leptospiras patógenas favorece la especificidad de la PCR respecto a otras utilizadas anteriormente,23 cuyas secuencias diana detectaban serovares de leptospiras tanto patógenas como no patógenas.24
En 3 casos positivos por el método molecular los resultados serológicos fueron negativos. Ello pudo relacionarse con que la única muestra de suero que existió en cada caso correspondía a la primera semana de evolución clínica de la enfermedad. En este período, los niveles de anticuerpos IgM suelen ser bajos o no detectables, por lo que la utilización de una única muestra de suero reduce la fiabilidad de la serología.
El diagnóstico de la leptospirosis humana aún se sostiene en el uso de técnicas serológicas que detecten anticuerpos específicos IgM o IgG frente a Leptospira spp. No obstante, su éxito dependerá en gran medida de contar con muestras de suero pareadas para demostrar la seroconversión o el aumento de los títulos de anticuerpos entre ambos. Por otra parte, el tratamiento precoz con antibióticos también puede disminuir la respuesta de anticuerpos.1,3,16
Uno de los principales retos en el diagnóstico de la leptospirosis humana es establecer un sistema que ofrezca confiabilidad y certeza cuando la MAT u otros sistemas serológicos ofrecen resultados negativos, o cuando no existe posibilidad de aplicarlos en las muestras que estén disponibles. Estudios precedentes han reportado las ventajas que ofrece el uso combinado de los sistemas serológicos y la PCR en el diagnóstico de esta enfermedad infecciosa, siempre que se cuente con las muestras adecuadas.25 Especialmente la PCR es una herramienta eficaz durante la primera fase de la enfermedad cuando los métodos serológicos fallan. Con su empleo, se logra la confirmación temprana en pacientes con sospecha de leptospirosis, de manera que pueda distinguirse de otros síndromes febriles,26 o realizar la confirmación postmortem en fallecidos sin una impresión diagnóstica definida.
Se recomienda el envío de sueros u otras muestras útiles de los fallecidos para su estudio en LNRL-IPK, junto con las muestras de tejidos frescos, y así contribuir a una realización integral del diagnóstico y, por tanto, la confirmación (o no) de casos de leptospirosis humana.
El presente trabajo permitió la confirmación de fallecidos por leptospirosis en Cuba mediante la detección rápida de ADN de leptospiras en muestras de tejidos frescos. La aplicación del diagnóstico molecular de esta enfermedad infecciosa en el LNRL-IPK, fortalece las capacidades del Instituto "Pedro Kourí", así como del sistema nacional de salud, en su premisa de brindar respuesta oportuna frente a enfermedades infecciosas de importancia en el país.
REFERENCIAS BIBLIOGRÁFICAS
1. Berdasquera D, Barreras BA, Barroso J, Pérez A, Fernández C, Ortega LM, et al. Mortalidad por leptospirosis humana. Ciudad de La Habana, 2005-2006. Rev Panam Infectol. 2009;11(4):19-26.
2. Ko AI, Reis MG, Dourado CMR, Johnson WD, Riley LW, et al. Urban epidemic of severe leptospirosis in Brasil. Lancet. 1999;354:820-5.
3. Roca B. Leptospirosis. Rev Med Univ Navarra. 2006;50(2):3-6 [citado 22 Jul 2012]. Disponible en: http://www.ncbi.nlm.nih.gov/pubmed/16999233
4. Verdasquera D, Barroso J, Barreras BA, Pérez A, Pérez K, Obregón AM, et al. Factores asociados a la morbilidad por Leptospirosis humana. Ciudad de La Habana, 2005-2006. Rev Panam Infectol. 2010;12(1):8-16.
5. Verdasquera D, Ortega LM, Fernández C, Obregón AM, Rodríguez I, Miyar R. Enfrentamiento a brotes epidémicos de leptospirosis humana. Rev Panam Infectol. 2011;13(1):28-35.
6. Verdasquera D, Galí L, Sánchez L, Alpízar D, Vega B, Calas V. Variación de la morbilidad y mortalidad por leptospirosis humana. Cuba, 1998-2007. Rev Panam Infectol. 2010;12(4):22-30.
7. Obregón AM, Fernández C, Rodríguez I, Balbis Y, Martínez B, Rodríguez J. Sistema de aglutinación con látex para el diagnóstico rápido de la leptospirosis en Cuba. Rev Panam Salud Publica. 2004;16(4):259-65.
8. Levett P. Leptospirosis. Clin Microbiol Rev. 2001;14(2):296-320.
9. Dassanayake DL, Wimalaratna H, Agampodi SB, Liyanapathirana VC, Piyarathna TA, Goonapienuwala BL. Evaluation of surveillance case definition in the diagnosis of leptospirosis, using the Microscopic Agglutination Test: a validation study. BMC Infect Dis. 2009;9:48.
10. Rodríguez I, Fernández C, Obregón AM, Zamora Y, Rodríguez JE, Rodríguez NM, et al. Confirmación microbiológica de 2 brotes emergentes de leptospirosis humana en Cuba. Rev Cubana Med Trop. 2007;59(1):19-23.
11. Fernández C, Obregón AM, Rodríguez I, Rodríguez Y, Mondeja BA, Noda AA, et al. Manual de operaciones y procederes para el diagnóstico de la leptospirosis humana. La Habana/ Panamá: Ediciones DAMPSA; 2012. p. 24,25,32,33,59,61,64, 65.
12. Brown PD, Gravekamp C, Carrington DG, van de Kemp H, Hartskeerl RA, Edwards CN, et al. Evaluation of the polymerase chain reaction for early diagnosis of leptospirosis. J Med Microbiol. 2006;43(3):110-14.
13. Adler B, de la Peña-Moctezuma A. Leptospira y leptospirosis. Vet Microbiol. 2010;140:287-96.
14. Levett P, Morey RE, Galloway RL, Turner DE, Steigerwalt AG, Mayer LW. Detection of pathogenic leptospires by real-time quantitative PCR. J Med Microbiol. 2005;54:45-9.
15. WHO-ILS. Human leptospirosis: guidance for diagnosis, surveillance and control. Malta: WHO; 2003. p. 5-7, 43, 63, 64, 77.
16. Verdasquera D, Ortega LM. Leptospirosis humana: pautas para su prevención y control. Ediciones DAMPSA: La Habana/Panamá; 2012. p. 49-57, 63.
17. Bacich DJ, Sobek KM, Cummings JL, Atwood AA, O´Keefe D. False negative results from using common PCR reagents. BMC Research Notes. 2011;4:457.
18. Daher E, Trevisan DM, Borges M, Abdulkader RCRM. Risk factors for death and changing patterns in leptospirosis acute renal failure. Am J Trop Med Hyg. 1999;61(4):630-4.
19. Gouveia E, Metcalfe J, de Carvalho AL, Aires TS, Villasboas-Bisneto JC, Queirroz A, et al. Leptospirosis-associated severe pulmonary hemorrhagic syndrome, Salvador, Brazil. Emerg Infec Dis. 2008;14(3):505-8.
20. Rodríguez I, Obregón AM, Rodríguez JE, Fernández C, Arzola A, Victoria B. Caracterización serológica de cepas aisladas de pacientes con leptospirosis humana en Cuba. Rev Cubana Hig Epidemiol. 2002;40(1):11-5.
21. Coyne VE, James MD, Reid SJ, Rybicki EP. Molecular Biology Techniques Manual. 3rd ed. University of Cape Town; 2003 [citado 22 Jul 2012]. Disponible en: http://www.mcb.uct.ac.za/manual//MolBiolManual.htm
22. Cullen PA, Xu X, Matsunaga J, Sanchez Y, Ko AI, Haake DA. Surfaceome of Leptospira spp. Infect Immun. 2005;73:4853-63.
23. Gravekamp C, van de Kemp H, Franzen M, Carrington D, Schoone GJ, van Eys G, et al. Detection of seven species of pathogenic leptospires by PCR using two sets of primers. J Gen Microbiol. 1993;139:1691-700.
24. Haake DA, Chao G, Zuerner RL, Barnett JK, Barnett D, Mazel M, et al. The leptospiral major outer membrane protein LipL32 is a lipoprotein expressed during mammalian infection. Infect Immun. 2000;68(4):2276-85.
25. Brown PD, Carrington DG, Gravekamp C, van de Kemp H, Edwards CN, Jones SR, et al. Direct detection of leptospiral material in human postmortem samples. Research Microbiology. 2003;154:581-6.
26. de Abreu C, Teixeira VL, Caló E, Spinosa C, Arroyo MC, da Silva MV, et. al. Polymerase chain reaction in comparison with serological tests for early diagnosis of human leptospirosis. Trop Med Int Health. 2006;2(2):1699-707.
Recibido: 6 de agosto de 2012.
Aprobado: 8 de febrero de 2013.
Yaindrys Rodríguez Olivera. Instituto de Medicina Tropical "Pedro Kourí". Autopista Novia del Mediodía, km 6 ½. AP 601, Marianao 13, La Habana, Cuba. La Habana. Cuba. Correo electrónico: yaindrys@ipk.sld.cu