Introducción
La microbiota normal se considera la primera línea de defensa contra agentes patógenos, y normalmente se encuentra en un equilibrio dinámico con el huésped, pero cuando hay una disbiosis microbiana1,2 aumenta el riesgo de desarrollar problemas de salud, incluso de desarrollar cáncer.3
Cada año las cifras del cáncer de mama aumentan, Globocan publicó que para el año 2018 fue la principal causa de muerte en mujeres (15 % anual), y para el 2020 una incidencia de 24,5 % y 16 % de muertes.4,5
Es importante destacar que las diferencias en la microbiota de las mujeres desempañan un papel fundamental en el desarrollo de esta enfermedad;6) el análisis microbiológico tanto de la leche materna como del tejido mamario muestra que la mama aloja microbiota única y diversa,7,8 una parte de esta microbiota podría provenir de bacterias derivadas del intestino. Jost y otros9) plantearon la presencia de un eje intestino-seno a lo largo del cual las bacterias presentes en el intestino materno pueden alcanzar la glándula mamaria a través de una ruta endógena que involucra células dendríticas.10,11,12
Sin embargo, a pesar de los hallazgos reportados y la amplia información que existe, los autores no han sido concluyentes con respecto a que una bacteria sea causante del cáncer de mama, pero sí han afirmado la presencia de diferentes especies que se relacionan de forma benéfica o perjudicial.13
Esta investigación se hizo por medio de un análisis microbiológico y metagenómico con la finalidad de conocer la microbiota presente en nódulos mamario, y el comportamiento morfológico (forma, tamaño y membrana) que experimenta la línea celular de cáncer de mama MCF-7 al ser infectada con una de las bacterias identificadas.
En la mayoría de las publicaciones las muestras provienen de mastectomía y lumpectomía, y hay muy pocos trabajos a partir de biopsias mamarias.7,10,13,14,15,16,17,18 Estos estudios han sido posibles con el uso de tecnologías de secuenciación de nueva generación.19
Métodos
Se realizó un estudio exploratorio experimental de una población de 57 mujeres con nódulos mamarios, pero sin síntomas clínicos de infección, intervenidas para biopsia por punción en CEDIUL S.A.S. Esta muestra fue tomada por conveniencia en una ventana de observación de 9 meses, y solo 17 muestras fueron escogidas al azar por medio de un muestreo no probabilístico para el análisis metagenómico. Para cultivo bacteriano se analizó la totalidad.
Como criterios de inclusión se tuvo en cuenta a mujeres mayores de 18 años, con presencia de nódulo mamario. Se excluyó a las pacientes expuestas a tratamiento con antibiótico inferior a un mes, presencia de lesión líquida, tratamiento de quimioterapia o radioterapia.
Después se procedió al diligenciamiento de una encuesta de datos personales y clínicos, y a la firma del consentimiento informado (aprobados por el comité científico y de ética de la Universidad Metropolitana y CEDIUL).
En el procedimiento estadístico se realizó una tabla de frecuencia y un análisis de regresión logística.
Toma de muestras:
Controles: En la piel y productos de asepsias se tomó con hisopo estéril, para control ambiental, y se empleó una caja de agar sangre abierta en el área de radiología.
Tejido mamario: Se tomó de la primera punción, y bajo condiciones de esterilidad. Se conservó una parte con medio tioglicolato y la otra en tubo de microcentrífuga a -20 °C.
Evaluación de la calidad del ADN total bacteriano: Para la extracción de ADN se utilizó QIAamp PowerFecal ADN (Quiagen) bajo cabina de bioseguridad y recomendaciones del fabricante.20 Luego se cuantificó mediante espectrofotometría a 230; 260 y 280 nm.21 Las relaciones de absorbancias (260/280-260/230) indican la presencia de diferentes contaminantes, ARN y proteínas, fenol y sales, y polisacáridos, respectivamente.22
Amplificación: se emplearon los cebadores:23
Condiciones de PCR: 1 U de la enzima Taq Polimerasa (Thermo), buffer ADN polimerasa (1X), dNTP’s (200 µM), H2O DEPC, iniciadores (0,4 µM) y aproximadamente 50 ng de ADN plantilla, todo a un volumen final de 20 µL, un ciclo de desnaturalización inicial a 94ºC durante 5 min, 25 ciclos de 94 ºC por 5 s, 55 ºC por 1 min, y una extensión final a 72 ºC por 10 min (termociclador Biorad).
Construcción de librería y análisis informático: Se inició con la amplificación de la región V3-V4 (341F-805R) del ARN ribosomal 16S, seguido de una construcción de la librería Herculase II Fusion ADN polymerase Nextera XT, Paired-end (la polimerasa es: KAPA HiFi HotStart ADN Polymerase). Luego se hizo una secuenciación usando la plataforma Miseq de Illumina (la longitud de las lecturas es de aproximadamente 300 bp 100 000 lecturas por muestra), ensamblaje de las lecturas por medio del programa FLASH (Fast Adjustment of Short reads), y preprocesamiento, seguido de la filtración de las lecturas, y corte de las secuencias de los adaptadores. Por último, se realizó la asignación taxonómica usando el programa QIIME2.24
Se usan las secuencias representativas de cada OTU para asignar la taxonomía (a nivel de especie).
Cebadores:
Identificación bacteriana: Inicialmente se realizó una siembra en estría por agotamiento en agar sangre, chocolate y MacConkey, seguido por la lectura y respectiva identificación en el equipo Phoenix 100 (Becton Dickinson).25 Por último, se congelaron a -20°C hasta el momento de la infección celular.26
Estandarización de las MCF-7: Para iniciar el cultivo celular in vitro, se descongelaron las células en Baño María a 37 ºC, y se siguió el procedimiento estandarizado.27
Determinación de la curva de crecimiento y viabilidad celular (MTT): Se realizó para definir la densidad del cultivo, el porcentaje de viabilidad y el día que el cultivo de MCF-7 alcanzaba la fase exponencial para la infección, es decir, el día que las células están metabólicamente activas.
Estandarización del cultivo bacteriano: Se realizó según Jasón Letoumeau.28 Se tuvo en cuenta que la multiplicidad de infección (MOI) puede variar entre 100:1 y 1:10.29 Una MOI muy superior o muy baja produce mayor variabilidad y las bacterias tienden a pegarse al fondo de la caja, por lo tanto, se inició en una MOI 1:1 que representa una probabilidad de infección del 63,2 %,30 y las células MCF-7 permanecieron constantes a 800 000 (Tabla 1).
Se emplearon tiempos de infección de 4 h y 24 h con S. aureus, y visualización en el microscopio de campo invertido: no se observó cambio en su morfología y la cantidad permaneció estable. Por tal motivo no se realizó el ensayo de viabilidad por MTT y se descartó este tiempo para la infección.31 Entonces se procedió a la infección, la bacteria se encontraba a -20°C, y para descongelarla se hizo un raspado en la parte superior del tubo con un asa estéril, se sembró directamente sobre la caja de agar sangre y se esperó de 24 a 48 h para el crecimiento óptimo.
Las lecturas del MTT se realizaron a 570 nm porque el formazán es un compuesto altamente cromógeno y la medición de su absorbancia produce resultados reproducibles a esta longitud de onda.32,33
Tabla 1 Estandarización del cultivo bacteriano30
| MOI | N.o de bacterias | Probabilidad de infección (%) | µL bacterias (MacFarland) |
|---|---|---|---|
| 1:1 | 800 000 | 63,2 | 8 |
| 2:1 | 1 600 000 | 86,5 | 16 |
| 3:1 | 2 400 000 | 95 | 24 |
| 4:1 | 2 800 000 | 98,2 | 28 |
| 5:1 | 3 200 000 | 99,3 | 32 |
| 6:1 | 4 000 000 | 99,8 | 40 |
| 7:1 | 4 800 000 | 100 | 48 |
| 8:1 | 5 600 000 | 100 | 56 |
| 9:1 | 6 400 000 | 100 | 64 |
| 10:1 | 7 200 000 | 100 | 72 |
Infección celular: Ya estandarizados ambos cultivos, se sembraron las células MCF-7 con condiciones de crecimiento favorable hasta lograr una confluencia del 80 %; luego se infectaron (Tabla 2). Después de 48 h, se visualizaron las características morfológicas por microscopio invertido.
Tabla 2 Relación de células-bacterias en la infección
| MOI | N. de MCF-7 | µL bacterias (MacFarland) | Probabilidad de infección (%) |
|---|---|---|---|
| 1:1 | 350 000 | 3,5 | 63,2 |
| 5:1 | 1 750 000 | 17,5 | 99,3 |
| 10:1 | 3 500 000 | 35 | 100 |
Análisis estadístico: Se tabularon los datos obtenidos en la encuesta, se aplicó el análisis estadístico usando Stapgraphics. Se calculó el porcentaje de crecimiento bacteriano total y se estableció el modelo de regresión logística para explicar cómo se modifica la variable dependiente, cuando cambian las independientes.
Resultados
Extracción del ADN total bacteriano: Se obtuvo un ADN poco concentrado que cumplió los parámetros de pureza (Tabla 3). Cabe resaltar que estas muestras incluyen ADN humano (tejido mamario) y bacteriano.
Con la PCR se obtuvo un fragmento de aproximadamente 240 pb del ADN ribosomal 16S para las 17 muestras analizadas.
Tabla 3 Concentración y pureza del ADN bacteriano
| N.o muestra | Cuantificación (ng/µL) | 260/280 | 260/230 |
|---|---|---|---|
| 1 | 21,2 | 1,8 | 1,9 |
| 2 | 53,1 | 1,8 | 1,9 |
| 3 | 1,9 | 1,9 | 1,8 |
| 4 | 7 | 1,8 | 1,7 |
| 5 | 16,2 | 1,7 | 1,7 |
| 6 | 7,8 | 1,7 | 1,8 |
| 7 | 8,2 | 1,7 | 1,8 |
| 8 | 11 | 1,7 | 1,9 |
| 9 | 12,1 | 1,8 | 1,9 |
| 10 | 94,7 | 1,9 | 1,7 |
| 11 | 11,7 | 1,9 | 1,7 |
| 12 | 11,4 | 1,8 | 1,7 |
| 13 | 4,3 | 1,7 | 1,9 |
| 14 | 7,4 | 1,7 | 1,8 |
| 15 | 6,4 | 1,6 | 1,8 |
| 16 | 20 | 1,7 | 1,7 |
| 17 | 9,4 | 1,8 | 1,7 |
Bacterioma a nivel de Phylum: A pesar de la baja cantidad del ADN obtenido en la extracción, se obtuvo una amplia diversidad bacteriana en cada muestra, el phylum más predominante fue Proteobacteria (47,0 %) y Firmicutes (26,4 %) y en menor proporción Bacteriodete (9,8 %) y Actinobacteria (8,7 %).
Bacterioma a nivel de género: Entre los géneros se encuentran de mayor a menor abundancia: Escherichia 9,4 %, Yokenella 8,2 %, Streptococcus 5,9 %, Staphylococcus 5,2 %, Prevotella 5 %, Methylobacterium 3,3 %, Pseudomonas 2,6 %, Burkholderia 2,6 %, Ralstonia 2,6 %, Acinetobacter 1,9 % y Bacteroides 1,7 %, entre otros.
Estas especies se asocian en proporciones similares en ambos grupos de pacientes, nódulos malignos y benignos, indicando que no hay diferencias significativas en ambos grupos.
Identificación microbiológica: En la tinción de Gram se observó para la mayoría de las muestras cocos en racimos y en tétradas, lo que indicó la presencia del género Staphylococcus en mayor abundancia.
La mayoría de los aislamientos crecieron en agar sangre y chocolate, a la vez, colonias de forma y aspecto diferentes; se hicieron repiques y posteriormente la identificación de las colonias ya purificadas (Tabla 4). En controles de piel, productos de asepsia y ambiental no se obtuvo crecimiento.
Solo dos pacientes resultaron con patología positiva para cáncer, entre ellas la bacteria común fue el S. saprophyticus, por lo tanto, fue la escogida para la infección celular.
Tabla 4 Identificación bacteriana por Phoenix 100
| No. muestra | Patología | Bacteria |
|---|---|---|
| 7 | Maligno | |
| 24 | Benigno | |
| 25 | Benigno | |
| 28 | Benigno | |
| 33 | Benigno | |
| 35 | Maligno | |
| 37 | Benigno |
Curva de crecimiento MCF-7 por ensayo de viabilidad MTT: La fase exponencial se determinó para el día quinto (Fig. 1), por lo tanto, se realizaron las infecciones este día.
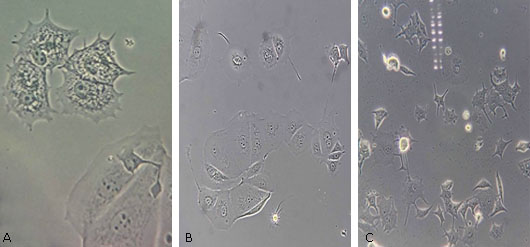
Cambios morfológicos luego de la infección: En la infección se obtuvo que a una MOI de 1:1 no se evidenciaron cambios en su morfología, y fueron muy parecidas a las células control MCF-7 (Fig. 2A).
A una MOI 5:1 se observa un comportamiento diferente, presencia de filopodias (prolongaciones delgadas de la membrana, altamente dinámicas que se extienden desde el borde de la célula), las células aumentaron en número y tamaño, crecían muy juntas con forma redonda y con núcleos contraídos (Fig. 2B).
A una MOI de 10:1 los cambios fueron más pronunciados, las filopodias crecieron en la mayoría de las células, deformadas completamente, no se observó el contenido celular al interior de esta, ausencia de membrana celular, células separadas unas de otras por la muerte que experimentaban poco a poco a causa de la concentración de la bacteria (Fig. 2C).
Análisis estadístico: De las 57 muestras tomadas, solo 7 pacientes dieron resultado positivo 12,28 % y en el resto (50) no hubo crecimiento bacteriano, lo que representó el 87,72 % del valor total.
No fue posible generar un modelo de regresión logística que asociara las variables de estudio con S. saprophyticus, porque ninguna de las variables independientes mostró una relación estadísticamente significativa, presentando valores de p mayores que 0,05 (Tabla 5).
Tabla 5 Pruebas de razón de verosimilitud
| Factor | Chi cuadrado | Gl | Valor p |
|---|---|---|---|
| Edad | 0,254545 | 1 | 0,6139 |
| Peso (kg) | 0,681772 | 1 | 0,4090 |
| Antecedentes de cáncer | 2,79792 | 1 | 0,0944 |
| Resultado | 0,542136 | 1 | 0,4615 |
Variable dependiente: S. saprophyticus.
Factores: edad, peso (kg), antecedentes de cáncer, resultado de la patología.
Discusión
Entre los resultados obtenidos en este trabajo radican en que el phylum más predominante fue Proteobacteria (47 %), seguido de Firmicutes (26,4 %) y en menor proporción Bacteriodete (9,8 %) y Actinobacteria (8,7 %), resultado que concuerda con Xuan7 y Urbaniak,16) donde describieron una comunidad caracterizada por el phylum Proteobacteria y Firmicutes, mientras que Hieken15 encontró un predominio de Bacteroidetes y muy poco Proteobacteria.
Urbaniak6) en el 2014 publicó Proteobacterias y Firmicutes como los phylum más abundantes en el tejido mamario en mujeres de Canadá e Irlanda, con y sin cáncer. Asimismo, Srikantha Thyagarajan en el año 2020, encontró los mismos phylum, y fue Proteobacteria el más abundante.34
Nuestros resultados coincidieron con que hubo mayor similitud que diferencia entre la microbiota de los pacientes, resultado que concuerda con lo reportado por Lara Costantini18 y Shen Meng17) con la del género Ralstonia y Methylobacterium de la comunidad Mediterránea, y el género Methylobacterium y Bacteriodete de la población china, respectivamente. Estos autores caracterizaron la microbiota del tejido mamario con muestras extraídas por biopsia, técnica igual a la usada para este proyecto.
También se coincidió con los resultados de Srikantha Thyagarajan en el año 2020, para quien los géneros Ralstonia y Staphylococcus fueron los más abundantes.34
Probablemente, estas similitudes se deben a la técnica empleada (metagenómica),35,36,37 que permite identificar microorganismos que no son fácilmente cultivables.38
Las dos pacientes positivas con cáncer (Tabla 4) mostraron crecimiento bacteriano de Enterobacterias y Staphylococcus, resultado muy parecido a lo mostrado por Urbaniak6 cuando encontró que el 30,8 % y 12,7 % de estas especies, respectivamente, estaban presentes en pacientes irlandesas con cáncer de mama. Urbaniak16 también indicó que el Staphylococcus fue uno de los taxones que encontraron con mayor abundancia en pacientes con cáncer de mama respecto a controles sanos.
La mayoría de las bacterias aisladas y purificadas en el cultivo bacteriano (Tabla 4) fueron grampositivas, lo que coincide con Kofteridis39 que indicó que desempeñan un papel importante como agentes causantes de infecciones de la piel y los tejidos blandos.
Las muestras número 7 y 35 presentaron una microbiota que coincide con los resultados publicados por Urbaniak:6 de 43 muestras canadienses cultivadas, identificó ocho cepas diferentes, entre ellas el S. saprophyticus, el cual hace parte de los estafilococos coagulasa negativos, considerados flora residente del ser humano y no dañina para los pacientes sanos. Sin embargo, han sido reconocidos como agentes etiológicos de procesos infecciosos para el hospedero con sistema inmunitario comprometido.
Hay investigaciones que muestran los cambios morfológicos que sufren las células in vitro cuando son infectadas por bacterias, como es el caso de Ludmila Alekseeva40 en el año 2013 cuando investigó el efecto citopático de las cepas de S. aureus de origen humano y animal y su capacidad para afectar el ciclo de la célula hospedadora en líneas celulares de células epiteliales HeLa y MAC-T bovina. La invasión de S. aureus ralentizó la proliferación celular e indujo un efecto citopático, lo que resultó en el agrandamiento de las células huésped. Ellos observaron una disminución dramática en el número de células mitóticas en los cultivos infectados.
En este estudio se intentó demostrar que las células MCF-7 tienen su comportamiento bien marcado a partir de la infección con S. saprophyticus a las 48 h con una MOI de 1:1, 5:1 y 10:1, obtuvimos como mayor efecto la formación de filopodias o lamelipodias en las células. Estas prolongaciones aparecen cuando las células están incubadas con alguna bacteria que las perturbe.40,41,42) Esta observación podría llevar a más ideas en la patogenicidad de las infecciones mamarias recurrentes donde está presente esta especie.
Conclusiones
El bacterioma encontrado en los nódulos es en su mayoría flora endógena de la mama. Al hacer comparaciones de nuestros resultados con la bibliografía, se infiere que en procesos tempranos se encuentra la flora normal y en procesos avanzados permanece, pero de forma aumentada, y se modifica cuando existen procesos infecciosos asociados.
Con respecto a la infección celular (MCF-7 y S. saprophyticus), las filopodias observadas aparecen cuando las células están perturbadas por un agente externo, lo que podría llevar a pensar sobre la patogenicidad de esta especie sobre la mama.
Con la metodología planteada no podemos ser concluyentes si la bacteria promueve o da un microambiente que favorece el crecimiento tumoral o, al contrario, si el tumor propicia las condiciones para que ellas proliferen. Probablemente en procesos avanzados se da la interacción de diferentes especies microbianas.