Introducción
El trasplante relacionado de células progenitoras hematopoyéticas (TCPH) es una alternativa terapéutica curativa para los pacientes con ciertos tipos de hemopatías o de inmunodeficiencias, en la que se selecciona como donante a un familiar del receptor.1,2
Tradicionalmente el TCPH requirió de una total identidad a nivel de los antígenos leucocitarios humanos (HLA, del inglés human leukocyteantigens);1 lo que dificultaba encontrar a un donante relacionado compatible, debido a que según las leyes mendelianas de la herencia, solo el 25 % los hermanos de madre y padre son HLA idénticos. (3
La probabilidad de encontrar donantes relacionados se ha incrementado recientemente con el desarrollo de técnicas para el llamado TCPH haploidéntico, porque la mayoría de los pacientes puede tener un familiar con al menos la mitad de los antígenos HLA en común.4
La dependencia entre el éxito de los TCPH y la compatibilidad HLA se apoya en los estudios de George D. Snell y Baruj Benacerraf sobre el complejo principal de histocompatibilidad (MHC, del inglés major histocompatibility complex), que es el locus génico donde se codifican los antígenos del sistema HLA, que describió a su vez Jean Dausset.5
La importancia de la compatibilidad entre donante y receptor comenzó a evidenciarse con los primeros TCPH en humanos, realizados en 1957 por E. Donnall Thomas, entre gemelos idénticos, y en 1958 por Georges Mathé, con donantes no relacionados. 6,7 Estos procedimientos se basaron en los postulados del siglo XIX de Artur Pappenheim y otros científicos, sobre la existencia de una célula precursora hematopoyética. (8-10
En Cuba, como en muchos países, no existen condiciones que permitan un amplio acceso a los TCPH no relacionados y la mayoría de los donantes deben seleccionarse dentro del seno familiar. (11 Teniendo en cuenta esta situación, se realizó la presente investigación con el objetivo determinar la frecuencia génica y de haplotipos de los loci HLA-A, HLA-B, HLA-DRB1 y HLA-DQB1, en los receptores cubanos de TCPH relacionado.
Métodos
Se realizó una investigación descriptiva y transversal en el departamento de Histocompatibilidad del Instituto de Hematología e Inmunología en la que se tipificó por técnicas biología molecular el HLA de los 117 pacientes de toda Cuba con criterio de TCPH relacionado, que se diagnosticaron en el periodo de enero de 2013 a diciembre de 2015. Se tuvieron en cuenta los aspectos éticos para las investigaciones en humanos que se recogen en la última revisión de la Declaración de Helsinki.
Se extrajeron 4 mL de sangre periférica que se dispensaron en tubos con ácido etilen diamino tetracético (EDTA). El aislamiento del ADN se realizó con el estuche “QIAamp DNA Blood Mini” automatizado con un “QIAcube” (Qiagen, Alemania). A su vez, la concentración y pureza del ADN se determinaron con un monocromador de microgotas “EPOCH” con el software “Gen 5” versión 2.00 (BioTek, EEUU).
Se utilizó un método de baja resolución de la reacción en cadena de la polimerasa con cebadores específicos de secuencia, (PCR-SSP, del inglés Polymerase Chain Reaction - Sequence Specific Primers), con estuches Olerup SSP® HLA-A-B-DR-DQ SSP, lotes 43R, 43V, 46N, 65Y y 70S (Olerup, Alemania).
Para la detección de los productos de PCR aplicó la electroforesis capilar en gel mediante el sistema QIAxcel Advance con cartuchos DNA Fast Analisys (Qiagen, Alemania); y para la interpretación y reporte de la tipificación HLA se utilizó el software Helmberg-Score versión 5.00.41 y la base de datos de alelos IMGT/HLA versión 3.16.0.07.44.
Para los análisis inmunogenéticos se utilizó el programa Arlequin versión 3.5. (Instituto suizo de bioinformática, Suiza). Las frecuencias alélicas y de haplotipos se estimaron mediante el algoritmo EM (esperanza-maximización, del inglés expectation-maximization), y el equilibrio de Hardy-Weinberg se determinó por la prueba exacta de Guo y Tompson. (12
Resultados
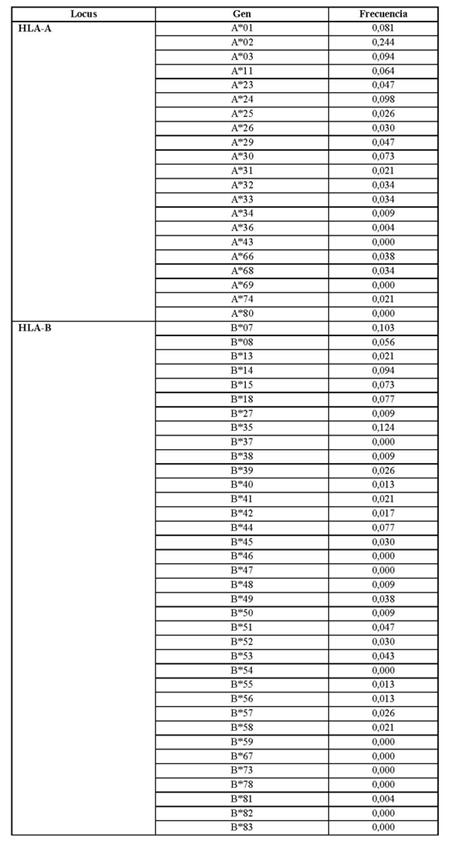
En los loci de HLA clase I, los cinco genes HLA-A de mayor frecuencia en orden decreciente fueron: A*02, A*24, A*03, A*01, A*30. De los 21 genes con posibilidades de detectarse en este locus no se encontraron: A*43, A*69, A*80. Por su parte, los cinco genes predominantes dentro de los 36 HLA-B posibles fueron: B*35, B*07, B*14, B*18 y B*44. No aparecieron B*37, B*46, B*47, B*54, B*59, B*67, B*73, B*78, B*82 y B*83 (Tabla 1).
Tabla 1- Frecuencia de genes HLA clase I en receptores cubanos de trasplante relacionado de células progenitoras hematopoyéticas
En los loci de HLA clase II, de los 13 genes HLA-DRB1, los cinco más frecuentes fueron: DRB1*03, DRB1*04, DRB1*15, DRB1*13, DRB1*01, coincidiendo en el primer lugar DRB1*03 y DRB1*04. A su vez, de los cinco genes HLA-DQB1, predominaron: DQB1*03, DQB1*02 y DQB1*06 (Tabla 2).
Tabla 2 Frecuencia de genes HLA clase II en receptores cubanos de trasplante relacionado de células progenitoras hematopoyéticas
| Locus | Gen | Frecuencia |
|---|---|---|
| HLA-DRB1 | DRB1*01 | 0,103 |
| DRB1*03 | 0,167 | |
| DRB1*04 | 0,167 | |
| DRB1*07 | 0,098 | |
| DRB1*08 | 0,038 | |
| DRB1*09 | 0,021 | |
| DRB1*10 | 0,021 | |
| DRB1*11 | 0,060 | |
| DRB1*12 | 0,017 | |
| DRB1*13 | 0,120 | |
| DRB1*14 | 0,034 | |
| DRB1*15 | 0,150 | |
| DRB1*16 | 0,004 | |
| HLA-DQB | DQB1*02 | 0,244 |
| DQB1*03 | 0,299 | |
| DQB1*04 | 0,047 | |
| DQB1*05 | 0,175 | |
| DQB1*06 | 0,235 |
Se estimaron 108 haplotipos de dos loci clase I HLA-A-B y 30 de clase II HLA-DRB1-DQB1. De ellos, los más frecuentes fueron HLA-A*02B*35, HLA-A*03B*07, HLA-A*30B*18, HLA-DQB1*02DRB1*03 y HLA-DQB1*03DRB1*04.
Por otro parte, se encontraron 166 haplotipos extendidos HLA-A-B-DRB1-DQB1; encabezando la lista HLA-A*02 B*15 DQB1*03 DRB1*04, HLA-A*02 B*35 DQB1*03 DRB1*04 y HLA-A*03 B*07 DQB1*06 DRB1*15, este último, con la mayor frecuencia.
Discusión
Los resultados de la presente investigación fueron similares para el locus HLA-A a los que se encontraron en otros estudios realizados en la población general de Cuba, entre ellos el de Ferrer y colaboradores en el Centro de Ingeniería Genética y Biotecnología (CIGB) de La Habana, en el que se realizó la tipificación molecular HLA clase I de 390 personas, y se obtuvo que los genes HLA-A más frecuentes fueron A*02, A*03 y A*24. (13
Paradoa y colaboradores, tipificaron 129 donantes de sangre de La Habana con hallazgos semejantes para A*02, A*03 y A*24; al igual que Morera y colaboradores en el IHI, que estudiaron la frecuencia antigénica en la población cubana según características étnicas. Todos estos grupos encontraron al A*02 como el gen más representativo del locus HLA-A. 14,15)
A nivel internacional, Mori y Cao en Estados Unidos de Norteamérica, encontraron a A*02, A*24, A*03, A*30, A*68 y A*01, como los más frecuentes, 16-18 y al igual que otros investigadores coinciden en que el gen del locus HLA-A con mayor representación en las poblaciones es el A*02. Dentro de ellos se encuentran Gonzales y colaboradores que tipificaron 411 muestras de pacientes en tratamiento de diálisis en Mar del Plata; 19 Peterson y colaboradores que estudiaron en el este de África a más de 2000 mujeres; (20 o Davidyuk y colaboradores que se realizaron la tipificación HLA de 483 posibles donantes para TCPH en Tatarstan, Rusia. (21
En cuanto al locus HLA-B, los presentes resultados coinciden con los de los grupos de Ferrer y Paradoa, que encontraron entre los genes más frecuentes al B*44 y B*35. (13,14 De igual forma, Williams y colaboradores en su análisis de la distribución del HLA-B en la población de cinco continentes, reportaron B*44, B*07 y B*14 dentro de los más representativos de los cubanos. 22
En el presente estudio, al igual que en los reportes del grupo de Davidyuk, se encontró alta frecuencia a los genes B*35, B*07, B*44. (21 Sin embargo en la población de Mar del Plata se reportaron B*35, B*44 y B*51; 19 lo que coincidió solo de forma parcial, porque el B*51 apareció en el octavo lugar entre los receptores cubanos de TCPH; lo que pudo deberse a que se trata de un gen mayormente asociado a otras etnias o a que Gonzales hizo su investigación en población con insuficiencia renal crónica.
Igual interpretación puede hacerse al comparar los presentes resultados con los de Osman y colaboradores en la población Saudí, en la que el gen HLA-B más frecuente fue también el B*51, (23 o con los de Peterson en el este de África, que reportaron el B*58,20 que en la presente investigación ocuparon octavo y el decimoséptimo lugar, respectivamente.
Las frecuencias de los genes de locus HLA-DRB1 que se determinaron fueron similares a las de Paradoa y colaboradores y de otros grupos internacionales como los liderados por Rodríguez y por Arias, ambos de Colombia, y el de Vargas de México, quienes encontraron una alta representación en sus poblaciones de DRB1*04, DRB1*07 y DRB1*13;(14, 24-26) al igual que Davidyuk, que reportó DRB1*04 y DRB1*07.21 Aunque el gen DRB1*07 no estuvo dentro de los que encabezaron la lista en la presente investigación, ocupó un sexo lugar de un total de 13.
Sin embargo, Vargas al estudiar la población mestiza de México en el año 2010, además de los genes mencionados, encontró que el de mayor frecuencia fue el DRB1*08,26 lo cual contrasta con los resultados del presente estudio. De igual forma, Peterson y colaboradores en el este de África, encontraron a DRB1*11,20 que entre los receptores de TCPH cubanos no es frecuente. Estos hallazgos pudieron deberse a las diferencias étnicas específicas de ambas poblaciones con la cubana.
Los resultados de la tipificación del locus HLA-DQB1 no pueden compararse correctamente con otros estudios publicados debido a la actualización de la nomenclatura del sistema HLA. Por ejemplo, en el año 1998 en Venezuela, Rivera y colaboradores encontraron en la población de Zulia una frecuencia de antígenos DQ similares a los que informaron Moscoso y colaboradores en el año 2006 en el grupo étnico Maya: DQ1, DQ3 y DQ2.27,28)
Según la nomenclatura actual, el antígeno DQ1 se refiere tanto al gen DQB1*05 como al DQB1*06, que aparecen separados en la presente investigación. De igual forma, el antes llamado DQ8, se corresponde con el alelo DQB1*03:02, lo cual dificulta las comparaciones.11 No obstante, el hallazgo de los grupos de Rivera y Moscoso de que los antígenos DQ2 y DQ3 fueron los más frecuentes, coinciden con los de la presente investigación para los genes DQB1*02 y DQB1*03. 27,28
En la citada población del este africano Peterson y colaboradores se encontraron al alelo DQB1*03:01:01 como el más frecuente, al igual que en el previamente mencionado estudio en Tatarstan. 21,22 A su vez, los genes DQB1*02, DQB1*03, y DQB1*06 fueron los de mayor frecuencia en el estudio de la población Saudi, 23 coincidente con la presente investigación.
Otro factor que complejiza la comparación de los resultados del presente estudio, es que no existen publicaciones que reporten las frecuencias para los genes del locus HLA-DQB1 en la población cubana. En el 2005 Morera y colaboradores estudiaron 721 cubanos que dividieron según color de la piel y solo reportaron las frecuencias de aquellos genes con diferencias estadísticamente significativas entre grupos, las cuales no se encontraron paracitado HLA-DQB1.15
En concordancia con la presente investigación, en los estudios de Gonzales en Mar del Plata se encontró con una elevada frecuencia al haplotipo de clase I HLA-A*03 B*07. Por el contrario, no reportaron a HLA-A*30 B*18, probablemente debido a las diferencias génicas poblacionales, que fue el tercero en los receptores cubanos de TCPH relacionado. (19
En Tatarstan, Davidyuk y colaboradores encontraron en el primer lugar a HLA-A*03 B*35; en Colombia, Rodrígues y colaboradores, el HLA-A*02 B*51 y en la población Saudí, Osman y colaboradores, HLA-A*02 B*50,21,23,24 que en la presente investigación ocuparon los lugares décimo segundo, decimonoveno y trigésimo quito, respectivamente, de los 108 que se estimaron.
En China, Cao y colaboradores encontraron en 4 541 pacientes cantoneses con insuficiencia renal crónica que el haplotipo HLA clase I más frecuente contenía el gen A*02 asociado con el B*46, 29 combinación que no apareció en ningún individuo en la presente investigación, lo que pudo deberse a que se presenta fundamentalmente en individuos asiáticos.
En la población mestiza de México, Vargas y colaboradores informaron que predominó el haplotipo de clase II HLA-DQB1*04 DRB1*08, seguido por HLA-DQB1*02 DRB1*07 y HLA-DQB1*03 DRB1*04;26 coincidiendo este último con el mayor frecuencia del presente trabajo. Por otra parte, en la población amerindia de Chile, Parga-Lozano y colaboradores estimaron que el HLA-DQB1*03 DRB1*14 y HLADQB1*04 DRB1*08,29 que resultaron los menos relevantes en los receptores cubanos de TCPH relacionado.
Sin embargo, entre los tres haplotipos extendidos clase I y II, formados por los loci HLA-A-B-DRB1-DQB1 más frecuentes en la citada población amerindia, estuvo HLA-A*03 B*07 DQB1*06 DRB1*15,30 que coincidentemente fue el número uno en el presente estudio. En el año 2015 Hajjej y colaboradores identificaron como haplotipos extendidos más relevantes en la población Tunecina el HLA-A*02 B*50 DQB1*02 DRB1*07 y el HLA-A*24 B*08 DQB1*02 DRB1*03, 31. El primero no se encontró en la población objeto de estudio y el segundo, apareció en el lugar 70 de 166.
Estas semejanzas y diferencias en cuanto a la frecuencia génica y de haplotipos pudieron deberse a que la mezcla genética de los cubanos está determinada por un contexto de migración y mestizaje entre españoles caucásicos, negros africanos y la población aborigen cubana. 32,33
Dentro de los genes HLA en receptores cubanos TCPH relacionado, se encuentran entre los de mayor frecuencia HLA-A*02, B*35, DQB1*03, DRB1*03 y DRB1*04; los haplotipos HLA-A*02-B*35, HLA-DQB1*03-DRB1*04 y el haplotipo extendido HLA-A*03-B*07-DQB1*06-DRB1*15 y poseen características similares a las descritas en otras poblaciones del país, Latinoamérica y el resto del mundo, aunque con características distintivas a nivel de algunos genes y haplotipos.














