Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Investigaciones Biomédicas
versión On-line ISSN 1561-3011
Rev Cubana Invest Bioméd v.21 n.1 Ciudad de la Habana ene.-mar. 2002
Técnicas
Centro de Investigaciones Biomédicas
Un modelo computacional de reacciones relacionadas con el mal de Alzheimer en el nivel molecular
Lic. Annia Galano Jiménez, Lic. Luis A. Montero Cabrera, Lic. Raúl Álvarez Idaboy, Lic. Addis Londaitsbehere Trujillo y Dr. José C. García Piñeiro
Resumen
El desarrollo tecnológico de los medios de cómputo ha hecho posible que se estudien sistemas cada vez mayores y con mayor grado de exactitud. El método empleado es el llamado semiempírico con la parametrización PM3 implementado en el programa MOPAC. En este trabajo se modeló la ruptura de los diferentes enlaces del carbono a, así como la del enlace peptídico. Los cálculos fueron hechos en procesadores Pentium Pro (200 MHz, 64 bytes RAM), Dual Pentium II (233 MHz, 128 bytes RAM), Dual Pentium Pro (200 MHz, 128 bytes RAM). La modelación de las reacciones radicálicas de los aminoácidos constituyentes del fragmento activo del péptido b-amiloide arrojó que era posible la reacción de formación de radicales libres y la ulterior reactividad de estos, que puede conducir a la degradación oxidativa de moléculas circundantes. Esta generación de radicales libres es significativa en las posiciones Ca y la asparagina aparece como el resto más activo.
DeCS: SIMULACION POR COMPUTADOR; MUERTE CELULAR; RADICALES LIBRES; QUIMICA CEREBRAL; PROTEINA BETA AMILOIDE; OXIDACION QUIMICA; ENFERMEDAD DE ALZHEIMER.
Una de las teorías más largamente sostenidas acerca del envejecimiento, sugiere que el deterioro gradual de las células nerviosas es causado por la acumulación de los daños provocados por la oxidación en el cuerpo humano. Muchos científicos creen que los radicales libres producidos mediante mecanismos oxidativos desempeñan un papel de importancia en muchas enfermedades, incluidos el cáncer y la enfermedad de Alzheimer (EA).
Un metabolismo saludable puede producir radicales libres oxigenados al nivel celular como una de sus actividades normales y estos se mantienen en un equilibrio propio del sistema particular. Sin embargo, cualquier desbalance en este equilibrio que aumente su proporción anormalmente en el citosol puede tener la reacción secundaria de dañar ciertos procesos celulares. Tal equilibrio puede ser alterado al llegar radicales libres o sus precursores desde el exterior (polución, luz ultravioleta, medicamentos e incluso algunos alimentos) o por problemas propios del sistema. Se afirma que el estrés tiende a afectar este proceso celular de forma negativa. Por su alta reactividad cuando ocurre una concentración anormalmente alta, los radicales libres atacan con facilidad a otras moléculas que se encuentren cercanas. De este modo se crea un nuevo radical a partir de la molécula atacada que está listo para repetir el proceso, estableciéndose una reacción en cadena que puede llegar a destruir células completas.
Se sospecha que los radicales libres desempeñan un papel en el desarrollo de la EA por varias razones. Ellos atacan moléculas lipídicas en las membranas celulares de las células nerviosas, con esto pueden alterar el delicado mecanismo que regula la entrada y salida de las sustancias a través de dichas membranas, como es el caso del Ca2+, que en exceso puede provocar la muerte de las células. Además de esto, las oxidaciones por causa de radicales libres pueden provocar alteraciones en las proteínas o sus restos, y estas nuevas formas que toman las proteínas como producto de la acción de radicales libres pueden asociarse con el desarrollo de la EA. Algunos de estos cambios se han encontrado en las placas características de la enfermedad, cuyo componente principal es el péptido b-amiloide.1 Este péptido se encuentra parcialmente fuera de las células nerviosas y parcialmente en la membrana celular, donde puede ocasionar la acción oxidativa y es liberado producto de la acción de una proteasa sobre la llamada proteína precursora del amiloide (PPA).
La secuencia aminoacídica de este péptido es:2
Asp-Ala-Glu-Cys-Arg-His-Asp-Ser-Gly-Tyr10 -Glu-Val-His-His-Gln- Lys-Leu-Val-Phe-Phe20 -Ala-Glu-Asp-Val-Gly-Ser-Asn-Lys-Gly-Ala30 -Ile-Ile-Gly-Leu-Met-Val-Gly-Gly-Val-Val40 -Ile-Ala-Thr
De la cual la sección 25-35 es al parecer la más involucrada en la reacción con radicales asociada con la EA3 y en este trabajo se enfatizará, consecuentemente, en los restos aminoacídicos de esta sección.
Estudios realizados4-6 han demostrado que las proteínas expuestas al radical hidroxilo o a la combinación de este con dioxígeno y el radical peroxilo ·OH + O2- + O2 constituyen un buen modelo que imita la exposición biológica a los radicales del oxígeno. La interacción provoca alteraciones de la estructura primaria, no ocurre así, u ocurre en mucha menor medida, cuando el radical hidroxilo no está presente. Por otra parte estos mismos estudios muestran que la fragmentación debe involucrar la reacción entre los radicales formados, producidos en la reacción con el ·OH y el dioxígeno, para formar radicales peroxilos que se descomponen y provocan la ruptura de la cadena polipeptídica. Esta se ve más favorecida por la posición a que por el enlace peptídico, y se deduce de que una vez ocurrida la ruptura se observa un incremento de grupos carbonilo sin aumento aparente de grupos amino libres.
Sobre la base de todo lo expuesto, en el presente trabajo se estudió la reacción de abstracción de átomos de hidrógeno por causa del ataque del radical hidroxilo sobre los diferentes sitios reactivos posibles de cada aminoácido (AAH) de interés:
AAH + ·OH AA· + H2O [1]
Así como sobre el dipéptido Gly-Ala, por estar presente en la región de interés del b-amiloide, y las posteriores reacciones de adición de dioxígeno, eliminación de oxígeno atómico y ruptura de la cadena.
Métodos
Los métodos de la física cuántica son empleados exitosamente para el estudio de diferentes problemas de interés químico, físico e incluso biológico. La aplicación en este último campo es algo más reciente que en el resto, por causa del tamaño generalmente grande de los sistemas a estudiar y a la gran demanda computacional de estos métodos. El desarrollo tecnológico de los medios de cómputo ha hecho posible que se estudien sistemas cada vez mayores y con mayor grado de exactitud. El método empleado es el llamado semiempírico con la parametrización PM3, implementado en el programa MOPAC,7 que ha sido empleado como primera aproximación en el estudio de los puntos estacionarios de la reacción entre el radical hidroxilo y los aminoácidos estudiados, por diferentes posiciones en todos los casos. También se usó para la posterior adición del O2 al sitio radicálico del dipéptido Gly-Ala y la eliminación de oxígeno atómico seguida de la ruptura molecular. Se modeló la ruptura de los diferentes enlaces del carbono a, así como la del enlace peptídico. Los cálculos fueron hechos en procesadores Pentium Pro (200 MHz, 64 bytes RAM), Dual Pentium II (233 MHz, 128 bytes RAM), Dual Pentium Pro (200 MHz, 128 bytes RAM).
Resultados
Este trabajo es un acercamiento al problema en el que se postula el primer paso como una abstracción de hidrógeno causada por el acercamiento del radical de hidroxilo a determinado sitio reactivo. Esto da como resultado un nuevo radical con el electrón desapareado en la posición atacada, tal y como se describe en la reacción [1]. Este primer paso fue modelado sin tener en cuenta la influencia del solvente ni de portador alguno para el radical hidroxilo, se optimizaron los diferentes puntos estacionarios del perfil de reacción (reaccionantes, estados de transición y productos) a partir de los cuales se calcularon las energías de activación y los calores de reacción correspondientes.
Como en todos los casos las reacciones aparecen muy favorecidas termodinámicamente, el proceso inverso a la temperatura ambiente (o corporal) sería muy poco probable, por lo que para analizar las reactividades relativas de las especies estudiadas se tiene que introducir también como parámetro significativo a la energía de activación, expresada como la diferencia de energías entre el complejo acitvado intermediario y las formas reaccionantes.
AAH +OH AA------ H·-----HO [2]
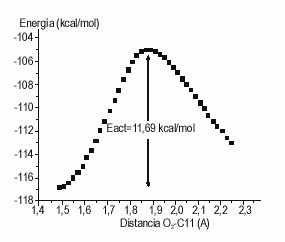
El segundo posible paso de este tipo de reacción es la adición de O2 al nuevo sitio radicálico, para formar un radical peroxilo. Este paso ha sido modelado con el método PM3 para el Ca de la alanina en el dipéptido Gly-Ala (fig. 1), obteniéndose una energía de activación de 48,9 kJ/mol. Para este paso fue modelado el camino de reacción (fig. 2). El valor obtenido de energía de activación debe estar sobrestimado, como ocurre con este tipo de reacciones en sistemas análogos.8 Aun así, este valor muestra que la reacción estudiada ocurre a velocidades apreciables a temperatura ambiente.
Fig. 1. Radical peroxilo del dipéptido Gly29 – Ala.30
Fig. 2. Camino de reacción de la adición de O2 al C de la alanina, en el dipéptido Gly-Ala.
El siguiente paso sería la ruptura de la cadena peptídica, según se infiere de lo reportado.4-6 Esto fue modelado y se encontró que la energía crece monótonamente en la medida en que se elonga cualquiera de los posibles enlaces involucrados en la ruptura (fig. 3). Como puede observarse, incluso para el enlace más débil (C11-C12) el aumento de la energía es demasiado elevado (alrededor de 150,6 kJ/mol) para que ocurra una ruptura con velocidad apreciable a temperatura ambiente. Lo anterior demuestra que es necesario un paso adicional anterior que favorezca tal ruptura de los enlaces, que es conocida por datos experimentales correspondientes a reacciones similares en química atmosférica en las que esto ocurre después de la eliminación de oxígeno atómico como abstracción del mismo por el radical NO.
Fig. 3. Variación de la energía durante la ruptura de enlaces en el radical peroxilo.
Teniendo en cuenta la variedad de radicales presentes en el cuerpo humano, se decidió hacer la modelación con el menos reactivo de ellos (oxígeno atómico), para garantizar que de ser esta posible, la abstracción con el radical escogido sería también posible con cualquier otro radical presente. En la figura 4 puede verse la variación de energía en la medida en que el oxígeno atómico se acerca al extremo del radical peroxilo. Como puede apreciarse la reacción de abstracción ocurre sin barrera alguna, lo que condiciona que esta sea una reacción controlada por la velocidad de difusión. Esto permite asumir que es muy probable que ocurra una reacción de este tipo como paso intermedio entre la adición de oxígeno molecular al radical peptídico y la ruptura de la cadena. Al modelarse la ruptura luego de la abstracción de oxígeno se encontró que en este caso el comportamiento energético depende del enlace que se pretenda disociar. Como se observa en la figura 5 los enlaces C6-C9 (enlace peptídico) y C11-N9 presentan un comportamiento análogo al caso anterior, pero para los enlaces C11-C14 y C11-C12 luego de una barrera alrededor de 63 y 33 kJ/mol respectivamente, ocurre la ruptura. Lo anterior lleva a predecir que el enlace más favorecido para la disociación es precisamente el C11-C12, ruptura que está favorecida no solo desde el punto de vista cinético sino también termodinámico. Esto concuerda con las evidencias experimentales4-6 de que al romperse la cadena peptídica aumenta el número de grupos carbonilo, y se mantiene constante la de grupos aminos libres.
Fig. 4. Camino de reacción de la abstracción de oxígeno.
Fig. 5. Variación de la energía durante la ruptura de enlaces en el radical alcoxilo.
Discusión
El mecanismo que rige la reacción entre aminoácidos y el radical hidroxilo, que termina con la ruptura de la cadena peptídica está aún desconocido experimentalmente.
Desde el punto de vista de los calores de reacción, se encontró que la reacción del radical hidroxilo con los aminoácidos muestra el rasgo distintivo de que aparece favorecida siempre en la posición del carbono a, con los valores más negativos para cada aminoácido comparado con otras posiciones. Desde el punto de vista de la energía de activación esto casi siempre se confirma también con los valores más bajos, excepto los casos de la isoleucina y la leucina.
Se encontró que la isoleucina sería el resto aminoacídico más lábil a la reacción con este radical libre si el sistema estuviera aislado (en ausencia de solvente u otro portador del radical libre), de acuerdo con el método de cálculo seleccionado. Debe anotarse que en estas condiciones, la diferencia de energías encontrada no permite conclusiones determinantes en este aspecto y será preciso realizar cálculos con mayor completamiento teórico y complejidad para confirmar o modificar esta hipótesis. Los sitios más favorecidos termodinámicamente son los Ca. Desde el punto de vista cinético serían más favorecidos los Cb de la isoleucina, el Ca de la serina y también el Cg de la leucina.
La modelación de las reacciones radicálicas de los aminoácidos constituyentes del fragmento activo del péptido b-amiloide arrojó que era posible la reacción de formación de radicales libres y la ulterior reactividad de estos que podía conducir a la degradación oxidativa de moléculas circundantes. Esta generación de radicales libres fue significativa en las posiciones Ca y la asparagina aparece como el resto más activo.
El mecanismo que conduce a la ruptura de la cadena peptídica parece contar, al menos, de los pasos siguientes: abstracción de hidrógeno por el radical libre atacante, adición de oxígeno molecular procedente del suero, abstracción de oxígeno atómico por cualquiera de los radicales presentes y ruptura de la cadena por un enlace C-C de esqueleto peptídico.
Los métodos mecánico-cuánticos resultan adecuados para modelar este tipo de problema biológico. En el presente caso es necesario refinar estos resultados elevando el nivel de cálculo e introduciendo moléculas del ambiente, como puede ser el agua, para que cumplan la función de portadoras y eventualmente de catalizadoras en la simulación.
Summary
The technological development of the computation means has made possible the study of increasingly large systems and with a higher degree of accuracy. The method used is the so-called semiempiric with the PM3 parametrization implemented in the MOPAC program. The rupture of the different bonds of carbon a, as well as that of the peptide bond were designed in this paper. The calculations were made in Pentium Pro (200 MHz, 64 bytes Ram), Dual Pentium II (233 MHz, 128 bytes RAM) and Dual Pentium Pro (200 MHz, 128 bytes RAM) processors. The design of the radical reactions of the aminoacids constituting the active fragment of the b-amyloid peptide showed that the reaction of formation of free radicals and the further reactivitiy of them that may lead to the oxidative degradation of the sorrounding molecules was possible. This generation of free radicals is significant in the positions Ca and asparagine appears as the most active rest.
Subject headings: COMPUTER SIMULATION; CELL DEATH; FREE RADICALS; BRAIN CHEMISTRY; AMYLOID BETA-PROTEIN; ALZHEIMER DISEASE; CHEMICAL OXIDATION; ALZHEIMER DISEASE.
Referencias bibliográficas
- Martin Citron, Thekla S. Diehl, Grace Gordon, Anja Leona Biere, Peter Seubert, Dennis J. Selkoe: Evidence that the 42 -and 40- amino acid forms of amyloid beta protein are generated from the beta-amyloid precursor protein by different protease activities. Proceedings of the National Academy of Sciences 1996;93(23):13170-75.
- Aksenov MY, Aksenova MV, Carney JM, Butterfield DA. Oxidative modification of glutamine synthetase by amyloid beta peptide. Free Radic Res 1997 Sep; 27(3):267-81.
- Butterfield DA, Martin L, Carney JM y Hensley K. A beta (25-35) peptide displays H2O2 like reactivity towards aqueous Fe2+, nitroside spin probes, and sinaptosomal membrane protein. Life Sci 1996;58(3):217-28.
- Davies KJ, Delsignore ME. Protein damage and degradation by oxygen radicals. III. Modification of secondary and tertiary structure. J Biol Chem 1987 Jul 15;262(20):9908-13.
- Davies KJ, Delsignore ME, Lin SW. Protein damage and degradation by oxigen radicals. II. Modification of amino acids. J Biol Chem 1987 Jul 15;262(20):9902-7.
- Davies KJ, Lin SW, Pacifici RE. Protein damage and degradation by oxygen radicals. IV. Degradation of denatured protein. J Biol Chem 1987 Jul 15;262(20):9914-20.
- Stewart J. J. P. MOPAC, v. 6, release for PC computers by L. A. Montero. In the Laboratory of Computational and Theoretical Chemis try, Universidad de La Habana, 1993-1997, and v. 7, for Linux system as implemented in the same laboratory, 1995.
- Díaz-Acosta I, Álvarez-Idaboy JR, Vivier-Bunge A. Mechanism of the OH-Propene-O2 Reaction: An ab-initio Study. J Chem Kinetics 1999;31(1):29-36.
Recibido: 3 de abril de 2000. Aprobado: 27 de junio de 2000.
Lic. Annia Galano Jiménez. Laboratorio de Química Computacional y Teórica, Facultad de Química, Universidad de La Habana, La Habana. CP 10400, Cuba. Correo electrónico. annia@fq.oc.uh.cu