Señor Editor:
El interesante artículo de Sotomayor y otros1 entrega información sistematizada y actualizada acerca de la estructura general, genómica y proteica de SARS-CoV-2, agente etiológico causante de la pandemia de enfermedad por coronavirus (COVID-19). Ante la necesidad de contar con información oportuna y veraz para enfrentar esta amenaza para la salud global, resulta también interesante referirse desde una perspectiva histórica y evolutiva a los coronavirus, con especial atención en el SARS-CoV-2.
Los coronavirus (familia Coronaviridae, subfamilia Coronavirinae) son importantes patógenos en aves y mamíferos2) y, con toda probabilidad, nos han estado acechando desde los albores de la humanidad, pues son previos a ella. Si bien los estudios de análisis de reloj molecular sugieren que el ancestro común más reciente de los coronavirus se encontraría hace unos 10 000 años atrás,2 esto ha sido cuestionado por investigaciones filogenéticas que plantean la posibilidad de que estos virus estén infectando quirópteros (murciélagos) y/o aves desde el origen de estos clados, hace decenas de millones de años o, incluso, desde mucho antes.2 Aunque la historia evolutiva de los coronavirus es extensa, estos agentes patógenos fueron descubiertos recién a principios de la década de 1930, cuando se demostró que una infección respiratoria aguda en pollos era causada por un virus, ahora conocido como virus de la bronquitis infecciosa aviar.3 Los primeros coronavirus humanos se descubrieron en la década de 1960: una investigación con voluntarios humanos en la Unidad de Resfriado Común cerca de Salisbury, Reino Unido, mostró que las muestras nasales que no contenían rinovirus podían inducir resfriados.3 Los experimentos in vitro posteriores, en los que muestras de hisopos nasales de estos voluntarios se inocularon en cultivos de órganos del tracto respiratorio, revelaron la presencia de virus envueltos con la morfología característica de los coronavirus, tal como se había descrito previamente para el virus de la bronquitis infecciosa aviar.3 Lo anterior permite reconocer que existe una larga historia de investigación sobre coronavirus (Fig. 1), la cual comenzó hace casi 90 años y que ha permitido tanto construir una gran base de conocimientos científicos, como desarrollar herramientas técnicas para investigar a estos agentes patógenos.4
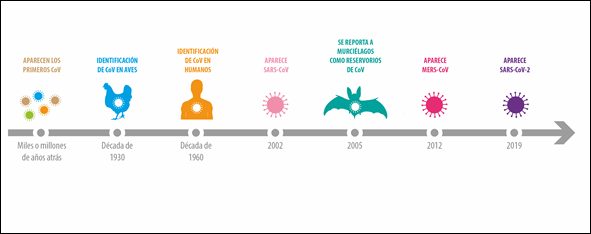 SARS-CoV: coronavirus del síndrome respiratorio agudo grave. MERS-CoV: coronavirus del síndrome respiratorio de Oriente Medio. SARS-CoV-2: Coronavirus tipo 2 del síndrome respiratorio agudo grave.
SARS-CoV: coronavirus del síndrome respiratorio agudo grave. MERS-CoV: coronavirus del síndrome respiratorio de Oriente Medio. SARS-CoV-2: Coronavirus tipo 2 del síndrome respiratorio agudo grave.Fig. 1 - Línea temporal que muestra algunos hitos importantes respecto a los coronavirus (CoV).
Los coronavirus se agrupan en cuatro géneros: Alfacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus. SARS-CoV-2 es un Betacoronavirus.1,5 Análisis filogenéticos recientes han revelado una gran diversidad de Alfacoronavirus y Betacoronavirus y una fuerte señal de coevolución entre los coronavirus y sus especies hospedadoras (quirópteros, por ejemplo), con evidencia limitada de cambio de hospedador, excepto para especies de murciélagos que comparten sitios de descanso diurno.6 Existe una relación filogenética cercana entre SARS-CoV-2, SARS-CoV, MERS-CoV y otros virus encontrados en murciélagos que son similares a SARS.5 Las diferencias estructurales y funcionales entre estos coronavirus han sido estudiadas mediante el análisis de las substituciones de los aminoácidos de diferentes proteínas.5 Se han encontrado 380 sustituciones de aminoácidos entre las secuencias de aminoácidos de SARS-CoV-2 (cepa HB01) y las secuencias consenso correspondientes de SARS y virus similares al SARS.5 Además, se encontraron 27 sustituciones de aminoácidos en la proteína espícula (S), subunidad S1.5 La proteína S es la que le otorga en las microfotografías electrónicas al virus su característico atuendo de «corona» y, en particular, se sabe que la subunidad S1 es altamente variable entre diferentes coronavirus.1)
Investigar estas diferencias en el futuro es importante, ya que podría dar luces sobre los mecanismos de tropismo del hospedador y la propiedad de transmisión de SARS-CoV-2 en comparación a otros coronavirus.5 El conocer bien la estructura y función de la proteína S es fundamental para el desarrollo de vacunas y de antivirales efectivos.7
En virtud de lo expuesto, se puede concluir que los coronavirus son enemigos antiguos, pero con diferentes «atuendos». Las estrategias de prevención y tratamiento ante los brotes epidémicos que puedieran ocasionar estos virus en el futuro requieren indudablemente de un enfoque multidimensional y multidisciplinario.














