Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión impresa ISSN 1010-2752
Rev. Protección Veg. vol.29 no.2 La Habana Mayo.-ago. 2014
ARTÍCULO ORIGINAL
Evaluación de dos técnicas para la detección molecular de rickettsia asociada a la enfermedad del cogollo arrepollado del papayo en Cuba
Evaluation of two techniques for the molecular diagnosis of rickettsia associated to papaya bunchy top disease in Cuba
Lester Hernández-Rodríguez*, Edel Pérez López, Maritza Luis Pantoja, Inés Peña Bárzaga
Instituto de Investigaciones en Fruticultura Tropical. Ave. 7ma No. 3005. Playa, La Habana. Cuba.
RESUMEN
El objetivo de este estudio fue optimizar una técnica de diagnóstico para rickettsia-PBT, bacteria asociada a la enfermedad cogollo arrepollado del papayo (PBT), basada en la hibridación de ácidos nucleicos no radiactiva (HANS-PBT), y comparar la factibilidad de su uso con respecto a la PCR convencional. Se optimizó un procedimiento de HANS-PBT que permitió la detección específica de hasta 15 pg de ADN del gen sdhA de rickettsia-PBT. La evaluación de 141 controles caracterizados mediante PCR convencional (PCR-PBT), mostró parámetros de desempeño de la HANS-PBT favorables, con una especificidad diagnóstica (ED) de 97,2% y sensibilidad diagnóstica (SD) de 98,5%. Los análisis simultáneos mediante las dos técnicas de 179 muestras colectadas mayormente en plantaciones del occidente de Cuba evidenciaron la presencia de rickettsia-PBT en 118 plantas. Los parámetros de desempeño de la PCR-PBT y HANS-PBT en la evaluación de estas muestras fueron similares, 96% y 98% de ED, 89% y 75% de SD, respectivamente. Aunque la HANS-PBT mostró valores de sensibilidad menores a la PCR-PBT, es factible su uso para programas de mejoramiento genético o prospecciones en estudios epidemiológicos de la diseminación de la bacteria.
Palabras clave: cogollo arrepollado del papayo, rickettsia, hibridación, papayo, especificidad, sensibilidad.
ABSTRACT
The aim of this study was to develop a technique based on non radioactive nucleic acid hybridization for the diagnosis of rickettsia-PBT bacterium associated to the papaya bunchy top disease (NASH-PBT), and to evaluate its use compared with the conventional PCR. The optimized NASH-PBT method allowed the specific detection of 15 pg of sdhA gen DNA of rickettsia-PBT. The validation parameters in the evaluation of 141 controls previously characterized by PCR evidenced the reliability of the technique, showing diagnosis sensitivity (DS) of 97.2% and diagnosis specificity (DE) of 98.5%. The simultaneous analysis by the NASH-PBT and conventional PCR (PCR-PBT) of 179 samples recovered from commercial fields, mostly of the western region of Cuba, evidenced the presence of rickettsia-PBT in 118 plants. The parameters of validations of PCR-PBT and NASH-PBT in the analysis of these samples were 89% and 75% for DS and 96% and 98% for DE, respectively. Despite the lower sensibility value showed respect to NASH-PBT PCR-PBT, its use for genetic improvement programs or surveys in epidemiological studies of this pathogen is realiable.
Key words: papaya bunchy top, rickettsia, hybridization, papayo, sensitivity, specificity.
INTRODUCCIÓN
El papayo (Carica papaya L.) es un cultivo extensivo en zonas tropicales y subtropicales, comercializado principalmente para consumo fresco, como productos elaborados y para la producción de papaína (1). En Cuba, anualmente se siembran entre 4000 y 5000 ha de este frutal (2) y en el 2012 se produjeron 88139,0 ton, principalmente de los cultivares Maradol amarilla y roja (3).
De las enfermedades sistémicas que afectan este frutal, se considera que la mancha anular del papayo (causada por Papaya ring spot virus, PRSV) y el cogollo arrepollado del papayo (PBT, siglas del nombre en inglés papaya bunchy top) son las de mayor impacto en los rendimientos del cultivo en Cuba. Esta última enfermedad se informó en el país en 1946, por Acuña y Zayas, y se denominó inicialmente como Mosaico tipo A, Mosaico común o «Bunchy top» (4). Hasta el momento, PBT constituye un problema fitosanitario que se encuentra extendido por toda la isla, por lo que resulta difícil encontrar una localidad en la que se cultive el papayo que esté completamente libre de la enfermedad (5).
Los síntomas característicos de PBT, informados en la literatura en otros países incluyen la clorosis, necrosis y reducción del tamaño de las hojas; entrenudos cortos; pecíolos endurecidos, pequeños y dispuestos en posición casi horizontal, y detención del crecimiento del meristemo apical. Adicionalmente en los estadios finales se produce una fuerte defoliación; ausencia o escasa emisión de látex, disminución del vigor; muerte regresiva y escasa producción de flores y frutos (6). Estudios relacionados con la enfermedad en Cuba, catalogaron estos síntomas dentro de un complejo denominado ¨síndrome de la enfermedad PBT¨, o Bunchy top symptom complex (BTS). En este complejo se adicionan a los síntomas descritos anteriormente el arrugamiento y mosaico de las hojas, frutos pequeños y otros (7).
El agente causal de los síntomas de PBT fue objeto de discusión a escala internacional. Primeramente fue asociado a un virus (8), posteriormente a un fitoplasma (9) y otros estudios lo asociaron a una bacteria fastidiosa del subgrupo 1-alfa de las proteobacterias, ubicada dentro del género Rickettsia (rickettsia-PBT) (10). La trasmisión y diseminación de estos agentes ocurre mediante injerto, y de forma natural por insectos del tipo saltahojas. Se identificó principalmente a Empoasca papayae Oman como vector de la enfermedad, aunque también se indicaron a Empoasca stevensi Young y Empoasca dilitara Delong et Davidson (10).
En Cuba se detectaron fitoplasmas y rickettsia en plantas con síntomas característicos de PBT, tanto por microscopia electrónica, como por reacción en cadena de la polimerasa. Estos estudios se complementaron con la secuenciación y el alineamiento con secuencias de referencia, los cuales mostraron un alto porcentaje de identidad nucleotídica con aislamientos caracterizados en otros países (7,11). Esta dualidad de agentes patógenos en el país, así como la alta presencia de infecciones mixtas encontradas en prospecciones realizadas en campos comerciales de papaya, pudieran explicar el complejo de síntomas asociados a la enfermedad que se fueron descritos en el país (7,11,12,13).
Ambos patógenos, tanto fitoplasmas como rickettsia-PBT, son microorganismos no cultivables en medios sintéticos, por lo que sólo se pueden diagnosticar mediante métodos moleculares. Se optimizaron varios métodos para la detección de rickettsia-PBT y fitoplasmas como son, la microscopia electrónica, PCR convencional y la PCR anidada (nPCR), e incluso ensayos dúplex de detección de los patógenos (11, 12). Estos procedimientos, aunque con alta especificidad y sensibilidad, tienen limitaciones debido al costo por análisis y la baja capacidad para procesar muestras en un mismo ensayo (14, 15, 16). El objetivo de este trabajo fue optimizar la técnica de hibridación de ácidos nucleicos no radiactiva (HANS) para la detección de rickettsia asociada a la enfermedad del cogollo arrepollado del papayo en Cuba y evaluar su desempeño al compararla con la PCR convencional.
MATERIALES Y MÉTODOS
Obtención de los extractos de ADN
Los ácidos nucleicos totales se extrajeron a partir de pecíolos de hojas de plantas de papayo, siguiendo la metodología de extracción de Murray y Thompson (17). La integridad del ADN se comprobó mediante electroforesis en gel de agarosa al 0,8% y tinción con bromuro de etidio (10 mg/ml) (18). Las concentraciones de los extractos de ADN se cuantificaron mediante espectrofotometría a 260 nm (Spectronic Genesys 5, Spectronic UNICAM, Cambridge, UK), y se verificó por estimación visual en geles de agarosa al 0,8% según Sambrook et al. (17).
Reacción en Cadena de la Polimerasa convencional para la detección de ADN de rickettsia-PBT
La detección de rickettsia-PBT mediante PCR convencional (PCR-PBT) se realizó con el uso de los cebadores PBTR1/PBTF1, que amplifican de forma específica un fragmento de 704 pb del gen que codifica para la enzima succinato deshidrogenasa A (sdhA) de esta bacteria (10). La reacción de PCR se realizó en un termociclador Mastercycler Gradient (Eppendorf, Hamburg, Alemania) con un volumen final de reacción de 30 µl. Los reactivos utilizados para la amplificación fueron los del sistema PCR Master (Roche Diagnostics, Mannheim, Alemania), con condiciones de reacción similares a las diseñadas por Davis et al. (10).
En las reacciones de diagnóstico de rickettsia-PBT mediante PCR-PBT se utilizaron como molde 300 ng de cada extracto de ADN de la planta en análisis. Como control positivo de la presencia de rickettsia-PBT, se utilizaron 300 ng de ADN de una planta de papayo (denominada PBT2003-01), en la que previamente se detectó rickettsia-PBT a través de microscopia electrónica, PCR y caracterización molecular del fragmento del gen sdhA (número de acceso en GenBank: FN825675) (11). Los productos del PCR fueron comprobados por electroforesis en gel de agarosa al 0,8% y tinción con bromuro de etidio (10 mg/ml) (18). La cuantificación de los productos de PCR en los ensayos requeridos se realizó de la misma forma que los extractos de ADN, descrito en el acápite anterior.
Marcaje de la sonda no radioactiva PBT para la detección de rickettsia-PBT
El marcaje de la sonda no radiactiva de ADN complementario (ADNc) se realizó con el sistema PCR-Dig-Probe según las especificaciones del fabricante (Roche Diagnostics) y utilizando los cebadores y condiciones de la PCR-PBT. En la reacción de marcaje se usó como molde 100 pg del plásmido pTPBT, el cual contiene clonado el fragmento de 704 nt del gen sdhA de rickettsia-PBT aislado a partir de extractos de ADN de la planta PBT2003-01 (11). Los productos de la PCR se analizaron mediante electroforesis en gel de agarosa al 0,8% con tinción de bromuro de etidio, y se incluyó como control del marcaje, el producto resultante de la PCR obtenido en un ensayo simultáneo sin la incorporación de Dig-11-dUTP en la mezcla de reacción de la PCR. Posteriormente a la PCR, la banda de la sonda se separó en un gel de agarosa de bajo punto de fusión y se purificó mediante el sistema High Pure PCR Product Purification Kit (Roche Diagnostic).
Hibridación de ácidos nucleicos
La técnica de hibridación de ácidos nucleicos utilizada fue similar a la descrita por Sambrook et al. (18). Los extractos de ácidos nucleicos de cada muestra (~10 µg), cuantificados según la metodología descrita anteriormente, se desnaturalizaron durante 5 minutos a 95ºC y se transfirieron a membranas de nylon (Hybond-N+, Amersham Biosciences), utilizando un sistema de transferencia por vacío Dot blot (Bio-Rad, California, EE.UU.). Posteriormente se fijaron en el horno de hibridación durante 2 horas a 80ºC y se prehibridaron durante 4 horas a 55ºC en el tampón de prehibridación (solución de Church) (18). La hibridación con la sonda (20 ng/cm2) se realizó durante toda la noche en tampón de prehibridación a la misma temperatura. Se hicieron dos lavados con SSC 2X (NaCl 0,3 M, SDS 0,1% y citrato de sodio 30 mM) a temperatura ambiente, seguido de un lavado con SSC 0,1% y SDS 0,1% durante una hora a 60ºC. Para la detección de los híbridos de ADN marcados con las sondas de digoxigenina se utilizó el conjugado anti-DIG-fosfatasa alcalina (fragmentos FAB) (Roche Diagnostics). La visualización se obtuvo con el sustrato quimioluminiscente CSPD (Roche Diagnostics) que al descomponerse y emitir luz sensibilizó las películas X-omat (KodaK), reacción que se llevó a cabo a 37ºC durante una hora.
Sensibilidad y especificidad analítica de la hibridación de ácidos nucleicos para la detección de rickettsia-PBT
Para determinar el límite de detección de la HANS-PBT se transfirieron a la membrana cantidades de 15, 1,5; 0,15; 0,015; y 0,0015 ng del producto de PCR-PBT, amplificado utilizando como molde el plásmido pTPBT. Las cantidades del producto de la PCR-PBT se mezclaron con 10 µl de extractos de ADN (100 µg/µl) de una planta de papayo obtenida en condiciones de aislamiento. Además, se transfirieron a la membrana cinco muestras con 10 µl de extractos de ADN (100 µg/µl) de una planta de papayo obtenida en condiciones de aislamiento (blancos experimentales), con una repetición intra-ensayo de cada muestra. La concentración de los extractos de ADN se determinó como se describió anteriormente. Para determinar la especificidad de la sonda se utilizaron en un ensayo de HANS-PBT 10 µl de extractos de ADN cromosomal (100 ng/µl) de 10 organismos no relacionados: Escherichia coli, Pichia pastoris, Candidatus Liberibacter asiaticus, Agrobacterium tumefaciens, Bradyrhizobium japonicum, Coconut lethal yellow phytoplasma, planta de vicaria (Catharanthus roseus) infectada con fitoplasma no caracterizado, Nicotiana tabacum L., Citrus sinensis Osbeck, cola de ratón (Mus musculus), células de ovario de hámster CHO (del inglés Chinese hamster ovary) y el insecto Planococcus citri (Risso). Se realizaron dos repeticiones intra-ensayo de cada muestra. Además se incluyeron cuatro extractos de plantas de papayo obtenidas a partir de la siembra de semillas y mantenidas en condiciones de aislamiento.
Cálculo de los parámetros de desempeño y evaluación de la HANS-PBT
Para estandarizar la HANS-PBT se utilizaron diez repeticiones inter-ensayo y cinco repeticiones intra-ensayo de cinco controles positivos y cinco negativos en cada ensayo. Con el objetivo de evaluar los parámetros de desempeño de la HANS-PBT se analizaron 141 controles de ADN previamente evaluados por PCR-PBT y se construyó una tabla de contingencia. De esta población de controles, 72 eran controles positivos (positivos PCR-PBT), y los restantes 69 como negativos (negativos PCR-PBT). En cada membrana se transfirieron las muestras de la curva utilizada en el ensayo de sensibilidad analítica con una réplica intra-ensayo de cada punto de la curva. Además, se transfirieron tres muestras, con una réplica cada una, de 10 µg de ADN de plantas sanas utilizadas como blancos experimentales y controles negativos del ensayo. El criterio de positividad de la muestra se correspondió con el determinado en el ensayo de sensibilidad analítica de la HANS-PBT. Los parámetros del desempeño se calcularon según Massart et al. (19) y la interpretación de los valores de RV se realizó según la escala descrita por Capote et al. (20). La coincidencia entre las técnicas de diagnóstico PCR-PBT y HANS-PBT se calculó mediante el índice Kappa de Cohen (21, 22, 23). Los intervalos de confianza (IC) para los parámetros estimados fueron calculados según Olmos et al. (23).
Comparación de la HANS-PBT y PCR-PBT en el diagnóstico de rickettsia-PBT en muestras de campo
Para comparar el desempeño de la HANS-PBT y la PCR-PBT en la evaluación de muestras de campo se analizó, de forma simultánea, una población de muestras colectadas en campos comerciales de papayo. Las muestras se colectaron durante prospecciones realizadas en plantaciones de papayo del cultivar Maradol rojo de seis provincias ubicadas en la región occidental, central y oriental de Cuba. Se colectaron un total de 179 muestras de hojas, 65 de La Habana, 35 de Artemisa, 17 de Mayabeque, 19 de Matanzas, 22 de Cienfuegos y 21 de Holguín. El criterio de planta enferma en este ensayo se consideró como las plantas con síntomas de porte bajo y aspecto arrepollado, acortamiento de los entrenudos, ausencia de emisión de látex y acumulación de ampollas de látex en la base de los peciolos (Fig. 1), descritos como característicos para la enfermedad PBT según Davis et al. (6,10).
Las extracciones de ADN y su cuantificación se realizaron como se detalló anteriormente. El diagnóstico por PCR-PBT se realizó tomando 300 ng de cada extracto como molde y las condiciones y reactivos fueron los descritos anteriormente. Para la HANS-PBT se transfirieron a las membranas 10 µg de ADN de cada muestra con una réplica por muestra, siguiendo el procedimiento detallado en el acápite anterior. En cada membrana se transfirió la curva de sensibilidad analítica y tres blancos experimentales con una réplica de cada punto. El criterio de positividad del resultado de la hibridación en este ensayo se consideró según el procedimiento descrito en el acápite de cálculo de la sensibilidad analítica. Los parámetros de desempeño se calcularon para ambas técnicas siguiendo la metodología descrita anteriormente.
RESULTADOS Y DISCUSIÓN
Sensibilidad y especificidad analítica de la hibridación de ácidos nucleicos para la detección de rickettsia-PBT
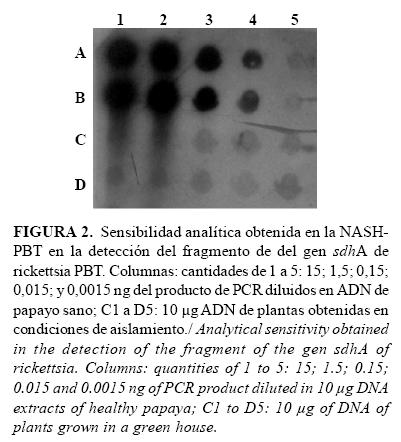
La menor cantidad del producto de PCR-PBT detectada, utilizando la técnica de HANS-PBT, fue de 15 pg (Fig. 2, carriles A4 y B4). En las posiciones correspondientes a la cantidad inferior (1,5 pg) se observaron señales débiles (Fig. 2, carriles A5 y B5), que se consideraron negativas por tener la misma intensidad de la señal de fondo obtenida en los blancos experimentales (Fig. 2, carriles C1 y B1 a C5 y B5). Sin embargo, la intensidad de la señal obtenida en las posiciones correspondientes a 15 pg sugiere que en una cantidad intermedia pudiera haberse obtenido una señal diferenciable de los blancos experimentales. En los siguientes experimentos se tomó como criterio de positividad aquellas señales que resultaran con intensidades tres veces mayores que las señales de fondo de los blancos experimentales, situando en todos los ensayos la misma curva de sensibilidad y varias réplicas de los blancos experimentales.
El límite de detección de 15 pg es adecuado para una técnica de diagnóstico de patógenos en plantas, si se toma en consideración trabajos realizados por varios autores que utilizaron digoxigenina como método para el marcaje de sondas en experimentos de dot blot. En este sentido se logró establecer límites de detección desde los 10 ng hasta 85 ng de ADN de Tomato yellow leaf curl virus (TYLCV) (24, 25), mientras que en el diagnóstico del viroide del tubérculo ahusado de la papa (PSTVd), se informaron límites en el rango de 2,0 a 63 pg de ARN viroidal (26, 27).
En la determinación de la especificidad analítica se evidenció que ninguno de los extractos de ADN de las muestras de diferentes organismos transferidas y fijadas a las membranas reaccionó de forma positiva con la sonda PBT. Además, se observaron señales fuertes en las posiciones donde se ubicaron los controles positivos, correspondientes con el extracto de ADN de una planta en la que se detectó la bacteria por microscopía electrónica, PCR y secuenciación del fragmento amplificado (planta PBT2003-01). Así mismo, el comportamiento de los resultados de las muestras de la curva de límite de detección y los blancos experimentales fue similar al obtenido en el experimento de sensibilidad analítica. Los resultados demostraron que la sonda PBT, conformada por una secuencia parcial del gen sdhA de rickettsia-PBT, detectó de forma específica la presencia de hasta 15 pg de ADN de este gen de la bacteria en los extractos de plantas de papayo con el procedimiento utilizado.
Evaluación de los parámetros de desempeño de la HANS-PBT en una población de controles caracterizados por PCR-PBT
La reproducibilidad de la técnica se evidenció al obtenerse señales de hibridación fuertes a partir de los controles positivos y ausencia de señal en los negativos en las diez repeticiones inter-ensayo y cinco repeticiones intra-ensayo de cinco controles positivos y cinco negativos. En la determinación de los parámetros de desempeño de la HANS-PBT al analizar un total de 141 controles previamente caracterizados por PCR-PBT, de los 72 positivos a la infección con rickettsia-PBT, sólo dos plantas no mostraron señal en la HANS-PBT (Tabla 1). Por otro lado, solamente una planta mostró señal en la hibridación de los 69 controles negativos utilizados (plantas obtenidas a partir de semillas y negativas por PCR-PBT), la cual se consideró falso positivo (Tabla 1).
Los valores de sensibilidad y especificidad diagnóstica de la HANS-PBT fueron superiores al 97%, al igual que en los valores predictivos negativos y positivos de la HANS-PBT (Tabla 2). Esto indica una alta capacidad de la técnica para detectar plantas infectadas, mientras que el valor predictivo positivo desarrollado por la técnica, evidencia la alta probabilidad de que un valor de positividad concuerde con una planta realmente infectada (23). Por otra parte, la alta especificidad permite afirmar que la HANS-PBT manifiesta una alta confiabilidad en la discriminación de la presencia de rickettsia-PBT asociada a la enfermedad PBT en las muestras analizadas, resultado sustentado por la baja probabilidad de obtener resultados falsos positivos y el alto valor predictivo negativo (23).
Los valores de las razones de verosimilitud negativa y positiva obtenidas (Tabla 2), indican una alta confiabilidad para la determinación de la presencia de rickettsia-PBT, independientemente de la prevalencia de la enfermedad (23). Resultados similares fueron descritos para el diagnóstico de estas bacterias mediante otras plataformas de diagnóstico (13), y evidencia la capacidad de la metodología para su uso en experimentos en los que la prevalencia de la enfermedad puede ser baja (28).
Al analizar la coincidencia de las dos técnicas empleadas en la evaluación de la población de controles utilizada se obtuvo un índice de kappa de Cohen de 0,958. Este valor determina una coincidencia casi perfecta de los dos métodos según lo establecido para los estándares aplicados en veterinaria y medicina para los análisis de diagnósticos. El índice de kappa de Cohen relaciona la concordancia en los resultados que se obtienen mediante el procesamiento individual por dos metodologías, y considera que dos métodos coinciden de forma casi perfecta cuando se obtienen valores de este parámetro entre 0,81 y 1,0 (20, 29). En su conjunto los resultados permiten recomendar la técnica evaluada para el diagnóstico de rickettsia-PBT en estudios de epidemiología, certificación de material de propagación, programas de mejoramiento y otros. Resultados similares en los parámetros de evaluación han permitido la utilización de otras plataformas de diagnóstico en procederes de certificación en otros cultivos (18, 28, 29).
Detección de rickettsia-PBT en plantas de papayo cultivar Maradol roja de campos comerciales
En la comparación de la eficacia en el diagnóstico de rickettsia-PBT al analizar un total de 179 muestras mediante la HANS-PBT y PCR-PBT, 100 de las plantas analizadas mostraron señal positiva en la HANS-PBT (Fig. 3), mientras que 118 fueron positivas a partir de los resultados de la PCR-PBT (Tabla 3). Por otro lado, de los 46 extractos de ADN de las plantas asintomáticas, solamente uno mostró señal positiva de hibridación, mientras que dos muestras amplificaron un producto en la PCR-PBT, los que constituyeron falsos negativos de los experimentos (Tabla 3).
En la evaluación de los parámetros de desempeño obtenidos de ambos métodos en el análisis de muestras de campo se encontraron algunas diferencias entre los resultados individuales de cada técnica (Tabla 4). La precisión y la sensibilidad diagnóstica de la PCR-PBT, así como los parámetros relacionados con estos, como son la tasa de falsos negativos y el valor predictivo negativo, fueron superiores a las obtenidas con la HANS-PBT. Estos valores sugieren una mayor confiabilidad en el descarte de la enfermedad, a partir del resultado negativo de la PCR-PBT que la HANS-PBT. Sin embargo, la especificidad diagnóstica y los valores relacionados con este parámetro (tasa de falsos positivos y el valor predictivo positivo), fueron superiores en la HANS-PBT. Esto indica una mayor confiabilidad en la discriminación de la presencia de rickettsia-PBT en las muestras a partir del resultado positivo de la HANS-PBT que la PCR-PBT (23).
La menor sensibilidad diagnóstica de la HANS-PBT puede justificar la obtención de 33 resultados falsos negativos en el experimento. Esto pudiera deberse a la distribución irregular de la bacteria sistémica en las plantas, o a la existencia de un bajo titulo del patógeno. Por otro lado, en la PCR-PBT se obtuvo un falso positivo más que en la evaluación mediante la HANS-PBT, evidencia de una menor especificidad de esta metodología con respecto a la HANS-PBT. Este problema en las técnicas de PCR está debidamente explicado en la literatura y es atribuido al alto poder de amplificación que induce la ocurrencia de contaminaciones cruzadas (28).
Al analizar la población de muestras de campo tomando como criterio de positividad los síntomas de PBT, ambos métodos de diagnóstico confirmaron la presencia de rickettsia-PBT con una misma prevalencia, mientras que la habilidad de ambos métodos para la detección de los positivos (razón de verosimilitud positiva) fue mayor de diez (Tabla 4). Este valor es el límite indicativo de que un método de diagnóstico confirma la presencia de un patógeno asociado a una enfermedad en muestras colectadas en condiciones de campo, según la escala de valores descrita por Capote et al. (20).
Ambas metodologías mostraron una moderada capacidad de discriminación de la enfermedad, debido a las altas razones de verosimilitud negativas que se obtuvieron. Los valores menores que 0,1 son los aceptados como de alta capacidad de descarte de la enfermedad para un método de diagnóstico (20). Al combinar las razones de verosimilitud y graficarlas se pudo observar que a partir del 10% de prevalencia de la enfermedad en el campo, se incrementó la probabilidad de un diagnóstico efectivo de rickettsia-PBT mediante ambas técnicas (Fig. 4). En este caso, fue mayor la probabilidad de que el resultado positivo de la HANS-PBT coincidiera con una planta infectada.
Al comparar los resultados de la HANS-PBT en la evaluación de controles positivos, caracterizados mediante PCR-PBT como técnica de referencia y de muestras de campo tomando como criterio de positividad de planta enferma la sintomatología PBT, se observaron algunas diferencias de los parámetros de evaluación. Una primera causa puede ser que en el último ensayo no se contó con una cantidad balanceada de controles positivos y negativos (enfermos y sanos), proporción que resulta la adecuada para este tipo de evaluaciones (30). Por otro lado, en el análisis de las plantas de campo el criterio de positividad para la colecta de muestras se basó en los síntomas referidos como característicos para la enfermedad PBT. Esto representa una posible fuente de error ya que se ha demostrado la asociación de fitoplasmas con plantas de papayo con síntomas similares a los de PBT en Cuba (7, 13), y se ha informado una alta incidencia de infecciones mixtas de estos dos patógenos (12, 13).
La frecuencia de detección de plantas infectadas con rickettsia-PBT fue alta en las provincias de La Habana (55/65), Artemisa (32/35), Mayabeque (16/17), Matanzas (12/19), mientras que en Cienfuegos y Holguín fue baja (2/22, 1/21, respectivamente). Estos resultados indicaron la distribución de rickettsia-PBT en las seis provincias recorridas, lo cual coincide con los informes de detección de este patógeno en Cuba informados con anterioridad a este trabajo (7, 11, 12, 13). Así mismo, la prospección realizada para la colecta de muestras amplía la información de la distribución de rickettsia-PBT en el país, al detectar la bacteria en plantas con síntomas de la enfermedad en cuatro nuevas provincias: Artemisa, Mayabeque, Matanzas y Cienfuegos. De la misma forma, este estudio sustenta mediante una aproximación diagnóstica diferente, la hibridación de ácidos nucleicos no radioactiva, la relación de esta bacteria con la enfermedad PBT, como se ha descrito en la literatura (6, 10, 11).
Se describió en la literatura que, para algunas interacciones planta-patógeno, los métodos moleculares para la detección del agente causal no brindan los niveles de sensibilidad requeridos para el diagnóstico (31). Factores como la desigual distribución de un patógeno en una planta, su presencia en títulos muy bajos o en infecciones latentes, y la presencia de compuestos que inhiben la actividad de las enzimas empleadas en las técnicas, constituyen retos para el analista encargado del diagnóstico (32, 33). La solución para estos problemas pudiera derivar del desarrollo de metodologías integrales o polifásicas del diagnóstico, o sea, combinar dos o varios métodos con principios diferentes y que brinden una mayor precisión al combinarse entre sí (14, 31).
En Cuba, se optimizaron varios métodos para el diagnóstico de los agentes causales asociados al complejo PBT, tanto fitoplasmas como rickettsia-PBT (7, 11, 12). Tomando en consideración las técnicas de diagnósticos disponibles, y la optimizada en esta investigación, se propone un esquema múltiple de detección para la toma de decisiones en el diagnóstico del o los agentes causales de los síntomas de PBT. Este esquema puede abarcar varias alternativas, dependiendo del objetivo del análisis que se esté realizando y las condiciones del laboratorio en que se esté ejecutando (Fig. 5).
De manera general, todas las alternativas deben iniciarse con la colecta de muestras en el campo y la obtención de los extractos de ADN (Fig. 5 ). En experimentos que requieran el procesamiento de un número alto de muestras se pueden combinar la alta especificidad diagnóstica de la HANS-PBT y su capacidad para el procesamiento de muchas muestras en un mismo experimento (Fig. 5¨variante 1¨). Se aplicaría inicialmente esta técnica para descartar los positivos y las muestras que resultaran negativas se confirmarían mediante la PCR-PBT. Se debe tener en cuenta la utilización de una correcta cantidad de réplicas en la PCR-PBT debido a los problemas de contaminaciones cruzadas que presenta la PCR (19, 28). Tomando en consideración que la PCR puede verse inhibida por varias razones aportando resultados falsos negativos al experimento (32), un examen final abarcaría la microscopia electrónica en aquellas muestras negativos que así lo requieran. No obstante, este análisis resulta muy costoso y tiene la desventaja de no poderse realizar a un número alto de muestras (14). Esta alternativa es válida para experimentos como la prospección y epidemiología para evaluar la diseminación temporal de rickettsia-PBT asociada a PBT, o programas de mejoramiento vegetal.
Sin embargo, este flujo de trabajo no estaría completo para experimentos de certificación de material de propagación de papaya o determinación del agente causal en plantas con síntomas de PBT si no se realiza el diagnóstico de fitoplasmas. En este caso, deberán procesarse paralelamente las muestras tanto por el flujo de diagnóstico de rickettsia-PBT como mediante la técnica de nPCR (Fig. 5 ¨variante 2¨). No obstante, este objetivo se puede lograr también con la utilización de la PCR anidada múltiple (Fig. 5 ¨variante 3¨) (12). Con esta técnica se simplifican las operaciones y se obtiene una disminución el costo del análisis al evaluar la presencia de los dos patógenos en la misma reacción. Los resultados de la evaluación de este método de diagnóstico múltiple fueron satisfactorios en el diagnóstico de muestras de campos con los síntomas de PBT y que contenían infecciones mixtas de fitoplasma y rickettsia-PBT (12).
AGRADECIMIENTOS
Este trabajo fue realizado gracias a la contribución del Ministerio de Ciencia, Tecnología y Medio Ambiente (CITMA) y del Grupo Empresarial Frutícola (GEF) de Cuba a través de los proyectos nacionales con códigos 00300243 y 0576, respectivamente.
REFERENCIAS
1. Teixeira da Silva JA, Rashid Z, Tan Nhut D, Sivakumar D, Gera A, Souza MTJr, et al. Papaya (Carica papaya L.) Biology and Biotechnology. Tree and Forestry Science and Biotechnology. 2007;1(1):47-73.
2. Alonso M, Tornet Y, Aranguren M, Ramos R, Rodríguez K, et al. Caracterización de los frutos de cuatro cultivares de papaya del grupo solo, introducidos en Cuba. Revista Agronomía Costarricense. 2008;32(2):169-175.
3. Anónimo 2012. Boletines de estadísticas del Ministerio de la Agricultura de Cuba.
4. Acuña J, Zayas F. El mosaico y otras plagas de la fruta bomba (Carica papaya L.). Est Exp Agr Cir. 1946; 85.
5. Peña I, López D, Hernández L, Luis M. Ocurrencia de las principales enfermedades sistémicas del papayo (Carica papaya L.) en Cuba. Citrifrut. 2010;27(1):47-263 53.
6. Davis MJ, Frences JB, Fewerda FH, Brunner BR. Association of a bacterium and not a phytoplasma with papaya bunchy top disease. Phytopathology. 1996;86:102-109.
7. Arocha Y, Piñol B, Acosta K, Almeida R, Devonshire J, Van de Meene A, et al. Detection of phytoplasma and potyvirus pathogens in papaya (Carica papaya L.) affected with Bunchy Top Symptom (BTS) in eastern Cube. J Crop Protection. 2009;28:640-646.
8. Bird J, Adsuar J. Viral nature of papaya bunchy top. J Agric Univ. P.R. 1952;35:5-11.
9. Story GE, Halliwell RS. Association of a mycoplasma-like organism with the bunchy top disease of papaya. Phytopathology. 1969;59:1336-1337.
10.Davis MJ, Kramer JB, Fewerda FH, Brunner BR. Rickettsia-PBT relative associated with papaya bunchy top disease. Current Microbiol. 1998;36:80-84.
11.Naranjo M, Luis M, Rohde W, Becker D, Rodríguez J, Rodríguez M, et al. Detección y caracterización de bacterias tipo Rickettsia-PBT en plantas con síntomas de cogollo arrepollado de la papaya en Cuba. En Actas del I Simposio Internacional sobre Vigilancia Fitosanitaria y su relación con la Protección del Entorno; Septiembre 2002; La Habana. Cuba.
12.Acosta K, Martínez Y, Zamora L, Fernández A, Santos-Cervantes ME, Leyva-López NE. PCR múltiple anidada para detección de fitoplasmas y rickettsia-PBT asociados con los síntomas del cogollo arrepollado (BTS) en papayo. Rev Protección Veg. 2011;26(3):156-163.
13.Acosta K, Zamora L, Piñol B, Fernández A, Chávez A, Flores G, et al. Identification and molecular characterization of phytoplasmas and rickettsia-PBT pathogens associated with `Bunchy Top Symptom' (BTS) and `Papaya Bunchy Top' (PBT) of papaya in Cuba. Crop Protection. 2013;45:49-56.
14.López MM, Llop P, Olmos A, Marco-Noales E, Cambra M, Bertolini E. Are molecular tools solving the challenges posed by detection of plant pathogenic bacteria and viruses? Curr Issues Mol Biol. 2009;11(1):13-46.
15.Rowhani A, Chay C, Golino DA, Falk B. Development of a Polymerase Chain Reaction Technique for the Detection of Grapevine Fanleaf Virus in Grapevine Tissue. Phythopathology. 1993;83:749-753.
16.Palacio-Bielsa A, Cambra M, López MM. PCR detection and identification of plant-pathogenic bacteria: updated review of protocols (1989-2007). J Plant Pathol. 2009;91(2):249-297.
17.Murray MG, Thompson WF. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Res. 1980;239:487-491.
18.Sambrook J, Fritsch EF, Maniatis T. Cold Spring Harbor Laboratory Press. Molecular cloning. A laboratory manual. 2nd edition Ed. New York. 1989.
19.Massart S, Brostaux Y, Barbarossa L, Battle A, Cesar V, et al. Inter-laboratory evaluation of two Reverse-transcriptase Polymerase Chain Reaction-based methods for detection of four fruit tree viruses. Ann Appl Biol. 2009;154:133-141.
20.Capote N, Bertolini E, Olmos A, Vidal E, Martinez MC, Cambra M. Direct sample preparation methods for detection of Plum pox virus by real-time RT-PCR. International Microbiology. 2009;12:1-6.
21.Cohen J. A coefficient of agreement of nominal scales. Educational and physiological measurement. 1960;20:37-46.
22.Landis JR, Koch GG. The measurement of observer agreement for categorical data. Biometrics. 1977;33:159-174.
23.Olmos-Castelló A, Bertolini E, Cambra-Álvarez M. Validación de métodos de detección y diagnóstico de patógenos y costes de la especificidad y sensibilidad. Boletín de la Sociedad Española de Fitopatología. 2008;63:7-11.
24.Caciagli P, Bosco D. Quantitative determination of tomato yellow leaf curl geminivirus DNA by chemiluminescent assay using digoxigenin-labelled probes. J Virol Methods. 1996;57(1):19-29.
25.Rodríguez R, Ramos PL, Doreste V, Velásquez K, Fuentes A, Pujol M. Establish of a non-radioactive nucleic acid hybridization technique for begomopvirus detection. Biotecnología Aplicada. 2003;20:164-169.
26.Nakahara KHT, Hataya T, Sugimoto T, Kimura I, Shigata E. A mixture of synthetic probes labelled with biotin for the sensitive detection of potato spindle tuber viroid. J Virol Methods. 1998;71:219-227.
27.Podleckis EV, Hammond RW, Hurt SS, Hadidi A. Chemiluminescent detection of potato and pome fruits viroid by digoxigenin-labelled dot blot and tissue blopt hybridization. J Virol Methods. 1993;43:147-158.
28.Collazo-Cordero C, Núñez-Luis JY, Luis-Pantoja M, Llauger-Riverón R. Optimización de la reacción en cadena de la polimerasa anidada para el diagnóstico de la enfermedad «huanglongbing» de los cítricos. Citrifrut. 2011;28(2):19-30.
29.Vidal E, Yokomi RK, Moreno A, Bertolini E, Cambra M. Calculation of Diagnostic Parameters of Advanced Serological and Molecular Tissue-Print Methods for Detection of Citrus tristeza virus: A Model for Other Plant Pathogens. Phytopathology. 2012;102(1):114-121.
30.OIE. Validation and quality control of polymerase chain reaction methods used for the diagnosis of infectious diseases. En: OIE (World Organization for Animal Health). Terrestial manual, France, 2008. pp 46-55.
31.Ramos-González PL, Hernández-Rodríguez L, Banguela-Castillo A. Plataformas genéricas para el diagnóstico a gran escala en fitopatología. CitriFrut. 2011;28(1):25-27.
32.Singh RP, Singh M, King RR. Poliphenolics compounds inhibit the Taq polymerase. J Virol Methods. 1998;74:231-235.
33.Galipienso L, Vives MaC, Navarro L, Moreno P, Guerra J. Detection of citrus leaf blotch virus using digoxigenin-labeled cDNA probes an RT-PCR. European Journal of Plant Pathology. 2004;110:175-181.
Recibido: 27-8-2013.
Aceptado: 5-2-2014.
*Autor para la correspondencia: Lester Hernández-Rodríguez. Correo electrónico:fitopatologia@iift.cu.