Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Habanera de Ciencias Médicas
versión On-line ISSN 1729-519X
Rev haban cienc méd v.9 n.4 Ciudad de La Habana oct.-nov. 2010
CIENCIAS CLÍNICAS Y PATOLÓGICAS
Hospital General Docente Héroes del Baire. Isla de la Juventud
Identificación de betalactamasas de espectro extendido en enterobacterias
Identification of extended spectrum â-lactamases in enterobacterias
MsC. Dr. Delfín Álvarez Almanza
Especialista Segundo grado en Microbiología. Auxiliar. Calle 49 número 2017 entre 20 y 22. Nueva Gerona Isla de la Juventud. Teléfono: 52616392. piro@ijv.sld.cu
RESUMEN
Las ß-lactamasas de espectro extendido (BLEE) son enzimas que fenotípicamente se caracterizan por conferir resistencia a penicilinas y cefalosporinas, incluyendo las de tercera y cuarta generación. Son en su mayoría producidas por enterobacterias especialmente frecuentes en Klebsiella pneumoniae y Escherichia coli. La identificación de este tipo de enzima es muy importante porque impone un plan de acción para el control de las infecciones que pudieran producirse. Por lo que se hace necesario diferenciar entre la producción de betalactamasas de espectro extendido (BLEE) y otros mecanismos de resistencia para evitar el tratamiento inadecuado de infecciones causadas por este tipo de cepas. Los procedimientos de laboratorio para la identificación de BLEE son muy importantes, existen varios métodos fenotípicos, de bioensayos, bioquímicos y genotípicos. No existe una metodología que aúne sensibilidad, especificidad y sencillez, dada la complejidad de la detección de las BLEE y sus variantes, por lo que se recomienda una adecuada interpretación de los perfiles de sensibilidad con los criterios habituales de lectura interpretada del antibiograma y posteriormente, se debe elegir el método de confirmación. Lo que permitirá conocer la verdadera dimensión del problema que representan, limitar su diseminación y adecuar las escasas opciones terapéuticas.
Palabras clave:ß-lactamasas espectro extendido. BLEE. Identificación. Enterobacterias. Mecanismos de resistencia, antibióticos.
ABSTRACT
The ß-lactamasses of extended spectrum (ESBL) are enzymes that are phenotypically characterized to confer resistance to penicillin and cephalosporin, including those of third and quarter generation. They are mainly produced by Enterobacteriaceae especially frequent in Klebsiella pneumoniae and Escherichia coli. The identification of this enzyme type is very important because it imposes an action plan for the control of the infections that could take place. Therefore it becomes necessary to differ among the production of betalactamasses of extended spectrum (ESBL) and other resistant mechanisms to avoid the inadequate treatment of infections caused by this type of stumps. The laboratory procedures for the identification of ESBL are very important; there are several phenotype methods, biologic, biochemical and genotype assay. A methodology that joints sensibility, specificity and simplicity, doesn't exist given the complexity of the detection of the ESBL and its variants, thus an appropriate interpretation of the profiles of sensibility is recommended with the habitual approaches of interpreted reading of the antibiogram and later on, the confirmation method should be chosen . What will allow to know the true dimension of the problem that it represents, to limit their dissemination and to adapt the scarce therapeutic options.
Key words: Extended spectrum ß _lactamasses (ESBL), Enterobacteriaceae Identification. Resistance mechanisms, antibiotics.
INTRODUCCIÓN
La aparición de microorganismos productores de b-lactamasas de espectro extendido (BLEE), capaces de inactivar potentes cefalosporinas, ha generado gran preocupación debido a las implicaciones clínicas y terapéuticas que tienen, debido a que son trasmitidas por plásmidos y por tanto pueden diseminarse a muchos microorganismos, la diseminación de la resistencia a las cefalosporinas de espectro extendido limita aún más el uso de los b-lactámicos y estimula el uso de antibióticos más costosos y mayor espectro; pero, además, estas cepas resistentes pueden no ser detectadas mediante los procedimientos microbiológicos de rutina y generar por tanto fallas terapéuticas frecuentes y, en ocasiones, fatales.1,2
Debido a la gravedad e importancia de algunas infecciones por cepas productoras de BLEE, a la dificultad de tratamiento que entrañan, al comportamiento epidemiológico, a la posibilidad de que puedan trasmitirse a otros enfermos o a bacterias de la microbiota comensal, y a las limitaciones con los criterios habituales de sensibilidad, el papel del laboratorio de microbiología es crucial. Este debe estar preparado para reconocer prontamente su presencia, dar la alerta ante la presencia de un fenotipo poco habitual e iniciar un plan racional para el control de las infecciones que pudieran producirse.2,3
Las ß-lactamasas de espectro extendido, son enzimas producidas por los bacilos Gram negativos, fundamentalmente enterobacterias frecuentes en Klebsiella pneumoniae y Escherichia coli, aunque también por microorganismos no fermentadores como Pseudomonas aeruginosa y otros. Son capaces de inactivar, además de las penicilinas y las cefalosporinas de primera y segunda generación, a las oximino-cefalosporinas y al aztreonam.1,2,4
Dado que la familia de enzimas BLEE está en continuo crecimiento, se reconocen según sus características funcionales y genotípicas, mediante la clasificación de Bush, Jacoby y Medeiros y su correlación con la clasificación molecular de Ambler. Las BLEE "clásicas" derivan de las ß-lactamasas pertenecientes al grupo 2b (TEM-1, TEM-2 y SHV-1). Estas ß-lactamasas 2b poseen actividad penicilinasa y son, en su mayoría, inhibibles por el ácido clavulánico. Las mutaciones en el centro activo de estas enzimas 2b, han determinado la aparición de las BLEE, capaces de hidrolizar también a las cefalosporinas de tercera generación y los monobactámicos. Las BLEE, por tanto, se clasifican dentro de ese gran grupo 2b, constituyendo el subgrupo 2be (clase molecular A). Hasta la fecha se han descrito más de cien variantes distintas de BLEE, derivadas de las ß-lactamasas TEM-1 o TEM-2 y más de cincuenta de las derivadas de SHV-1.4,5,6
Existen otros tipos de ß-lactamasas algunas de ellas se clasifican en grupos distintos al 2be; ejemplo: ciertas oxacilinasas del grupo 2d, clase molecular D, que predominan en Pseudomonas aeruginosa pero también se han detectado en otras bacilos Gram negativos, BLEE como cefotaximasas o CTX-M y otras BLEE poco frecuentes, que no se relacionan claramente con las familias de ß-lactamasas establecidas hasta ahora, por ejemplo, las de tipo PER, la VEB-1, la CME-1, la SFO-1, la TLA-1, las de tipo GES/IBC (GES-1, GES-2, IBC-1, caracterizadas por hidrolizar a la ceftazidima de forma más eficiente que a otros betalactámicos.5,6
Las cepas que producen BLEE, en su mayoría enterobacterias, y en particular Klebsiella pneumoniae y Escherichia coli, son resistentes a todos los antibióticos ß-lactámicos con la excepción de las carbapenemas, las cefamicinas y las combinaciones de ß-lactámicos con inhibidores de ß-lactamasas. Además de las BLEE clásicas, de naturaleza plasmídica, existe una serie de microorganismos que producen ß-lactamasas cromosómicas, que, en el caso de una hiperproducción, confieren fenotipos de resistencia similares al que determinan las BLEE. Entre las enterobacterias que producen de forma natural este tipo de ß-lactamasas se encuentran Yersinia enterocolitica, Klebsiella oxytoca, Citrobacter diversus y distintas especies del género Kluyvera. En los últimos años, han adquirido gran relevancia el tipo de BLEE plasmídicas, denominadas CTX-M que derivan de la ß-lactamasa cromosómica de distintas especies del género Kluyvera. Por lo general, cuando se habla de BLEE se hace referencia generalmente a las enzimas de codificación plasmídica ya que son estas las que suponen un mayor problema epidemiológico debido a su elevada capacidad de diseminación.7,8,9
DESARROLLO
Para la detección de BLEE desde el punto de vista técnico, el laboratorio de microbiología se enfrenta a dos retos. Por una parte, el elevado número de enzimas con características de BLEE que se han descrito y cuyas diferencias fenotípicas, a veces, son muy sutiles. Por otra, la posibilidad de encontrar cepas de enterobacterias que producen enzimas que no deben ser consideradas estrictamente como BLEE, como la hiperproducción de la ß-lactamasa cromosómica AmpC o la codificación plasmídica del gen AmpC por parte de diversos miembros de la familia Enterobacteriaceae, pero cuyo fenotipo puede parecerse al de las BLEE.
Las BLEE se pueden inferir con pruebas simples, aunque no existe una metodología que aúne sensibilidad, especificidad y sencillez, dada la complejidad de la detección de las BLEE y sus variantes. Se necesita la identificación de especie, probar paneles amplios de antibióticos y saber cuáles fenotipos son inusuales.
Las distintas BLEE confieren un grado de resistencia muy variable. La intensidad de hidrólisis de un determinado antibiótico difiere según las cepas consideradas, pudiendo incluso no tener efecto fenotípicamente detectable, en algunos casos únicamente tiene lugar una reducción de los halos de inhibición o aumento de los valores de la concentración mínima inhibitoria (CMI), pero permanece en el intervalo de sensibilidad. No es infrecuente que preliminarmente se informe un aislamiento de una enterobacteria como sensible a cefalosporinas de tercera generación y aztreonam y que posteriormente, por pruebas adicionales o ante un fracaso clínico, se constate que se trata de una cepa portadora de BLEE. Por eso ante el aislamiento de una cepa de un Gram negativo con unas CMI de cefalosporinas de tercera generación elevadas con respecto a lo esperado para dicha cepa, o ante el hallazgo de multirresistencia en las pruebas de sensibilidad, es prioritario descartar la presencia de BLEE, según los criterios recomendados por la Clinical and Laboratory Standards Institute (CLSI).7, 9,10, 11
Las pruebas fenotípicas presentan limitaciones que convienen tener en consideración, como la hiperproducción de algunas betalactamasas por ciertos microorganismos, como Enterobacter cloacae, Enterobacter aerogenes, Citrobacter freundii, Serratia marcescens, Morganella morganii, Providencia spp. que producen ß-lactamasas cromosómicas inducible de clase 1 ó AmpC, que puede llevar a errores de identificación como se ha mencionado.2,7,8,9.
Anteriormente, se defendía la utilización de la cefpodoxima en las pruebas de sensibilidad como mejor sensor para sospechar la presencia de BLEE. Sin embargo, este antibiótico puede verse también afectado por la hiperproducción de AmpC o por alteraciones de la permeabilidad. El mejor sistema es aquel que utiliza varias cefalosporinas simultáneamente y emplea criterios habituales en la lectura interpretada del antibiograma. En la actualidad, los sistemas automáticos para la determinación de la sensibilidad a los antimicrobianos han recogido esta experiencia y tienden a incluir simultáneamente cefotaxima y ceftazidima con ácido clavulánico e iguales recomendaciones deben establecerse con los discos o el E-test® con el ácido clavulánico.2,9,12
Es posible que una misma cepa origine distintas betalactamasas, pudiéndose determinar, por ejemplo, microorganismos con BLEE de tipo CTX-M y SHV o BLEE de tipo CTX-M y con betalactamasas cromosómicas AmpC, por lo que los fenotipos pueden variar respecto a los esperados. Se recomienda evaluar en la detección de cepas BLEE con fenotipos complicados (escasa hidrólisis de algunos de los substratos o superposición con otros mecanismos de resistencia) el empleo de sistemas automatizados, técnicas especiales (isoelectroenfoque, reacción en cadena de la polimerasa [PCR], secuenciación, etcétera).7, 9,12
Elementos a considerar para desarrollar el tamízaje de BLEE
- Se requiere la utilización de, al menos, los discos de cefotaxima (o cefriaxona) y ceftazidima para detectar con mayor eficiencia la presencia de BLEE.
- Ceftazidima detecta más eficientemente las BLEE derivadas de TEM, SHV y PER-2; mientras que cefotaxima detecta mejor las de tipo CTXM.
- La resistencia a las cefalosporinas de tercera generación con sensibilidad a cefoxitina (no hidrolizada por las BLEE) es útil para diferenciar BLEE de AmpC, considerándose un buen indicador de la presencia de BLEE.7,8
Métodos para detectar BLEE en Enterobacterias
La mayoría de los métodos descritos para detectar microorganismos productores de BLEE han sido diseñados para enterobacterias y se fundamentan en el carácter inhibible de estas enzimas por los inhibidores de ß-lactamasas. Sin embargo, la aplicación de cualquiera de estos métodos debe ir precedida de un riguroso análisis del perfil de sensibilidad a los antimicrobianos, con los criterios habituales de lectura interpretada del antibiograma, que permita detectar fenotipos compatibles con su presencia.
Métodos fenotípicos
A. Métodos basados en la utilización de inhibidores de ß-lactamasas
Técnica de la doble difusión con discos: se basa en la sinergia de doble disco. Se utiliza una placa de agar Mueller-Hinton inoculada con una suspensión bacteriana sobre la que se colocan los discos de cefalosporina y el disco con el inhibidor de betalactamasa a determinada distancia (30 mm o 20 mm si se desea aumentar la sensibilidad) de los discos de ácido clavulánico. Si aparece una ampliación entre los halos de inhibición en alguno de los antimicrobianos y el disco con el inhibidor de betalactamasa se considera que existe BLEE.
En las enterobacterias productoras de AmpC inducible, puede utilizarse la aproximación de cefepima (CFP) _ ampicilina/ ácido clavulánico (AMC), para observar el efecto de ampliación del halo de inhibición.10,11,13, 14

B. Prueba de combinación de discos
Utilización de discos con cefalosporinas de 3º generación, sola y con ácido clavulánico. Actualmente, se cuenta con discos de ceftazidima/ac. Clavulánico (30/10mg), cefotaxima/ac. clavulánico (30/10mg) y cefpodoxima/ac. clavulánico (10/10 mg). Se confirma la presencia de BLEE cuando el halo de inhibición de la combinación es = 5 mm respecto de la cefalosporina sola.10, 11,13,14
Sinergia con inhibidores de betalactamasas mediante la determinación de las CMI
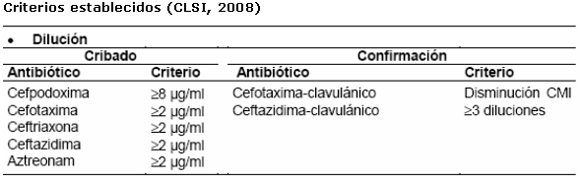
Técnica de la sinergia con inhibidores de betalactamasas: consiste en la determinación de la CMI por macro o microdilución de cefalosporinas de tercera generación, con y sin inhibidor de ß-lactamasas. Conviene realizarla con más de un antibiótico. La existencia de BLEE queda confirmada ante la reducción de la CMI en tres diluciones (ocho veces) en presencia de ácido clavulánico.5,7,10,11
C. Técnica de E-test
Se emplean tiras de papel impregnadas con antibióticos. Una mitad contiene cefalosporina en concentración decreciente y la otra mitad cefalosporina también en concentración decreciente con ácido clavulánico con una concentración fija (2 µg) por cada concentración. Se considera positiva la sinergia con el ácido clavulánico cuando la CMI disminuye en dos o más diluciones. Se recomienda utilizar tiras de ceftazidima y cefotaxima ya que todas las enzimas no hidrolizan por igual estos antibióticos. En caso de sospecha de AmpC, colocar tira con cefepima.5, 6, 10
Métodos de Bioensayos
La utilización de bioensayos, como la prueba de Masuda o el método tridimensional, pueden ser adecuados para demostrar la presencia de estas enzimas en cepas en las que existan dudas acerca de su producción. Ambas pruebas, dependiendo de los substratos que se utilicen, son útiles para cualquier tipo de ß-lactamasa. A pesar de su versatilidad, son engorrosas de realizar y han tenido poco éxito. La prueba de Masuda, consiste en la disposición de un microorganismo indicador sensible (generalmente E. coli ATCC 25922) a los substratos hidrolizados (en este caso cefotaxima, ceftazidima o aztreonam) por la ß-lactamasa que se pretende detectar. Estos substratos se colocan en un disco (sirven los discos comerciales) y en las zonas marginales del halo de inhibición, discos de papel impregnados con el extracto, generalmente obtenido por sonicación, del microorganismo a estudiar. La distorsión de halo de inhibición en el microorganismo sensor nos indica la presencia de la BLEE. Aparte de la laboriosidad, tiene el inconveniente de que se obtienen resultados poco interpretables cuando existe más de un enzima en el microorganismo a estudiar, sobre todo, en los casos en los que las ß-lactamasas compartan substratos en su hidrólisis, como es el caso de los productores de AmpC que supuestamente produzcan una BLEE. El método tridimensional es igualmente engorroso de realizar, pero tiene la ventaja de utilizar diferentes substratos. Este hecho permite, al menos cualitativamente, determinar el perfil de substrato del enzima presente en el microorganismo a estudiar. Consiste en la disposición de un microorganismo sensor sensible a los antibióticos ß-lactámicos (generalmente E. coli ATCC 25922), realizar un surco (puede ser circular) en el que se dispone el extracto sonicado del microorganismo a estudiar y colocar discos con antibióticos a una distancia de unos 3 mm del surco. La distorsión de los halos de inhibición nos indicará el perfil de substrato del enzima.5,6
Métodos bioquímicos
La mayoría de estos métodos se aplican una vez confirmada la presencia de la BLEE en el aislado correspondiente y sirven para caracterizar dicha enzima. Los métodos bioquímicos incluyen el isoelectroenfoque, el análisis del perfil de substrato, la cinética enzimática y la determinación de los IC50 para diferentes inhibidores de ß-lactamasas. En la actualidad, se utiliza poco debido a la descripción de diferentes BLEE que comparten idéntico puntos isoeléctricos. Sin embargo, es muy útil si se combina con el análisis del fenotipo de sensibilidad ya que puede orientar el tipo de BLEE para un análisis posterior del perfil de substrato o estudio molecular.6,10
Métodos genotípicos
Permiten identificar las BLEE y llevar a cabo la investigación epidemiológica, saber que los brotes son epidemiológicamente complejos, pudiéndose tratar de la proliferación clonal de una cepa productora de una única BLEE, pero también diseminarse diversas BLEE en el mismo brote, por la proliferación clonal de varias cepas con distintas BLEE o por la existencia de diferentes plásmidos entre los miembros de una misma cepa. Adicionalmente, microorganismos no relacionados genotípicamente pueden producir la misma BLEE mediante transferencia plasmídica y la misma BLEE puede ser mediada por plásmidos distintos. La capacidad de propagación de las BLEE es extraordinaria y, de hecho, se ha comprobado la transmisión interhospitalaria, interurbana e incluso entre países. De lo que se deduce que toda precaución para controlar este fenómeno es poca. Los métodos más utilizados son el perfiles plasmídicos, electroforesis en gel de campos pulsados (PFGE), ribotipificación y otros métodos basados en la reacción en cadena de la polimerasa (PCR). El análisis de restricción de fragmento amplificado del ADN ribosómico (PCR-RFLP) y la amplificación polimorfa del ADN (AFLP, Amplified Fragment Length Polymorphism) parecen resultar de especial utilidad en este contexto.4,5,10,13
Interpretación de los resultados
- Cuando no se confirma la presencia de BLEE, utilizar los puntos de corte generales para enterobacterias, independientemente del género aislado.
- Cuando se confirme fenotípicamente la presencia de BLEE, deberán informarse resistentes a todas las penicilinas, cefalosporinas y aztreonam.
- Las combinaciones de beta-lactámicos con IBL pueden ser activas in vitro, pero su eficacia clínica es discutida y depende de la localización de la infección, particularmente cuando el microorganismo se encuentra en altas concentraciones.7,8
CONCLUSIONES
Los métodos de detección de enterobacterias productoras de BLEE deben comenzar por una adecuada interpretación de los perfiles de sensibilidad con los criterios habituales de lectura interpretada del antibiograma. Posteriormente, se deben elegir métodos de confirmación basados en la inhibición del enzima por los inhibidores de ß-lactamasa, generalmente utilizando ácido clavulánico. Deben emplearse varios substratos, esencialmente ceftazidima y cefotaxima o ceftriaxona, para permitir la detección de enzimas con baja capacidad hidrolítica para alguno de los substratos. En el caso de las enterobacterias productoras de AmpC, la cefepima constituye el substrato que mejor detecta la presencia de estas enzimas. La adecuada detección de los microorganismos productores de BLEE es esencial para conocer la verdadera dimensión del problema que representan, limitar su diseminación y adecuar las escasas opciones terapéuticas.
REFERENCIAS BIBLIOGRÁFICAS
1. Guillermo Prada GT. ß-lactamasas de espectro extendido: Perspectivas y Tratamiento. Publicada por la asociación Panamericana de Infectología. Revista panamericana de Infectologia. Enero-diciembre de 2002; 5 (1). [Sitio de Internet] 2002. Disponible en: http://www.encolombia.com/medicina/infectologia/panamericana5102 -blactamasas.htm [Acceso 2 de Marzo 2010].
2. Carrillo RA, García BA. Betalactamasas de Espectro Extendido importancia clínica. Taller del Laboratorio Clínico. Asociación Española de Biopatología Médica. Curso 2007 - 2008 Nº 2 Page 2. [Sitio de Internet] 2008. Disponible en:
http://www.aebm.org/.../distancia%202007.../2.-%20betalactamasas.pdf [Acceso 22 de Marzo 2010].
3. Cantón R, Oliver A, Coque TM, Varela MC, Pérez-Diaz JC, Baquero F. Epidemiology of extended-spectrum ß-lactamase-producing Enterobacter isolates in a Spanish hospital during a 12-year period. J Clin Microbiol. 2002; 40:1237-1243.
4. Oliver A, Cantón R. Enterobacterias productoras de ß-lactamasas plasmídicas de espectro extendido [Sitio de Internet] 2002. Disponible en: http://www.seimc.org/control/revi_Bacte/pdf/Blees.pdf [Acceso 2 de Marzo 2010].
5. Sánchez AB. Betalactamasas de espectro extendido (BLEE) Revista Electrónica de Medicina Intensiva. Agosto 2004; 4(8).
6. Pérez J, Gimeno C. ß-lactamasas plasmídicas de espectro ampliado: Experiencia del programa de control de calidad, dos años después. Servicios de Microbiología, Ciudad Sanitaria y Universitaria de Bellvitge, Hospitalet de Llobregat, y Hospital Clínico Universitario, Valencia [Sitio de Internet] 2008. Disponible en:http://www.seimc.org/control/revi_Bacte/bleas.htm [Acceso 22 de Marzo 2010].
7. Navarro RF, et al. Lectura interpretada del antibiograma de enterobacterias. En puesta al día en métodos microbiológicos para el diagnóstico clínico. [Sitio de Internet] 2002. Disponible en: http://www.external.elsevier.es/espacioformacion/eimc/temas /m2t2.pdf [Acceso: 2 de Marzo 2010]. 8. Navarro RF, Miró CE, Mirelis OB. Lectura interpretada del antibiograma de enterobacterias. Enferm Infecc Microbiol Clin. 2002; 20(5):225-34. 9. Truppia LA, Mollerach A, di Conza JA. Comparación de tres métodos microbiológicos para la detección de betalactamasas de espectro extendido en enterobacterias aisladas en Santa Fe (Argentina). Enfermedades infecciosas y microbiología clínica. 2005; 23(9): 525-528. 10. Famiglietti A, Quinteros M, Vázquez M, Marín M, Incola F, Radice M, et al. Consenso sobre las pruebas de sensibilidad a los antimicrobianos en Enterobacteriaceae. Rev. argent. microbiol. 2005; 37(1) [Sitio de Internet] 2005. Disponible en: 11. Clinical and Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing: Eighteenth Informational Supplement. 2008;28 (1):M100-S18. 12. Treviño M, Martínez-Lamas L, Romero-Jung P, Varón C, Moldes L, et al. Comparación entre las pruebas para la detección de betalactamasas de espectro extendido de los sistemas Vitek2 y Phoenix Enferm Infecc Microbiol Clin. 2009. [Sitio de Internet] 2009. Disponible en: http://www.elsevier.es/eimc [Acceso 22 de Marzo 2010]. 13. Perozo M, Armindo J, Castellano G, Maribel J. Detección de Betalactamasas de Espectro Extendido en cepas de la familia Enterobacteriaceae. Kasmera. [online]. jun. 2009;.37(1): [citado 22 Marzo 2010].25-37. Disponible en la World Wide Web: http://www.scielo.org.ve/scielo.php?script=sci_arttext&pid=S0075 -52222009000100004&lng=es&nrm=iso ISSN 0075-5222. 14. Zemelman R, Valenzuela L, Dominguez M, Bello H, González G, et al. Detección de ß-lactamasas de espectro extendido en el laboratorio de microbiología. Rev Chil Infect. (2002); 19 (Supl. 2):92-95.
http://www.scielo.org.ar/scielo.php?pid=S0325 -75412005000100008&script=sci_arttext [Acceso: 22 de Marzo 2010].














