INTRODUCCIÓN
Las plantas, como organismos sésiles, son capaces de responder a una diversidad de estímulos abióticos y bióticos (Samach y Wigge, 2005; Knudsen y otros, 2018), estas respuestas pueden tener efectos sobre los parámetros morfológicos observables, tales como la longitud de raíz o del vástago (Hanci y Cebeci, 2015; Grossi y otros, 2021), como en parámetros bioquímicos tales como la cuantificación del contenido de clorofila, de ácido ascórbico, prolina y del azúcar soluble total, entre otros (Tripathi y Gautam, 2007; Rahneshan, Nasibi y Moghadam, 2018).
Los datos recolectados u obtenidos en una investigación con plantas deben ser necesariamente analizados estadísticamente e interpretados con criterio técnico y científico. El lenguaje de programación R permite la reproducibilidad en el análisis de datos mediante el uso de funciones dentro de herramientas llamadas paquetes, los cuales son desarrollados por la comunidad (Tippmann, 2015; Vuorre y Crump, 2021). Una función en el lenguaje R es un objeto que contiene tres elementos: un argumento, un cuerpo y un ambiente, y está programado para dar una respuesta de salida basándose en los elementos de entrada que se le otorgue. El argumento es un conjunto de condiciones de entrada, ya sean datos o parámetros que el usuario necesite procesar. El cuerpo contiene una o más líneas de código que detallan el algoritmo de procesamiento de los datos, utilizando las condiciones provistas en el argumento, y finalmente, el ambiente es el lugar virtual donde se desarrolló la función, y contiene todos los elementos que ésta necesita para operar correctamente (R Core Team, 2017).
Los paquetes pueden encontrarse en distintos repositorios en línea, por lo que son de fácil acceso y pueden ser utilizados de manera gratuita (Salas, 2008). El tipo de licencia del paquete es importante ya que permite la distribución o la utilización de los mismos bajo ciertas condiciones. Las licencias GPL son de código libre y pueden ser utilizadas sin ningún costo (Wolf, Miller y Grodzinsky, 2009), a su vez, las licencias MIT tampoco tienen restricciones, y se puede utilizar o copiar el paquete siempre que se haga mención al mismo en los derechos de autor (Saltzer, 2020).
En el presente trabajo se presenta un paquete del tipo MIT diseñado para el análisis de variables biométricas. Para su validación se llevó a cabo un ensayo con plántulas de Arabidopsis thaliana crecidas in vitro en condiciones control y bajo estrés salino u osmótico, con el fin de recolectar datos de la longitud de la raíz primaria durante una semana y observar cuando se produce una diferencia significativa en el desarrollo.
MATERIALES Y MÉTODOS
Desarrollo y aplicación del paquete
Diseño del paquete
Para desarrollar el paquete se utilizó el lenguaje de programación R, a través de la interfaz gráfica RStudio (R Core Team, 2021), y se creó una cuenta en el repositorio GitHub (Winnie, 2021). Mediante RStudio se descargó el paquete roxygen2 para generar los scripts de Plantico así como la descripción del mismo. Este paquete permite realizar el Análisis de Varianza (ANOVA) y el test de Tukey para comparaciones múltiples de datos experimentales. Los supuestos teóricos de Normalidad y Homocedasticidad se evaluaron con las pruebas de Shapiro-Wilk y Levene, respectivamente (Parra-Frutos, 2016).
Recolección de datos a partir de plántulas de Arabidopsis thaliana cultivadas in vitro bajo condiciones control o de estrés abiótico
Se desinfectaron las semillas de Arabidopsis thaliana Columbia (Col-0) con etanol al 70% por 15 minutos y etanol al 96% por 5 minutos y posteriormente se realizaron 3 enjuagues con agua destilada estéril. Luego de vernalizar durante dos días a 4°C, se cultivaron in vitro por una semana en el medio de cultivo MS 0,5X con la adición de 0,8% (m/v) de agar (Murashige y Skoog, 1962) con fotoperíodo de 16 horas de luz y 8 de oscuridad a una temperatura de 21 a 23°C. Se seleccionaron aquellas plántulas que presentaban un crecimiento similar y se establecieron en placas de Petri (Grossi, y otros, 2020). Para el tratamiento control se utilizaron 3 placas con MS 0,5X y para los tratamientos de estrés abiótico se prepararon 4 placas con MS 0,5X conteniendo 75 mM NaCl (salino) o 100 mM manitol (osmótico). Se colocaron 4 plántulas por placa; la medición de la longitud de la raíz primaria de cada plántula fue realizada diariamente durante 7 días tanto a las plantas control como a las tratadas utilizando el software ImageJ (Rueden y Eliceiri, 2019).
Utilización del paquete mediante datos obtenidos del ensayo de estrés abiótico
Los datos obtenidos fueron guardados con el nombre de “DatosTabla” en una tabla en formato Excel, con las columnas Tratamiento, Tiempo y Longitud. Estos fueron posteriormente cargados al ambiente de RStudio. Las columnas Tratamiento y Tiempo fueron importadas como datos de carácter y la columna Longitud como dato numérico. Las funciones del paquete Plantico fueron llamadas de la siguiente manera:
>Resumentiempo_t (DatosTabla, t)
>Tabla_t_A(DatosTabla, t)
>Tabla_tukey_A(DatosTabla, t)
>Graficadebarratiempo_t(DatosTabla, t)
>Grafico_lineas_tiempo_t(DatosTabla, t)
Donde t representa los diferentes tiempos de medición del ensayo experimental con plantas.
RESULTADOS Y DISCUSIÓN
Se logró crear el paquete “Plantico”, el cual consta de 5 funciones para obtener los estadísticos descriptivos de los datos del análisis de estrés abiótico en plantas. Estas 5 funciones pudieron ser descargadas junto con Plantico directamente desde el repositorio de GitHub previa descarga del paquete devtools (Wickham y otros, 2020). Cuando se instaló Plantico, el paquete importó a otros paquetes que ayudan al análisis de datos tales como readxl que hace uso de los datos de Microsoft Excel importados al entorno de R (Wickham y Bryan 2019), dplyr (Mailund, 2019) que permite trabajar con datos tabulados de manera sencilla (Wickham, 2021), psych que utiliza la función describeBy para agrupar los datos por tratamiento y calcular su respectiva media, mediana y otros parámetros (Revelle, 2021). Para poder realizar gráficos el paquete utiliza a ggplot2 (Wickham 2016). Todos estos paquetes poseen sus respectivas licencias de uso. Los paquetes devtools y psych tienen licencia GPL≥ 2, readxl tiene licencia GPL 3 y los paquetes ggplot2 y dplyr tienen licencia MIT por lo cual se pueden utilizar de manera libre y gratuita. La licencia de Plantico es del tipo MIT, y se escogió este tipo de licencia para que cualquier usuario interesado lo pueda utilizar (Kapitsaki, Kramer y Tselikas, 2017). Cabe destacar que también el lenguaje R está disponible como software libre bajo los términos de licencia GNU (Chen y Peace, 2013) lo cual constituye una ventaja respecto a otros softwares estadísticos que son pagos. Si bien el lenguaje R cuenta con funciones de base que pueden realizar muchos análisis estadísticos, el paquete desarrollado simplifica el análisis de los datos con el uso de pocas funciones que permiten generar gráficos de barras, de líneas y de cajas, sujetos a un tiempo “t” modificable por el usuario. Todas las funciones generadas requieren dos argumentos para su uso: los datos a ser procesados en forma tabular y el tiempo del que se desea obtener el resultado de la función. Esto último permite que se puedan hacer análisis a lo largo del experimento y no solo en el tiempo final. Posterior al desarrollo e instalación del paquete se realizó la aplicación del mismo usando los datos experimentales del cultivo in vitro de plántulas de Arabidopsis. En la Figura 1 se observa que las plantas que tuvieron mayor crecimiento de raíces al final del ensayo fueron las de la condición control.
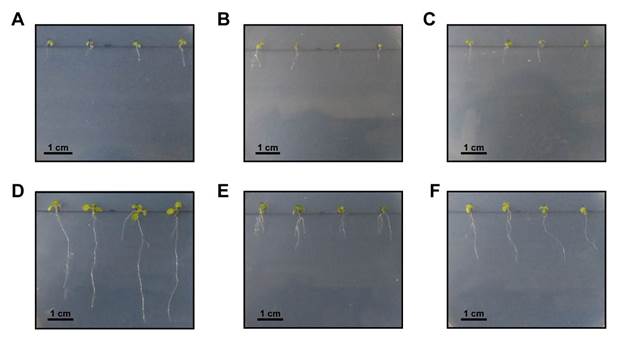
Fig. 1 Imágenes representativas de plántulas de A. thaliana cultivadas en condiciones control (MS 0,5X; A y D) o bajo estrés osmótico (MS 0,5X + 100 mM manitol; B y E) o salino (MS 0,5X con 75 mM NaCl; C y F) a diferentes tiempos: día 1 (A-C), día 7 (D-F) .
Luego de cargar los datos de longitud de raíces en el archivo Excel se los importó al ambiente de RStudio y se usó el paquete Plantico. Para visualizar los datos como un gráfico de líneas con la desviación estándar como sombreado, se utilizó la función >Grafico_lineas_tiempo_t capaz de plasmar los resultados obtenidos a lo largo de 7 días. Como se observa en la Figura 2, en todos los tiempos del ensayo, las plantas que presentaron mayor longitud de raíces fueron las de la condición control, seguido por las condiciones de estrés con NaCl y manitol, respectivamente.
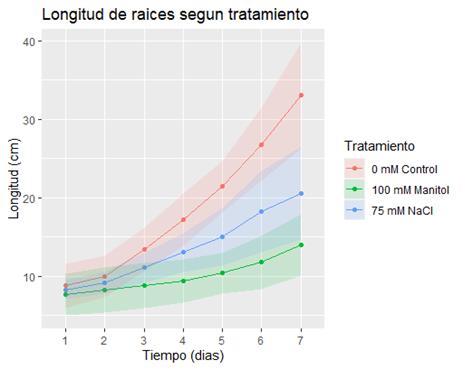
Fig. 2 Gráfica de líneas correspondiente al ensayo con los distintos tratamientos por el periodo de 7 días.
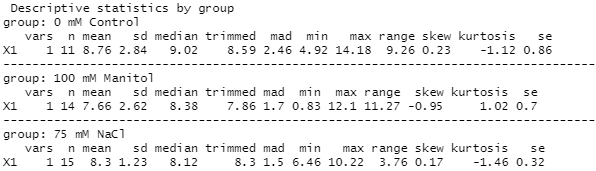
Para obtener datos resumidos de los estadísticos más comunes se creó la función Resumentiempo_t, esta función procesó los datos del tiempo “t” y devolvió la cantidad de variables analizadas (vars), la cantidad de muestras por grupo (n), la media, la desviación estándar (sd), la mediana (mean), el valor mínimo (min) y máximo (max), el rango (range), el error estándar (SE), la media truncada (trimmed), la desviación absoluta media (mad) y los coeficientes skew y kurtosis. Como se observa en la Figura 3 la función ya separa los datos en grupos de acuerdo a los tratamientos del estudio.
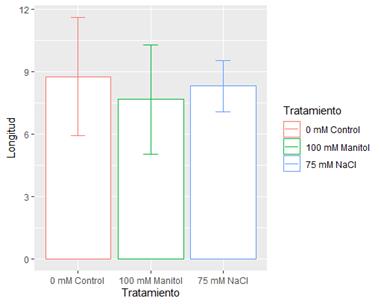
Para realizar un gráfico de barra con desviación estándar, se utilizó la función >Graficadebarratiempo_t. En la Figura 4 se pueden observar los resultados del tiempo 1, sin embargo, también se podría utilizar para el tiempo que el usuario necesite reemplazando por el tiempo t.
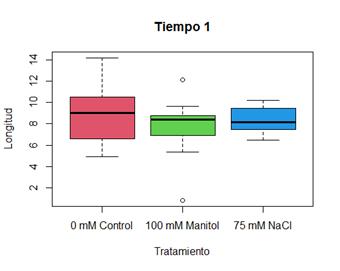
El análisis de varianza de los resultados en cada tiempo puede ser de interés particular, es por ello que se diseñó una función que permita realizar el ANOVA entre tratamientos como se observa en la Figura 5 A. El ANOVA es un método paramétrico que necesariamente requiere el cumplimiento de algunos supuestos teóricos como normalidad y homocedasticidad. Existen diversidad de pruebas para verificar los suposiciones del ANOVA, desde métodos gráficos donde se visualiza el comportamiento de datos hasta métodos formales de contraste, en este caso se utilizó la combinación de las pruebas formales de Shapiro-Wilk y Levene debido a su amplia utilización como métodos de verificación dentro de la comunidad científica, no obstante existen otras combinaciones que podrían tener mayor robustez como la combinación de Shapiro-Wilk seguido por las pruebas de Bartlett y Brown-Forsythe (Parra-Frutos,2016 ; Kozak y Piepho, 2018). A continuación se aplicó el test de Tukey que permite realizar comparaciones múltiples. En la Figura 5B, el análisis de los valores de p indicó que en el tiempo 1 no hay una diferencia significativa entre los tratamientos.
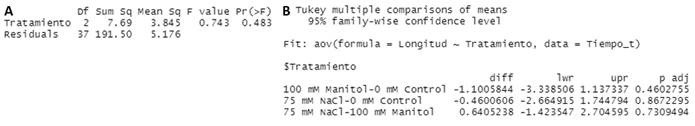
Fig. 5 (A) Resultados de ANOVA mediante uso de la función >Tabla_t_A para el tiempo 1. (B) Resultados del test de Tukey al utilizar la función >Tabla_tukey_A para el tiempo 1.
La función de Plantico que realiza el ANOVA también permite realizar el diagrama de cajas como se visualiza en la Figura 6. Este tipo de diagrama facilita la representación gráfica de la variable cuantitativa (tal como longitud) y distinguir los grupos numéricos a través de cuartiles. Además, se pueden observar los valores mínimos y máximos.
Una vez obtenidos los resultados se procedió a copiarlos en un archivo como lo expuesto en la Tabla 1, y se estableció la significancia entre los distintos tratamientos.
Tabla 1 Resultados obtenidos del análisis de varianza luego de procesar los datos con el paquete Plantico.
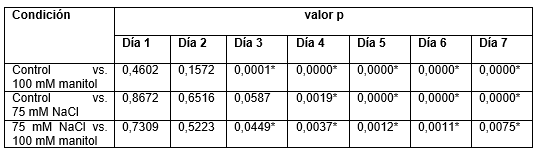
Se establece que hay diferencia significativa para valores de p < 0,05 que son indicados con *.
Este tipo de estudio permite observar los efectos de la sal en las plantas incluyen estrés osmótico y toxicidad de iones (Qi y Zhang, 2020). El desbalance de nutrientes y las deficiencias ocasionan daño en las membranas celulares, disminuyen la expansión, generan cambios en procesos metabólicos, estrés oxidativo y genotoxicidad. Generalmente, la fase temprana del crecimiento vegetativo de plántulas es más sensible que las fases tardías. El sistema radicular no se desarrolla en forma óptima, es menos profundo, menos ramificado, pesa menos y es menos eficiente en la absorción de agua y nutrientes. Se reportó que el estrés salino disminuye la división celular epidérmica de la raíz y la tasa de alargamiento, reduciendo el crecimiento de raíces primarias e inhibiendo la iniciación lateral (Julkowska y otros, 2017).
Este paquete permitió el análisis estadístico de los datos recopilados a partir de plántulas de A thaliana que fueron sometidas a estrés salino y osmótico. Las figuras 3, 4, 5 y 6 corresponden al tiempo 1, momento en el que no se observaron diferencias entre los tratamientos. Sin embargo, como se observa en las Figura 1 y Figura 2 y en la Tabla 1, se observó un acortamiento significativo (p < 0,05) en la longitud de las raíces, a partir del día 3 del ensayo, entre la condición control y tratamiento con manitol y también entre manitol y NaCl. A partir del día 4, también se observan diferencias entre el control y el tratamiento con NaCl. Estas diferencias se mantienen hasta el final del ensayo (día 7). Los datos reportados en este trabajo son concordantes con los de otros autores (Van der Does y otros, 2017; Nath y otros, 2019) y con lo reportado previamente por nuestro grupo (Grossi y otros, 2020).
CONCLUSIONES
Este paquete de uso libre fue desarrollado con el fin de ayudar al análisis estadístico de resultados experimentales. El estrés salino y el estrés osmótico ocasionaron una reducción en la longitud de la raíz primaria y el análisis de los datos con el paquete Plantico permitió identificar el momento a partir del cual las diferencias observables fueron significativas. Plántico podría ser utilizado en otros ensayos de estrés.