Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina
versión impresa ISSN 0034-7523
Rev cubana med vol.51 no.4 Ciudad de la Habana oct.-dic. 2012
TEMA ACTUALIZADO
MicroRNAs en el cáncer: de la investigación a la práctica clínica
MicroRNAs in cancer - from research to the clinical practice
MSc. Maydelín Frontela Noda
Instituto Nacional de Oncología y Radiobiología. La Habana, Cuba.
RESUMEN
Los microRNAs (miRNAS) son una clase de RNAs no codificantes que actúan como reguladores de la expresión génica. Las secuencias de muchos de ellos están conservadas entre organismos no relacionados, lo cual sugiere que estas moléculas participan en procesos biológicos esenciales. Frecuentemente, la expresión de los miRNAs está alterada en muchos tipos de cánceres humanos y resultan críticos en la carcinogénesis, ya que funcionan como genes supresores tumorales o como oncogenes. Estudios recientes han demostrado que los miRNAs están muy relacionados con la progresión del cáncer, que incluye el crecimiento del tumor, la diferenciación, la adhesión, la apoptosis, la invasión y la formación de metástasis. En esta revisión se presenta la investigación básica y traslacional que ha emergido en la última década, un período que puede ser considerado como el de la "revolución de los miRNAs" en la oncología molecular. En ella se incluyen las características de los miRNAs, su biogénesis y procesamiento, sus funciones y los mecanismos que alteran su expresión en cáncer. Los miRNAs han generado un gran interés por sus aplicaciones potenciales en medicina, por lo que, además, se refieren sus perspectivas futuras en el diagnóstico, pronóstico y terapia del cáncer.
Palabras clave: miRNAs, cáncer humano, diagnóstico, pronóstico, terapia.
ABSTRACT
The microRNAs (miRNAs) are a type of non-coding RNAs that act as gene expression regulators. The sequences of many of them are preserved into unrelated organisms, all of which suggests that these molecules do participate in essential biological processes. The miRNA expressions are often altered in many types of human cancers and they are critical to carcinogenesis, since they work as tumor suppressor genes or as oncogenes. Recent studies have proved that miRNAs are very associated to the cancer progression including the growth of tumor, the tumoral differentiation, the adhesion, the apoptosis, the invasion and the formation of metastasis. This literature review presented the basic and translational research carried out in the last decade, a period that may be called "the revolution of miRNAs" in the molecular oncology. It covered the characteristics, the biogenesis and processing and the functions of miRNAs as well as the mechanisms that modify their expression in cancer. The miRNAs have aroused great interest because of their potential applications in the field of medicine; therefore, the future perspectives in the diagnosis, prognosis and treatment of cancer by using miRNAs were also addressed.
Key words: miRNAs, human cancer, diagnosis, prognosis, therapy.
INTRODUCCIÓN
La transformación maligna es un proceso que involucra alteraciones genéticas, como las mutaciones puntuales y las anomalías cromosómicas, y alteraciones epigenéticas, como la metilación del ADN, las modificaciones postraduccionales de las histonas y la hipermetilación de los promotores. Todas estas variaciones influyen en el crecimiento celular, la apoptosis y la diferenciación. En etapas más avanzadas de la carcinogénesis, otros cambios pueden promover la angiogénesis, la invasión de los tejidos adyacentes y la metástasis en sitios distantes. Los genes más conocidos, que se relacionan con el cáncer, codifican para proteínas y se clasifican en oncogenes y genes supresores de tumores. La identificación de los cambios genéticos y epigenéticos que afectan sus funciones es lo que ha permitido el reconocimiento de su asociación con el cáncer humano.
Sin embargo, una de las grandes sorpresas de la biología moderna fue el descubrimiento de que solamente alrededor de 2 % de los genes que conforman el genoma humano codifica para proteínas.1 En años recientes, con la introducción de las técnicas de alto procesamiento para el estudio de la expresión génica como los microarrays y la secuenciación del transcriptoma completo, se determinó que al menos 90 % del genoma se transcribe activamente2,3 y que el transcriptoma humano es más complejo que el conjunto de genes que codifican proteínas, ya que exhibe una cuantiosa expresión de RNAs no codificantes.4-6 Entre ellos se encuentran los microRNAs (miRNAs), los cuales participan en los mecanismos de regulación génica7,8 y se ha demostrado que su expresión alterada desempeña un papel importante en la transformación maligna de las células humanas.9
CARACTERÍSTICAS DE LOS miRNAs
Los miRNAs son moléculas de RNA pequeñas (aproximadamente 22 nucleótidos) que están conservadas a través de la evolución y que participan en el silenciamiento postranscripcional de los genes. Están presentes en organismos multicelulares y en virus. Se localizan en todos los cromosomas humanos, excepto en el cromosoma Y. Cerca del 50 % se encuentran agrupados y la transcripción es policistrónica. Se localizan frecuentemente en sitios frágiles, y en regiones de amplificación o pérdida de heterocigocidad, asociadas a cáncer. Pueden estar situados en regiones intergénicas o en intrones de genes que codifican proteínas, con menor frecuencia residen en los exones, pero con una orientación anti-sentido con respecto al gen que codifica proteínas.10
Se estima que 1-5 % del genoma humano corresponde a los miRNAs, lo cuales pueden regular al menos 30 % de los genes que codifican proteínas.11 Cada miRNA puede interactuar con cientos de RNA mensajeros (RNAm)12 ya sea directa o indirectamente. De igual forma, la expresión de un simple RNAm puede ser modulada cooperativamente por múltiples miRNAs.13
BIOGÉNESIS Y PROCESAMIENTO DE LOS miRNAs
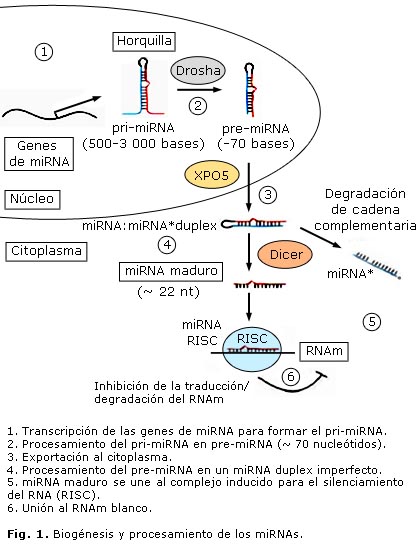
La biogénesis de los miRNAs comienza en el núcleo con la transcripción de los genes correspondientes por la RNA polimerasa II14 o III15 para dar lugar a un miRNA primario (pri-miRNA), que está poliadenilado y tiene unido un grupo 7-metil-guanilato (m7G) en el extremo 5´, lo que se conoce como caperuza.16 Subsecuentemente, este transcrito primario se procesa por la enzima Drosha17,18 para formar un miRNA precursor (pre-miRNA), de aproximadamente 70 nucleótidos, que se asemeja a una horquilla. Este es transportado hacia el citoplasma por la exportina-5 (XPO5), donde la enzima Dicer lo procesa en un miRNA duplex de 22 nucleótidos. Una de las cadenas del duplex interactúa con el complejo inducido para el silenciamiento del RNA (RISC, por sus siglas en inglés), y de esta manera interactúa con el RNAm blanco19 (fig.), mientras que la cadena complementaria se degrada. Existen 2 mecanismos de regulación negativa de los RNAm por los miRNAs. El hecho de que ocurra uno u otro está regido por el grado de complementariedad entre el miRNA y el RNAm blanco. Si esta es imperfecta se reprime la traducción del RNAm. Los sitios de complementariedad para los miRNAs que utilizan este mecanismo están localizados en las regiones que no se traducen, ubicadas hacia el extremo 3´ del RNAm (regiones 3´UTR, por sus siglas en inglés). Si por el contrario, la complementariedad es perfecta, se induce un corte y posteriormente ocurre la degradación del RNAm. Los miRNAs que utilizan este mecanismo encuentran sus sitios de complementariedad en secuencias codificantes de los RNAm.20
FUNCIONES DE LOS miRNAs
Hasta la fecha se han identificado más de 1 000 miRNAs en seres humanos, a través de los experimentos de clonación y la utilización de la bioinformática. Ellos participan en la regulación de numerosas vías metabólicas y celulares, notablemente en aquellas que controlan las modificaciones que ocurren durante el desarrollo, la embriogénesis, la preservación de las células madre, la diferenciación de las células hematopoyéticas y el desarrollo del cerebro.21 La expresión alterada de los miRNAs es probable que contribuya a la enfermedad humana, y entre otros procesos, se ha relacionado con la progresión tumoral,22 que incluye el crecimiento del tumor, la diferenciación, la adhesión, la apoptosis, la invasión y la formación de metástasis.
Los primeros reportes sobre los miRNAs fueron en el gusano Caenorhabditis elegans, donde las investigaciones condujeron a la descripción de los genes lin-423 y let-724, que codifican para RNAs pequeños que están relacionados con cambios del desarrollo en esa especie. Hasta ese momento, se creía que era un fenómeno exclusivo en nemátodos, pero se realizó una serie de trabajos que permitieron identificar y clonar miRNAs de diferentes organismos, incluyendo los humanos, y se descubrió que las secuencias nucleotídicas estaban filogenéticamente conservadas.25-28
La primera evidencia que relaciona los miRNAs con el cáncer proviene de un estudio realizado en pacientes con leucemia linfocítica crónica (CLL), que consistió en examinar una deleción recurrente localizada en el cromosoma 13q14.3. Se encontró que la región común más pequeña de la deleción codifica para 2 miRNAs: miR-15a y miR-16-1, lo que sugiere su papel como genes supresores de tumores.29 Cuando estos miRNAs se expresan normalmente se unen a la región 3´UTR del RNAm de la proteína anti-apoptótica BCL2, lo que provoca la inhibición de su traducción, y pueden activarse los mecanismos usuales de muerte celular programada. La ausencia de miR-15a y miR-16-1 induce niveles elevados de esta proteína y el bloqueo de la apoptosis.30,31 Otros ejemplos de miRNAs que funcionan como supresores tumorales son las familias de let-732 y miR-3433,34 (tabla).
Los miRNAs también pueden actuar como oncogenes. El ejemplo mejor estudiado es el del cluster miR-17-92. Este incluye 6 miRNAs maduros (miR-17, miR-18a, miR-19a, miR19b-1, miR20a y miR-92-1) que comparten un transcrito primario común generado del loci 13q31.3.35 El cluster está amplificado en varios tipos de linfomas y también en cáncer de pulmón, colon, páncreas y próstata.36-38 Su expresión puede ser regulada directamente por los factores de transcripción c-myc y E2F.39,40 La sobreexpresión de este cluster se asocia con el desarrollo del tumor.38 De igual forma, miR21,41,42 miR15543 y miR272/miR27344 son otros ejemplos de miRNAs que actúan como oncogenes (tabla).
En síntesis, cuando se altera la expresión de los miRNAs se desencadena su ganancia o pérdida de función en las células cancerígenas, por lo que las definiciones de oncogenes y supresores tumorales se han expandido para incluir a los miRNAs, además de los clásicos genes que codifican proteínas.22,45 Otro aspecto de interés es que los patrones de expresión de los miRNAs son tejido-específicos, por lo que un mismo miRNA puede actuar como un oncogen o un supresor tumoral en dependencia del contexto.46
MECANISMOS QUE ALTERAN LA EXPRESIÓN DE miRNAs EN CÁNCER HUMANO
La expresión alterada de los miRNAs es el principal mecanismo que desencadena su ganacia o pérdida de función en las células cancerígenas. La activación de factores de transcripción oncogénicos como myc es otro importante mecanismo que altera la expresión de los miRNAs.47 Otra vía puede ser la ocurrencia de aberraciones cromosómicas, ya que el aumento de la expresión de los miRNAs se ha asociado con amplificación genómica38 y la disminución de su expresión se ha asociado con deleción cromosomal, además de otros mecanismos como las mutaciones puntuales y la metilación aberrante de los promotores.48 Por otra parte, la represión global de la biogénesis de los miRNAs emerge como un mecanismo cáncer-específico, ya que las mutaciones en componentes clave de la maquinaria de procesamiento de los miRNAs, como Drosha, DICER1 y XPO5 promueven la transformación maligna y la carcinogénesis.49
APLICACIONES CLÍNICAS DE LOS miRNAs
Teniendo en cuenta que la expresión alterada de los miRNAs está relacionada con el desarrollo del cáncer y la formación de metástasis, ellos tienen un gran potencial para funcionar como biomarcadores para el estado de la enfermedad y la progresión, así como para el diagnóstico, el pronóstico, la clasificación y la evaluación de factores de riesgo. En este sentido, ellos presentan algunas ventajas como el hecho de que los miRNAs maduros son relativamente estables, el estudio de su expresión no requiere de grandes cantidades de muestra, se pueden medir en biopsias de tejido fresco e incluso se han detectado en tejido fijado en formalina y embebido en parafina.50 Estudios recientes demuestran que también pueden ser medidos en algunos fluidos biológicos como suero/plasma o saliva, lo que ofrece una vía menos invasiva para el pesquisaje.51,52 Los perfiles de expresión de miRNAs han sido utilizados para distinguir muestras tumorales de tejidos normales, para identificar tejido tumoral de origen desconocido o de tumores pobremente diferenciados, así como para distinguir diferentes subtipos de tumores.53 Algunas alteraciones de los miRNAs ocurren en pacientes a etapas tempranas, por lo que pueden ser útiles para la detección precoz del cáncer.54
Desde el punto de vista pronóstico, se ha demostrado su utilidad como indicador del resultado clínico,55 de la tendencia a la recurrencia y la metástasis56 y, adicionalmente, puede ser predictor de la respuesta a un determinado tratamiento.57 Los miRNAs no se han detectado solo en el tejido canceroso, sino también en el tejido circundante, por lo que pueden servir para detectar alteraciones en el microambiente del tumor.58 Se sospecha que el polimorfismo de único nucleótido (SNPs, por sus siglas en inglés) dentro de los genes que codifican miRNAs o sus blancos moleculares, es perjudicial y puede aumentar el riesgo de un individuo a desarrollar enfermedades como el cáncer.59
Se han explorado algunas estrategias con fines terapéuticos para normalizar la expresión de los miRNAs. Una de ellas tiene el objetivo de reducir la expresión de los miRNAs con acción oncogénica. Para ello se han sintetizado oligonucleótidos-modificados anti-miRNAs (OMAs), conocidos como "antagomirs", que son complementarios a los miRNAs endógenos y permiten su inhibición de una manera específica.60 Para su aplicación en la clínica será necesario alcanzar su liberación efectiva en el tejido blanco, aspecto que se encuentra en investigación.61 De igual forma, se ha desarrollado un nuevo tipo de inhibidores de miRNAs llamado "miRNA esponjas", que contienen múltiples sitios para unir a los miRNAs diana y son capaces de inhibirlos con la misma eficacia que los OMAs.62 Otra estrategia consiste en elevar la expresión de miRNAs con función de supresores tumorales. Esto puede lograrse utilizando liposomas, polímeros, nanopartículas o vectores virales, que contengan los miRNAs con expresión reducida y de esta forma restaurar sus niveles normales. Estos novedosos diseños todavía requieren de una evaluación más exhaustiva para que constituyan oportunidades terapéuticas para los pacientes con cáncer.61
PERSPECTIVAS FUTURAS
Las investigaciones sobre miRNAs deben enfocarse en la identificación de firmas moleculares tejido-específicas que regulen las metástasis; explorar los miRNAs que desempeñan un papel importante en la regulación de las células madres cancerígenas; traducir los avances del laboratorio en el desarrollo de nuevos marcadores pronósticos y nuevas estrategias terapéuticas, así como desarrollar nuevas técnicas para la detección de miRNAs.
REFERENCIAS BIBLIOGRÁFICAS
1. Ponting CP, Belgard TG. Transcribed dark matter: meaning or myth? Hum Mol Genet. 2010;19(R2):R162-68.
2. Birney E, Stamatoyannopoulos JA, Dutta A, Guigo R, Gingeras TR, Margulies EH et al. Identification and analysis of functional elements in 1 % of the human genome by the ENCODE pilot project. Nature. 2007;447(7146):799-816.
3. Costa FF. Non-coding RNAs. Meet thy masters. Bioessays. 2010;32(7):599-608.
4. Van Bakel H, Nislow C, Blencowe BJ, Hughes TR. Most "dark matter" transcripts are associated with known genes. PLoS Biol. 010;8(5):e1000371.
5. Khachane AN, Harrison PM. Mining mammalian transcript data for functional long non-coding RNAs. PLoS One. 2010;5(4):e10316.
6. Gibb EA, Brown CJ, Lam WL. The functional role of long non-coding RNA in human carcinomas. Molecular Cancer. 2011;10:38 [citado 20 Dic 2011]. Disponible en: http://www.molecular-cancer.com/content/10/1/38
7. Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions. Nat Rev Genet. 2009;10(3):155-9.
8. Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs. Cell. 2009;136(4):629-41.
9. Esquela-Kerscher A, Snack FJ. Oncomirs-microRNAs with a role in cancer. Nat Rev Cancer. 2006;6(4):259-69.
10. Kim VN, Nam JW. Genomics of microRNA. Trends Genet. 2006;22:165-73.
11. Stanczyk J, Pedrioli DM, Brentano F, Sanchez-Pernaute O, Kolling C, Gay RE, et al. Altered expression of MicroRNA in synovial fibroblasts and synovial tissue in rheumatoid arthritis. Arthritis Rheum. 2008;58:1001-9.
12. Krek A, Grun D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, et al. Combinatorial microRNA target predictions. Nat Genet. 2005;37:495-500.
13. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism and function. Cell. 2004; 116:281-97.
14. Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, et al. MicroRNA genes are transcribed by RNA polymerase II. EMBO J. 2004;23:4051-60.
15. Gu TJ, Yi X, Zhao XW, Zhao Y, Yin JQ. Alu-directed transcriptional regulation of some novel miRNAs. BMC Genomics. 2009;10:563.
16. Cai X, Hagedorn CH, Cullen BR. Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs. RNA. 2004;10:1957-66.
17. Han J, Lee Y, Yeom KH, Kim YK, Jin H, Kim VN. The Drosha-DGCR8 complex in primary microRNA processing. Genes Dev. 2004;18:3016-27.
18. Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK. Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex. Cell. 2006;125:887-901.
19. Tang G. siRNA and miRNA: an insight into RISCs. Trends Biochem Sci. 2005;30:106-14.
20. Budhu A, Ji J, Wang XW. The clinical potential of microRNAs. J Hemathol Oncol. 2010;3:37. [citado 23 Dic 2011]. Disponible en: http://www.jhoonline.org/content/3/1/37
21. Liu J, Zheng M, Tang Y, Liang X, Yang Q. MicroRNAs, an active and versatile group in cancers. Int J Oral Sci. 2011;3:165-75.
22. Garzon R, Fabbri M, Cimmino A, Calin GA, Croce CM. MicroRNA expression and function in cancer. Trends Mol Med. 2006;12:580-7.
23. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-54.
24. Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature. 2000;403:901-6.
25. Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 2000;408:86-9.
26. Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T. Identification of novel genes coding for small expressed RNAs. Science. 2001;294:853-8.
27. Lau NC, Lim LP, Weinstein EG, Bartel DP. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans. Science. 2001;294:858-62.
28. Lee RC, Ambros V. An extensive class of small RNAs in Caenorhabditis elegans. Science. 2001;294:862-4.
29. Cimmino A, Calin GA, Fabbri M, Iorio MV, Ferracin M, Shimizu M, et al. miR-15 and miR-16 induce apoptosis by targeting BCL2. Proc Natl Acad Sci USA. 2005;102:13944-9.
30. Jiang X, Wang X. Cytochrome C-mediated apoptosis. Annu Rev Biochem. 2004;73:87-106.
31. Cory S, Huang DC, Adams JM. The Bcl-2 family: roles in cell survival and oncogenesis. Oncogene. 2003;22:8590-607.
32. Johnson SM, Grosshans H, Shingara J, Byrom M, Jarvis R, Cheng A, et al. RAS is regulated by the let-7 microRNA family. Cell. 2005;120:635-47.
33. Welch C, Chen Y, Stallings RL. MicroRNA-34a functions as a potential tumor suppressor by inducing apoptosis in neuroblastoma cells. Oncogene. 2007;26:5017-22.
34. Tazawa H, Tsuchiya N, Izumiya M, Nakagama H. Tumor-suppressive miR-34a induces senescence-like growth arrest through modulation of the E2F pathway in human colon cancer cells. Proc Natl Acad Sci USA. 2007;104:15472-7.
35. Mendell JT. miRiad roles for the miR-17-92 cluster in development and disease. Cell. 2008;133:217-22.
36. Petrocca F, Visone R, Onelli MR, Shah MH, Nicoloso MS, de Martino I, et al. E2F1-regulated microRNAs impair TGFbeta-dependent cell-cycle arrest and apoptosis in gastric cancer. Cancer Cell. 2008;13:272-86.
37. Hayashita Y, Osada H, Tatematsu Y, Yamada H, Yanagisawa K, Tomida S, et al. A polycistronic microRNA cluster, miR-17-92, is overexpressed in human lung cancers and enhances cell proliferation. Cancer Res. 2005;65:9628-32.
38. He L, Thomson JM, Hemann MT, Hernando-Monge E, Mu D, Goodson S, et al. A microRNA polycistron as a potential human oncogene. Nature. 2005;435:828-33.
39. Woods K, Thomson JM, Hammond SM. Direct regulation of an oncogenic micro-RNA cluster by E2F transcription factors. J Biol Chem. 2007;282:2130-4.
40. Sylvestre Y, De Guire V, Querido E, Mukhopadhyay UK, Bourdeau V, Major F, et al. An E2F/miR-20a autoregulatory feedback loop. J Biol Chem. 2007;282:2135-43.
41. Meng F, Henson R, Wehbe-Janek H, Ghoshal K, Jacob ST, Patel T. MicroRNA-21 regulates expression of the PTEN tumor suppressor gene in human hepatocellular cancer. Gastroenterology. 2007;133:647-58.
42. Frankel LB, Christoffersen NR, Jacobsen A, Lindow M, Krogh A, Lund AH. Programmed Cell Death 4 (PDCD4) Is an Important Functional Target of the MicroRNA miR-21 in Breast Cancer Cells. J Biol Chem. 2008;283:1026-33.
43. Vigorito E, Perks KL, Abreu-Goodger C, Bunting S, Xiang Z, Kohlhaas S, et al. microRNA-155 Regulates the Generation of Immunoglobulin Class-Switched Plasma Cells. Immunity. 2007;27:847-59.
44. Voorhoeve PM, le Sage C, Schrier M, Gillis AJ, Stoop H, Nagel R, et al. A genetic screen implicates miRNA-372 and miRNA-373 as oncogenes in testicular germ cell tumors. Adv Exp Med Biol. 2007;604:17-46.
45. Wu W, Sun M, Zou GM, Chen J. MicroRNA and cancer: Current status and prospective. Int J Cancer. 2007;120:953-60.
46. MacFarlane LA, Murphy PR. MicroRNA: Biogenesis, function and role in cancer. Current Genomics. 2010;7(11):537-61.
47. O'Donnell KA, Wentzel EA, Zeller KI, Dang CV, Mendell JT. c-Myc-regulated microRNAs modulate E2F1 expression. Nature. 2005;435:839-43.
48. Calin GA, Ferracin M, Cimmino A, Di Leva G, Shimizu M, Wojcik SE, et al. A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia. N Engl J Med. 2005;353:1793-801.
49. Melo SA, Esteller M. A precursor microRNA in a cancer cell nucleus. Get me out of here! Cell Cycle. 2011; 6(10):922-5.
50. Hoshida Y, Villanueva A, Kobayashi M, Peix J, Chiang DY, Camargo A, et al. Gene Expression in Fixed Tissues and Outcome in Hepatocellular Carcinoma. N Engl J Med. 2008;19(359):1995-2004.
51. Park NJ, Zhou H, Elashoff D, Henson BS, Kastratovic DA, Abemayor E, et al. Salivary microRNA: discovery, characterization and clinical utility for oral cancer detection. Clin Cancer Res. 2009;15:5473-7.
52. Wang J, Chen J, Chang P, Leblanc A, Li D, Abbruzzesse JL, et al. MicroRNAs in plasma of pancreatic ductal adenocarcinoma patients as novel blood-based biomarkers of disease. Cancer Prev Res. 2009;2:807-13.
53. Lu J, Getz G, Miska EA. MicroRNA expression profiles classify human cancers. Nature. 2005;435:834-8.
54. Yanaihara N, Caplen N, Bowman E, Seike M, Kumamoto K, Yi M, et al. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell. 2006;9:189-98.
55. Chung GE, Yoon JH, Myung SJ, Lee JH, Lee SH, Lee SM, et al. High expression of microRNA-15b predicts a low risk of tumor recurrence following curative resection of hepatocellular carcinoma. Oncol Rep. 2010;23:113-9.
56. Coulouarn C, Factor VM, Andersen JB, Durkin ME, Thorgeirsson SS. Loss of miR-122 expression in liver cancer correlates with suppression of the hepatic phenotype and gain of metastatic properties. Oncogene. 2009;28:3526-36.
57. Weizhu Z, Wenyi Q, Ulus A, Edward RS. Circulating microRNAs in breast cancer and healthy subjects. BMC Research Notes. 2009;2:89.
58. Huang YS, Dai Y, Yu XF, Bao SY, Yin YB, Tang M, et al. Microarray analysis of microRNA expression in hepatocellular carcinoma and non-tumorous tissues without viral hepatitis. J Gastroenterol Hepatol. 2008;23:87-94.
59. Chin LJ, Ratner E, Leng S. A SNP in a let-7 microRNA complementary site in the KRAS 30 untranslated region increases non-small cell lung cancer risk. Cancer Res. 2008;68:8535-40.
60. Soifer HS, Rossi JJ, Saetrom P. MicroRNAs in disease and potential therapeutic applications. Mol Ther. 2007;15:2070-9.
61. Palmero EI, de Campos SGP, Campos M. Mechanisms and role of microRNA deregulation in cancer onset and progression. Genetics Molecular Biology. 2011;(34)3:363-70.
62. Ebert MS, Neilson JR, Sharp PA. MicroRNA sponges: Competitive inhibitors of small RNAs in mammalian cells. Nat Methods. 2007;4:721-6.
Recibido: 26 de enero de 2012.
Aprobado: 9 de agosto de 2012.
MSc. Maydelín Frontela Noda. Instituto Nacional de Oncología y Radiobiología. Calle 29 y D, El Vedado, La Habana, Cuba. efrontela@infomed.sld.cu