Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Salud Animal
versión impresa ISSN 0253-570X
Rev Salud Anim. v.31 n.1 La Habana ene.-abr. 2009
Comunicación corta
DESARROLLO DE CONTROLES POSITIVOS PARA MÉTODOS MOLECULARES DE DETECCIÓN DE VIRUS DE INFLUENZA AVIAR
DEVELOPMENT OF POSITIVE CONTROLS FOR MOLECULAR METHODS OF AVIAN INFLUENZA VIRUS DETECTION
Ana María Acevedo*, Elaine Santana**, Heidy Díaz de Arce*, L.J. Pérez* , A. Caballero**, L. Suárez** y O. Sánchez**
*Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, La Habana, Cuba. Correo electrónico: acevedo@censa.edu.cu; ** Centro de Ingeniería Genética y Biotecnología (CIGB). Ave 31 e/ 158 y 190, Playa. La Habana, Cuba
RESUMEN
La influenza aviar (IA) constituye una gran amenaza para la salud animal y humana por lo que es una prioridad a nivel mundial el fortalecimiento de los sistemas de diagnóstico frente a probables brotes, de ahí la importancia de la detección por métodos moleculares (RT-PCR y Real time RT-PCR) por su elevada rapidez, sensibilidad y especificidad de estos que permiten tomar las medidas de control para evitar la diseminación del agente. En este trabajo se describe la obtención y evaluación de controles positivos de amplificación y cuantificación para RT-PCR y Real time RT-PCR (RRT-PCR) obtenidos a partir del clonaje de dos productos de PCR, uno correspondiente a una región del gen de la hemaglutinina del subtipo H5 y el otro al gen de la matriz (M) para Influenza tipo A. Los cebadores se seleccionaron teniendo en cuenta que los productos de PCR incluyeran las regiones correspondientes de parejas de cebadores de H5 e influenza tipo A reportados tanto por RT-PCR y RRT-PCR de laboratorios de referencia de la OIE/FAO para el diagnóstico de esta enfermedad. De un RNA extraído a partir de la cepa H5N2 (gentilmente donado por el laboratorio de referencia para Influenza aviar de la OIE) se obtuvo el cDNA que se utilizó como molde para la amplificación con los cebadores J3/J1C para H5 y M2/M+25 para Influenza tipo A, recomendados por laboratorios de referencia. Se seleccionó un clón a partir del cual se logró amplificar un fragmento de la talla esperada, que se ha utilizado como control positivo en los ensayos de detección de ácidos nucleicos para el subtipo H5 y para influenza tipo A. Además, el plásmido de H5 se evaluó por RRT-PCR y se obtuvo una curva estándar con un Ct de 15. El control de amplificación obtenido para la detección del subtipo H5 permitirá estandarizar los ensayos y evaluar posibles falsos negativos para la detección rápida de este subtipo en Cuba y en el caso del control positivo de influenza tipo A se podrá usar para cuantificación en ensayos de RRT-PCR.
Palabras clave: Influenza aviar; controles positivos de amplificación y cuantificación; diagnóstico molecular influenza
ABSTRACT
Avian influenza is a great threat for animal and human health, thus strengthening diagnostic systems against probable outbreaks is a priority all over the world. That is why detection is important by the use of molecular methods (RT-PCR and Real Time RT-PCR) due to their high speed, sensitivity and specificity allowing to take control measures in order to avoid the dissemination of the agent. In this work, the obtaining and evaluation of amplification and quantification positive controls for RT-PCR and Real Time RT-PCR (RRT-PCR) have been described. They were obtained from the cloning of two PCR products, one of them corresponding to a region of the subtype H5 hemagglutinin gene and the other to the matrix gene (M) for Influenza type A. The primers were selected keeping in mind that the PCR products included the corresponding regions of H5 and influenza type A primer couples reported by RT-PCR and RRT-PCR of OIE/ FAO reference laboratories for the diagnosis of this disease. From an RNA extracted from H5N2 strain (kindly donated by the OIE reference laboratory for avian influenza), the cDNA used as a mold for the amplification with the primers J3/J1C for H5 subtype and M2 M+25 for Influenza type A, recommended by reference laboratories, was obtained. A clone from which a fragment of the size expected was amplified, was selected as a positive control in the detection assays of nucleic acids for the H5 subtype and influenza A type. Also, the H5 plasmid was evaluated for RRT-PCR and a standard curve with a Ct of 15 was obtained. The amplification control obtained for H5 subtype detection will allow to standardize assays and evaluate possible negative falses for the quick detection of this subtype in Cuba; and in the case of the positive control for influenza type A it will be used for quantification in RRT-PCR assays.
Key words: Avian influenza; amplification and quantification positive controls; molecular diagnostic; influenza
La influenza aviar (IA) es una enfermedad emergente que resurge como un problema mundial para la salud animal y humana. Es causada por virus que son miembros de la familia Orthomyxoviridae, género Influenzavirus A (1). Esta enfermedad afecta muchas especies de aves con una amplia variedad de signos clínicos en dependencia de factores relacionados con el ambiente, el hospedero y la cepa de virus (2).
Los subtipos de IA están establecidos por la presencia de la proteína hemaglutinina de la envoltura viral en una de sus 16 variantes antigénicas (H1-H16). Teniendo en cuenta su patogenicidad en aves, se dividen en dos grupos: los de baja patogenicidad (no notificables) y los altamente patógenos (notificables); estos últimos asociados con los subtipos H5 y H7, los que han sido causa de brotes en países como Vietnam, China y Tailandia (3, 4). En la actualidad, la emergencia del subtipo H5N1 en el sudeste asiático, ha convertido a la enfermedad en el problema zoosanitario más importante a nivel mundial. Su elevada difusión al detectarse en 53 países (5, 6) así como su capacidad de sobrepasar la barrera inter-especies y causar la muerte de seres humanos (7), ha establecido como prioridad a nivel mundial el fortalecimiento de los sistemas de diagnóstico frente a probables brotes.
Para el diagnóstico viral se utilizan los ensayos basados en ácidos nucleicos por la ventaja que brindan con relación a los métodos de cultivo y el aislamiento de virus, los que consumen tiempo y son muy laboriosos (8). La técnica de reacción en cadena de la polimerasa, seguido a la trascripción reversa del ARN (RT-PCR), se ha empleado ampliamente para la detección de IA (9, 10). También se ha usado, por la celeridad que se requiere en el diagnóstico, la tecnología de RRT-PCR, una técnica relativamente nueva, la cual permite detectar productos específicos con gran sensibilidad y especificidad en un tiempo menor de 24 horas, indispensable para la reducción de la propagación de esta enfermedad, ha sido exitosamente aplicada en el monitoreo de muestras de campo de virus influenza y constituye una alternativa confiable al aislamiento viral en embrión de pollo (10, 11).
Para el perfeccionamiento de estas técnicas es preciso contar con controles positivos que garanticen el desarrollo e interpretación de los ensayos. Los controles positivos de amplificación se emplean en la estandarización de los ensayos y permiten evaluar posibles falsos negativos, además tienen un gran valor en el caso de países libres de la enfermedad como el nuestro donde no se cuenta con cepas para la detección del subtipo H5 por ser un virus exótico. Para la detección de Influenza tipo A aunque se cuenta con una cepa de baja patogenicidad de otro subtipo que pudiera ser empleada como control positivo de amplificación, contar con un ADN clonado de un plásmido nos facilita la detección por el número de copias a través de la obtención de transcriptos in vitro a partir de este plásmido con lo cual se puede conocer la concentración de una muestra problema en los ensayos de RRT-PCR.
El objetivo del presente trabajo es obtener y evaluar controles positivos de amplificación del subtipo H5 e Influenza tipo A para lo que nos proponemos clonar una secuencia de ADN que incluya la diana de los cebadores recomendados por los laboratorios de referencia para la detección de ambos tanto por RT-PCR como por RRT-PCR (Tabla 1).
Con el fin de seleccionar la secuencia de ADN a clonar que incluyera las parejas de cebadores seleccionados se realizaron alineamientos múltiples con el empleo del programa DNAman, versión 4.02 (12) con estos cebadores y secuencias del gen de la matriz (IvPR8-34 H1N1 Gen M (segmento 7) y el gen de la hemaglutinina del subtipo H5 (Influenza A virus (A/turkey/England/N28/73 (H5N2) AY500365); disponibles en el Banco Internacional de genes (GenBank), site: http://www.ncbi.nlm.nih.gov/Genbank/.
Además se emplearon los programas Oligo 6.31 (13), el cual analiza la eficiencia de los cebadores, así como las condiciones teóricas de termo perfil. (Las parejas de cebadores se describen en la Tabla 1).
Como resultado del alineamiento se decidió utilizar los cebadores J3/J1C para detección de H5 y M2/M+25 porque incluyen los cebadores del RRT-PCR y RT-PCR convencional.
A partir del RNA total extraído de la cepa H5N2 (gentilmente donado por el Laboratorio de referencia para influenza aviar de la OIE) se sintetizó la cadena de ADN complementaria al RNA con el uso de la enzima reverso transcriptasa del virus de la leucemia Moloney-Murina (M-MLV RT) (Promega, Madison, WI, USA) en un volumen final de reacción de 20 µL. Del ARN extraído 5 µL se incubaron con 1 µL de cebadores random (50 ng/uL) (Promega, Madison, WI, USA) y 4 µl de agua libre de nucleasas (Promega, Madison, WI, USA) a 70oC por 10 min, posteriormente, la mezcla se colocó en hielo durante 5 min para mantener el ácido nucleico desnaturalizado. Luego de la incubación se le añadieron a la mezcla 3.5 µL de agua libre de nucleasa (Promega, Madison, WI, USA), 4µL de la solución tampón de reacción 5X [250 mM Tris-HCl (pH 8.3 a 25oC), 375 mM KCl, 15 mM MgCl2, 50 mM DTT], 1µL de cada dNTP 10mM, 0.5µL de inhibidor de ribonucleasas RNAsin 40U/µL (Promega, Madison, WI, USA) y 0.8 µL de M-MLV RT 200U/µL (Promega, Madison, WI, USA), la mezcla de reacción se incubó a 25oC durante 15 min, seguida de 37oC durante 1 hora y desnaturalización final a 94oC durante 5 min.
El ADNc obtenido se utilizó como molde para amplificar los fragmentos de genes, con los cebadores J3/J1C y M2/M+25 (Tabla 1). La talla estimada de los segmentos amplificados fue de 840 pb para el gen de la hemaglutinina (H5) y 826 pb para el gen de la matriz (M). La reacción se desarrolló empleando el juego de reactivos comerciales de Promega (EE.UU), en un volumen final de 50 mL, usando tampón PCR; MgCl2 (25 mM); dNTPs (10 mM) y Taq ADN Polimerasa (5 U/µL). La amplificación se llevó a cabo en un bloque térmico automatizado MastercyclerÒ (Eppendorf, Alemania) usando un programa de 94 oC/2.5 min, seguido de 35 ciclos de 94 oC/30 seg, 55 oC/30 seg, 72 oC/30 seg y 72 oC/7 min. Los productos de PCR se analizaron mediante electroforesis en gel de agarosa al 2%, en presencia de Bromuro de Etidio (0,8µg/mL), a 100 volts durante 30 min, en tampón TBE Tris-Borato (Tris 50mM, ácido borato 50 mM y EDTA 10 mM, pH 8.4).
Los fragmentos obtenidos de la talla esperada, se purificaron usando el juego de reactivos comerciales de extracción de bandas EZNA (OMEGA BIO-TEK, EE.UU). Las bandas obtenidas se semicuantificaron en gel de agarosa, quedando cada una a concentración aproximada de 100 ng/µL. Estos fragmentos se ligaron por separado con el vector comercial pGEM-T Easy (Promega, EE.UU). El vector y la banda se dejaron ligando por 16h a 16 °C y luego se precipitaron con una solución de acetato de sodio (3M, pH 5.2) en relación 1/10 v y con 2.5 volúmenes de Etanol. Posteriormente el ADN precipitado en un volumen final de 5mL se transformó por electroporación (cubetas de 0.2 mM, Bio-Rad, U.K.) y se le dio un shock eléctrico en un equipo de electroporación (Bio-Rad, U.K.) bajo los siguientes parámetros: 2.5 KV, 25 mF y 200 Ù. en las células de E. coli Top-10 (Invitrogen, EE.UU). Finalmente se sembró en medio rico Luria Bertani sólido suplementado con ampicillina (LBA), isopropil-1-tio-â-D-galactopiranósido (IPTG) y 5-bromo-4-cloro-3-indolil-â-D-galactopiranósido (X-gal): 10 g/L triptona, 5 g/L extracto de levadura, 10 g/L NaCl, 15 g/L de Agar Bacteriológico, 50 mg/mL de ampicillina, 0.5 mM de IPTG y 50 mg/mL de X-gal, seleccionando las colonias blancas recombinantes.
Los plásmidos se digirieron con la enzima de restricción EcoRI (Promega, EEUU), identificando clones recombinantes que rendían las bandas a la talla esperada. La concentración del ADN plasmídico de H5 fue de 5 mg/mL y de 7.7 mg/mL para el de Influenza tipo A.
Con estos productos purificados se realizó la reacción de PCR para comprobar la calidad del material, usando 1 mL de los plásmidos diluidos serialmente hasta 10-7, buscando el límite de detección.
Los cebadores seleccionados permiten la obtención de bandas de las tallas esperadas para cada fragmento, 840 pb para H5 y 826 pb para Influenza tipo A (Figuras 1 y 2).

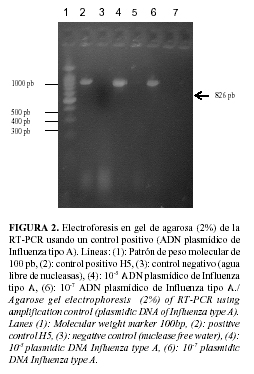
Estos resultados brindan la posibilidad de contar con controles positivos de amplificación (ADN plasmídico) que pueden ser utilizados como controles imprescindibles en los métodos de diagnóstico rápido como la RT-PCR y RRT-PCR, estos tienen la ventaja de que en un solo fragmento clonado se incluyeron todas las dianas que se emplearían en cada ensayo.
Los controles son un elemento esencial en la interpretación de los resultados, sin ellos no se puede desarrollar un ensayo. En el caso de países libres donde no se cuenta con cepas para la detección de virus, tal es el caso del subtipo H5 en nuestro país, estos controles de amplificación permitirán estandarizar los ensayos y evaluar posibles falsos negativos para la detección rápida de este subtipo en Cuba, lo cual permitirá tomar medidas de control más eficaces para enfrentar esta grave amenaza para la salud humana y la seguridad alimentaria en Cuba.
Constituye una gran ventaja contar con el ADN clonado de un plásmido, ya que en los ensayos de Real time PCR no solo garantiza el control de amplificación para la detección de falsos negativos, sino que permite la detección por el número de copias con lo cual se puede conocer la concentración de una muestra problema, pudieran emplearse en futuros estudios para conocer la patogenia de una enfermedad porque la carga viral y la severidad de la misma están relacionadas, por lo que servirían para monitorear la progresión de la enfermedad, además estos clones de ADN de las dianas de amplificación pudieran emplearse para conocer la eficacia de terapias antivirales y en la evaluación de vacunas para conocer la excreción de virus (17).
Estos controles positivos, además, nos ofrecen la ventaja de no tener que manipular muestras infecciosas en las repetidas veces que se elaboren estos ensayos.
REFERENCIAS
1. Manual of Diagnostic Test and Vaccines for Terrestrial Animals. Part 2. Section 2.1. Chapter 2.1.14.Avian Influenza. NB: VERSION ADOPTED MAY 2005. CHAPTER 2.7.12. Avian Influenza [updated 08.07.]. Available from: http://www.oie.int/Eng/Normes/Mmanual/A_00037.htm
2. Chang-Wong L, Senne DA, Suárez DL. Development and application of reference antisera against hemagglutinin subtypes of influenza virus by DNA vaccine chickens. Clin Vaccine Immunol. 2006;13(3):395-402.
3. Fouchier RA, Schneeberger PM, Rozendaal FW. Avian influenza A virus (H7N7) associated with human conjunctivitis and a fatal case of acute respiratory distress syndrome. Proc Natl Acad Sci U S A. 2004;101:1356-1361.
4. CDC/WHO Avian Influenza Response Team Outbreaks of avian influenza A (H5N1) in Asia and interim recommendations for evaluation and reporting of suspected cases-United States. Morb Mortal Wkly Rep. 2004;53:97-99.
5. Webster RG, Peiris M, Chen H. H5N1 outbreaks and enzootic influenza. Emerg Infect Dis. 2006;12(1):3-8.
6. World Health Organization (WHO). (2005): Geographical spread of H5N1 avian influenza in birds. Available from: http://www.who.int/csr/don/2005_08_18/en/index.html
7. World Health Organization (WHO). (2006): Update on Avian Flu Influenza -7 January 2005: Thailand. [update 2006 January 7; cited January 8). Available from: http://w3.whosea.org/
8. Murayama N, Suzuki H, Arakawa M, Nerome K, Mizuta K. Two outbreaks of influenza A (H3N2) in a Japanese nursing home in the winter of 1996-1997, with differing vaccine efficacy. Tohoku. J Exp Med. 1999;188:289-298.
9. Starick E, Werner O. Detection of H7 Avian Influenza virus directly from poultry specimens. Avian Dis. 2003;47(3):1187-1189.
10.Spackman E, Senne DA, Myers TJ, Bulaga LL, Garber LP, Perdue ML, et al. Development of a real-time reverse transcriptase PCR assay for type A influenza virus and the avian H5 and H7 hemagglutinin subtypes. J Clin Microbiol. 2002;40:3256-3260.
11.Chang-Wong L, Súarez DL. Application of real_time RT-PCR for the quantitation and competitive replication study of H5 and H7 subtype avian influenza virus. J Virological Methods. 2004;119:151-158.
12.DNA MAN Version 4.02. Lynnon BioSoft Copyright 1994-1998. BBA, Germany.
13.Oligo Primer Analysis Sofware, version 6.31.Wojciech and Rychlik 1989-2000. Molecular Biology Insights, Inc, 8685 U.S. Highway 24 West. Cascade, CO 80 809, USA.
14.Starick E, Romer-Oberdorfer A, Werrer O. Type and subtype specific RT-PCR assays for avian virus (AIV). J Vet Med. 2000;47:295-301.
15.Slomka MJ, Coward VJ, Banks J, Lo¨ndt BZ, Brown IJ, Voermans J, et al. Identification of Sensitive and Specific Avian Influenza Polymerase Chain Reaction Methods Through Blind Ring Trials Organized in the European Union. Avian Dis. 2007;51:227-234.
16.Ming-Shiuh L, Poa-Chun Ch, Jui-Hung S, Ming-Chu Ch, Happy KS. Identification and subtyping of avian influenza viruses by reverse transcription-PCR. Journal of Virological Methods. 2001;97:13-22.
17.Mackay IM, Arden KE, Nitsche A. Real time PCR in virology. Nucleic Acids Res. 2002;30:1292-1305.
(Recibido 7-11-2007; Aceptado 5-8-2008)














