My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de Salud Animal
Print version ISSN 0253-570X
Rev Salud Anim. vol.31 no.3 La Habana Sept.-Dec. 2009
Comunicación corta
IDENTIFICACIÓN DEL GEN TOX A EN AISLAMIENTOS CUBANOS DE Pasteurella multocida PROCEDENTES DE CERDOS MEDIANTE LA REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
IDENTIFICATION OF TOX A GENE IN CUBAN ISOLATES OF Pasteurella multocida FROM SWINE USING THE POLYMERASE CHAIN REACTION (PCR)
Ivette Espinosa, P. Alfonso, Siomara Martínez
Dpto. Biología Molecular, Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, La Habana, Cuba. Correo electrónico: espinosa@censa.edu.cu
RESUMEN
Pasteurella multocida es parte de la flora comensal del tracto respiratorio superior de cerdos. Usualmente, las cepas de P. multocida del serotipo A se asocian a neumonía en animales de crecimiento y las del serotipo D codifican para una toxina de 145 kDa asociada a la rinitis progresiva. En este trabajo se realizó la caracterización bioquímica y molecular de 30 cepas de P.multocida aisladas a partir de cerdos con clínica respiratoria. Todos los aislamientos presuntivos de P.multocida fueron positivos para los cebadores KMT1SP6 y KMT1T7, amplificando un producto de 460 pb. La presencia del gen tox A solo se detectó en 4 muestras, demostrando la baja frecuencia de cepas toxigénicas y mayor prevalencia de cepas de P.multocida asociadas a procesos neumónicos en las granjas estudiadas. Estos resultados revelan por primera vez en el país la factibilidad del uso de la PCR a partir de una simple colonia para el monitoreo de cepas toxigénicas o no toxigénicas de P.multocida.
Palabras clave: Pasteurella multocida; PCR; rinitis atrófica; cerdos
ABSTRACT
Pasteurella multocida is part of the commensal flora in the upper respiratory tract of pigs. Usually, P. multocida type A strains are associated to pneumonia in grower pigs and the ones from serotype D encode for a 145 kDa toxin associated to progressive rhinitis. Thirty Pasteurella multocida strains isolated in Cuba from swine samples with respiratory signs were identified and characterized. All the isolates showed an amplification product of 460 pb when KMT1SP6 and KMT1T7 primers were used. Only four toxigenic strains of P. multocida were identified by PCR. These data presented revealed low frequency of toxigenic P.multocida and a higher incidence of non toxigenic P.multocida associated to pneumonie.The PCR specifically for identifing P.multocida and the specific primers for tox A gene have been used for the first time in the country from a simple colony showing its feactibility for the monitoring of toxigenic and non toxigenic P.multocida strains.
Key words: Pasteurella multocida; PCR; atrophic rhinitis; pigs
Pasteurella multocida es un patógeno oportunista de amplia distribución mundial tanto en veterinaria como para la salud humana (1,2). Algunos serotipos están relacionados con varios tipos de pasteurelosis, tales como: cólera aviar en especies aviares (3), septicemia hemorrágica en el ganado (4), rinitis atrófica o neumonía en el cerdo (5). Las infecciones humanas por P. multocida usualmente pueden ocurrir a partir de mordeduras de perros y gatos provocando linfangitis, en pacientes inmunocomprometidos, así como meningitis y endocarditis (6,7). Se han identificado 5 serotipos capsulares de P. multocida (A, B, D, E y F), cada uno está generalmente asociado con un hospedero específico, pero no completamente restringido al mismo (8, 9,10).
La rinitis atrófica progresiva (RAP) en el cerdo es de considerable importancia económica. Se caracteriza por la atrofia del tabique nasal y en casos severos puede ocurrir distorsión facial. Esta patología está precedida de una infección primaria o simultánea por un agente como el virus Aujezky, bacterias como Mycoplasma hyopneumoniae y Bordetella bronchiseptica que pueden erosionar los tejidos facilitando la penetración de P.multocida (11,12). Los aislamientos de P. multocida asociados a RAP producen una toxina dermonecrótica de 145 kDa, codificada por el gen tox A ubicado en el cromosoma. Esta toxina induce osteolisis en el tabique. Las cepas que portan este gen, por lo general, corresponden al tipo capsular D. También existen cepas de P. multocida que desempeñan una función fundamental en la pasteurelosis neumónica porcina, por lo general son no toxigénicas y corresponden al tipo capsular A (13,14,15).
Los métodos utilizados en la identificación tradicional de P.multocida se basan en el cultivo, las pruebas bioquímicas y métodos serológicos para la detección de serotipos capsulares, pero estos procedimientos además de consumir tiempo no distinguen la presencia de cepas toxigénicas que intervienen en la RAP de las no toxigénicas relacionadas fundamentalmente con neumonía. La aplicación de la tecnología de la PCR para distinguir entre estas cepas ha sido utilizada demostrando alta sensibilidad y especificidad (16). Por las razones anteriores nos planteamos como objetivo de este trabajo detectar la presencia de cepas de P.multocida portadoras del gen tox A en el país mediante la reacción en cadena de la polimerasa.
Aislamiento e identificación de cepas de P. multocida: se realizó el cultivo a partir de 100 muestras de pulmones y 30 de tonsilas de cerdos procedentes de unidades productoras de las provincias La Habana, Camaguey y Cienfuegos durante el período del 2003 al 2008. Las muestras se sembraron en los siguientes medios: agar sangre Columbia (Oxoid) suplementado con sangre de carnero al 5 %, agar Mc Conkey (Oxoid) y agar Charcoal (Oxoid). Los cultivos se incubaron a 37oC durante 48 horas, las colonias presuntivas del género Pasteurella sp se identificaron según los siguientes criterios: presencia de colonias mucoides medianas, cocobacilos gramnegativos en la tinción de gram y pruebas bioquímicas mediante api 20 NE Biomeriux, France.
Preparación del ADN molde: se utilizaron dos procedimientos: el primero consistió en lisis por calor, para lo cual se resuspendió una colonia en 25 µL de agua estéril, se sometió a ebullición a 1000C durante 5 minutos, se incubó a 0°C durante 10 minutos y se centrifugó a 3000g, se utilizaron 5 µL del sobrenadante en la reacción de PCR (17).
El segundo método consistió en la extracción del ADN a partir de cultivos líquidos de 5 mL en caldo cerebro corazón (oxoid), los cuales se concentraron por centrifugación a 13000g por 5 minutos, se resuspendieron en solución tampón TE (Tris 0.01M pH7.5 y EDTA 0.005M pH 8.0). A continuación se realizó la ruptura con 100mg de lisozima y la lisis alcalina se realizó por la adición de dodecil sulfato de sodio al 20% en solución tampón TE y proteinaza k a 20mg/mL. Se realizaron extracciones con cloroformo y fenol equilibrado con tris pH 7.5. El ADN se precipitó del sobrenadante con etanol (18).
Realización de la PCR:
Las muestras de ADN de las cepas de P.multocida se analizaron por PCR con la pareja de cebadores tox a1 y tox a2 que amplifican un fragmento de 846 pb del gen de la toxina dermonecrótica. Los cebadores presentan las siguientes secuencias:5'-CTTAGATGAGCGACAAGG-3' y 5'-GAATGCCACACCTCTATAG-3' respectivamente (19).
La amplificación se realizó en un volumen final de 25 µL que contenía 10mM de Tris-HCl, pH 8.3; 50 mM de KCl; 3 mM de MgCl2; 0.1mg/mL de BSA, 10 mM de cada dNTP, 20 pmol de cada cebador y 1 ul de amplicen (2u/ µL), CENSA, Cuba. Se adicionaron 2 µL del ADN molde o 5 µL según el procedimiento de extracción de ADN utilizado y se tuvo en cuenta para la adición del volumen de agua. Como control negativo de la reacción se sustituyó el ADN molde por el mismo volumen de agua libre de nucleasas. La mezcla de reacción se sometió a 40 ciclos de: 94°C por 30 segundos, 55°C durante 30 segundos y 72°C por 30 segundos.
Las cepas de P. multocida en las cuales se detectó un producto de amplificación de 846 pb que indican la presencia de un fragmento del gen tox A, se analizaron por PCR con una segunda pareja de cebadores (KMT1SP6 y KMT1T7) presentaron la siguientes secuencias: 5'-GCTGTAAACGAACTCGCCAC-3' y 5'-ATCCGCTATTTACCCAGTGG-3' respectivamente. Estos cebadores amplifican un fragmento de 460 pb según lo reportado en la literatura (20). Para esta reacción de PCR se utilizó el siguiente programa de amplificación: un paso de desnaturalización inicial a 95°C por 4 minutos, seguido de 30 ciclos de: 95°C -1 minuto, 55°C - 1 minuto, 72°C- 1 minuto y una extensión final a 72°C durante 9 minutos.
Todos los cebadores utilizados se sintetizaron en el Centro de Ingeniería Genética y Biotecnología, La Habana, Cuba. Las reacciones de amplificación se realizaron en un termociclador Mastercycler personal eppendorf.
Los productos de las reacciones de amplificación se analizaron por electroforesis en geles de agarosa al 2% en buffer TBE 0.5X y teñido con bromuro de etidio 0,5 µg/mL. La corrida se realizó en tampón TBE 0.5X a un voltaje constante de 100 V/30 min. Para determinar la talla aproximada del producto se incluyó un patrón de peso molecular de 100 pb DNA Ladder (Promega) con un rango de tallas de 100-1500pb. Las bandas se visualizaron en un transiluminador de luz ultravioleta LKB 2011 y se fotografiaron con cámara digital para recoger los resultados.
A partir de las muestras de pulmón de cerdos, se identificaron 30 cepas de P.multocida, las cuales mostraron colonias grises mucosas, confluentes sin presencia de hemólisis en placas de agar sangre. Estas colonias no crecieron en el medio agar Mc Conkey y mostraron la presencia de cocobacilos gramnegativos en la coloración de Gram. Se detectó un único perfil de respuesta bioquímica por el api NE 20 (3-0-0-0-0-0-0) común para todos los aislamientos.
En la Figura 1 se observa la amplificación de un producto de aproximadamente 846 pb cuando se utilizaron los cebadores tox a1 y tox a 2. Solamente se detectaron 4 cepas de P.multocida toxigénicas (P3, P10, P14, P20) aisladas a partir de pulmones de cerdo. La presencia de esta toxina se asocia a RAP fundamentalmente cuando se detecta en cepas aisladas a partir de tonsilas y exudados nasales, sin embargo algunos autores sugieren que cepas de P.multocida toxigénicas pueden ser procedentes de lesiones pulmonares y luego colonizan la cavidad nasal y viceversa (13).
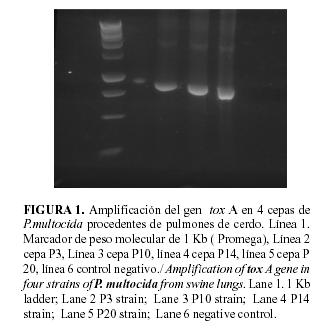
Los resultados de este trabajo revelan una mayor frecuencia de cepas de P. multocida que no portan el gen de la toxina y las cuales están asociadas a procesos neumónicos. Esta situación es similar a la reportada en otras regiones de Suramérica como Brazil y Argentina donde la prevalencia de cepas toxigénicas también es inferior (20, 21, 22). En los últimos años en otras regiones como New Zealand y Canadá se reportan escasos aislamientos de cepas toxigénicas, debido fundamentalmente al incremento en el uso de vacunas para el control de la RAP (23, 24).
La relación entre el tipo capsular de P. multocida, la producción de la toxina dermonecrótica y la neumonía en cerdos aun no está bien definida. La presencia de esta toxina se asocia fundamentalmente al serotipo capsular D y a la RAP, mientras el serotipo A se relaciona a los procesos neumónicos (25). Nagai et al en 1994 (26) y Lichtensteiger et al en 1996 (18) plantean que los cebadores utilizados para la amplificación del gen tox A presentan la misma localización en el cromosoma de aislamientos de ambos tipos capsulares A y D.
Algunos autores no observan consistencia al utilizar técnicas para evaluar la actividad de la toxina in vivo y la amplificación del gen por PCR, por ejemplo Lichtensteiger et al (18) compararon la PCR con el ensayo de letalidad en ratones y concluyeron que la PCR mostró 100% de sensibilidad en la detección del gen en cepas de P. multocida, lo cual no ocurrió en el ensayo de letalidad. Boroski et al. detectaron un total de 24 cepas de P. multocida, de las cuales 9 resultaron positivas para la amplificación por PCR del gen de la toxina; sin embargo, solamente una cepa fue positiva en el ensayo de letalidad en ratones, por lo que sugieren que esto puede deberse a una supresión de la expresión génica de la toxina in vivo después del subcultivo de las cepas, o a la presencia de genes defectivos o no funcionales (25).
La Figura 2 ilustra una banda de aproximadamente 460 pb al utilizar la pareja de cebadores KMT1SP6 y KMT1T7 en las 4 cepas (P3, P10, P14 y P20) que resultaron positivas por PCR en la amplificación con cebadores positivos par el gen tox A. La talla de este amplicón nos confirma la presencia de cepas de P. multocida (subsp. multocida).

Estos oligonucleótidos según lo reportado en la literatura amplifican un fragmento de esta talla en diferentes subespecies de P. multocida (subsp. multocida, subsp. gallicida, and subsp. septica), y en el biotipo 2 de Pasteurella canis (19).
La aplicación del ensayo de PCR específico para subespecies de P. multocida permite la identificación rápida de esta entidad, sin requerir la diferenciación fenotípica que puede tardar hasta dos semanas, antes de informar un biotipo definitivo. Este protocolo de PCR permite, además la detección de P. multocida en cultivos mezclados, lo cual es común cuando las muestras clínicas se obtiene de áreas contaminadas del animal como la garganta y el hocico (26,27).
La aplicación de métodos genotípicos para la identificación de bacterias resulta de gran utilidad y permiten superar limitaciones de los procedimientos fenotípicos tradicionales, al poder detectar la presencia del microorganismo directamente en la muestra clínica o a partir de pequeñas cantidades como una simple colonia, lo cual en cultivos mezclados evita los subcultivos necesarios para la caracterización bioquímica convencional (28).
Los ensayos moleculares específicamente la PCR es de gran interés para el monitoreo y vigilancia de la RAP y la pasteurelosis neumónica mediante la discriminación entre cepas toxigénicas y no toxigénicas (16). Este estudio ha permitido confirmar la presencia de P. multocida por métodos moleculares por primera vez en el país, y demostrar por estos ensayos que las infecciones por P.multocida en las unidades estudiadas se deben fundamentalmente a procesos neumónicos atribuidos a cepas no toxigénicas. Para esclarecer si la expresión de la toxina dermonecrótica es un factor importante también en cepas aisladas de lesiones pulmonares será necesario realizar estudios adicionales en cepas aisladas de esta condición.
REFERENCIAS
1. Hunt ML, Adler B, Townsend KM. The molecular biology of Pasteurella multocida. Vet Microbiol. 2000;72:3-25.
2. Hunt Meredith L, Boucher David J, Boyce John D, Adler Ben. In Vivo-Expressed Genes of Pasteurella multocida Infec Imm. 2001;69(5):3004-3012.
3. Christensen JP, Bisgaard M, Fowl cholera. Rev Sci Tech Off Int Epiz. 2000;19:626-637.
4. Carter G R, De Alwis MCL. Haemorrhagic septicaemia, p. 131-160. In C. Adlam, and J. M. Rutter (ed.), Pasteurella and pasteurellosis. Academic Press Limited, London, England. 1989.
5. Pijoan C. Pneumonic pasteurellosis. In BE. Straw, S. D`Alliare, WL. Mengeling, and DJ. Taylor (ed.), Diseases of swine, 8th ed. Iowa State University Press, Ames, Iowa. 1999. p. 511_520.
6. Kehrenberg C, Salmon SA, Watts JL, Schwarz S. Tetracycline resistance genes in isolates of Pasteurella multocida, Mannheimia haemolytica, Mannheimia glucosida and Mannheimia varigena from bovine and swine respiratory disease: intergenic spread of the tet(H) plasmid pMHT1. J Antimicrob Chemother. 2001;48:631-40.
7. Ewers C, Lübke-Becker A, Wieler LH. Pasteurella: insights into the virulence determinants of a heterogenous bacterium, Berl. Munch. Tierarztl. Wochenschr. 2004;9(10):367-38.
8. Mutters R, Mannheim W, Bisgaard M. Taxonomy of the group. In: C. Adlam and J.M. Rutter, Editors, Pasteurella and Pasteurellosis, Academic Press, London.pp.1989. p.3-34.
9. Townsend KM, Boyce JD, Chung JY, Frost AJ, Adler, B.Genetic organization of Pasteurella multocida cap loci and development of a multiplex capsular PCR typing system. J Clin Microbiol. 2001;39:924-929.
10.Gautam K, Kumar AA, Singh VP, Singh VP, Dutta TK, Shivachandra SB. Specific identification of Pasteurella multocida serogroup A isolates by PCR assay. Res Vete Sci. 2004;76:179-85.
11.De Jong MF. Progressive and nonprogressive atrophic rhinitis. In: Straw BE, Zimmerman JJ, D'Allaire S, et al., editors. Diseases of Swine. 9. Ames, Iowa: Blackwell Pub. 2006 p. 577-602.
12.Sreekumari Rajeev, Nair Rajeev V, Kania Stephen A, David A. Bemis Expression of a truncated Pasteurella multocida toxin antigen in Bordetella bronchiseptica. Vet Microbiol. 2003;94:313-323.
13.Fussing V, Nielsen JP, Bisgaard M, Meyling A. Development of a typing system for epidemiological studies of porcine toxin-producing Pasteurella multocida ssp. multocida in Denmark. Vet Microbiol. 1999;65:61-74.
14.Xibiao Tang, Zhanqin Zhao, Junyong Hu, Bin Wu, Xuwang Cai, Qigai He, Huanchun Chen. Isolation, Antimicrobial Resistance, and Virulence Genes of Pasteurella multocida Strains from Swine in China. J Clin Microbiol. 2009;47(4):951-958.
15.Espinosa Ivette, Martínez Siomara. Pasteurella multocida, Bordetella bronchiseptica y Streptococcus suis en el complejo respiratorio porcino. Rev. Salud Anim.2008;30(3):1-8.
16.Register Karen B, Keith D. DeJong. Analytical verification of a multiplex PCR for identification of Bordetella bronchiseptica and Pasteurella multocida from swine. Vet Microbiol. 2006;117: 201-210.
17.Luther Douglas A, Almeida Raúl A, Oliver Stephen P. Elucidation of the DNA sequence of Streptococcus uberis adhesion molecule gene (sua) and detection of sua in strains of Streptococcus uberis isolated from geographically diverse locations. Vet Microbiol. 2008;128 (3):304-312.
18.Lichtensteiger CA, Steenbergen SM, Lee RM, Polson DD, Vimr ER. Direct PCR analysis for toxigenic Pasteurella multocida. JClin Microbiol. 1996;34(12):3035-3039.
19.Townsend Kirsty M, Frost Alan J, Lee Chiang W, Lee Papadimitriou, John M, Hugh JS, et al. Development of PCR Assays for Species- and Type-Specific Identification of Pasteurella multocida Isolates. JClin Microbiol. 1998;36(4):1096-1100.
20.Cameron RDA, O`Boyle D, Frost A J, Fregan N. An outbreak of haemorrhagic septicaemia associated
with Pasteurella multocida subsp. gallicida in a large pig herd. Aust Vet J. 1996;73:27-29.
21.Moreno M, Baccaro MR, Ferreira APJ, Pestana de Castro AF. Use of Single-Enzyme Amplified Fragment Length Polymorphism for Typing Pasteurella multocida subsp. multocida Isolates from Pigs Clin Microbiol. 2003;41(4):1743-1746.
22.Leotta GA, Vigo GB, Chinen I, Prieto M, Callejo R, Rivas M. Identificación, biotipificación y caracterización de cepas de Pasteurella multocida aisladas en la Argentina. Rev. Argent. Microbiol. 2006;38(3):123-129.
23.Jamaludin R, Blackall PJ, Hansen MF, Humphrey S, Styles M. Phenotypic and genotypic characterisation of Pasteurella multocida isolated from pigs at slaughter in New Zealand. N Z Vet J. 2005; 53:203-207.
24.MacInnes Janet I, Gottschalk Marcelo, Lone Abdul G, Metcalf Devon S, Ojha Shivani, Rosendal Thomas, et al. Prevalence of Actinobacillus pleuropneumoniae, Actinobacillus suis, Haemophilus parasuis, Pasteurella multocida, and Streptococcus suis in representative Ontario swine herds Can J Vet Res. 2008;72(3):242-248.
25.Boroswki SM, Silva SC, Schrank I, Cardoso M. Detection in Pasteurella multocida strains isolated from swine lungs in the state of Rio Grande du sul, Brazil. Arquivos da Faculdad de Veterinária. UFRGS. 2001;29(2):79-85.
26.Nagai S, Someno S, Yagihashi T. Differentiation of toxigenic from nontoxigenic isolates of Pasteurella multocida by PCR. J. Clin. Microbiol. 1994;32:1004-1010.
27.Kamp EM, Bokken GC, Vermeulen TM, de Jong MF, Buys HE, Reek FH, et al. A specific and sensitive PCR assay suitable for large-scale detection of toxigenic Pasteurella multocida in nasal and tonsillar swab specimens of pigs. J Vet Diagn Invest. 1996;8:304-309.
28.Relman DA, Persing DH. Genotypic methods for microbial identification, p. 3-31. In D. H. Persing (ed.), PCR protocols for emerging infectious diseases: a supplement to Diagnostic Molecular Microbiology: Principles and Applications. ASM Press, Washington, D.C. 1996.
(Recibido 9-11-2008; Aceptado 10-2-2009)














