INTRODUCCIÓN
Las Enfermedades de Transmisión Alimentaria (ETA) constituyen una importante causa de morbilidad y mortalidad, así como un significativo impedimento al desarrollo socioeconómico en todo el mundo (1). Los brotes de ETA provocados por leche y productos lácteos contaminados se han descrito a nivel mundial; como los principales agentes causales se reportan Salmonella spp., Campylobacter spp., Escherichia coli, Listeria monocytogenes, Staphylococcus aureus, Yersinia enterocolítica, Mycobacterium bovis, Brucella spp. y Coxiella burnetti (2,3,4).
Salmonella spp. es una de las principales bacterias patógenas que causa daños a los seres humanos y a los animales debido al consumo de alimentos. Se ha reportado como la principal causa de gastroenteritis aguda en varios países (5). En la leche cruda alcanza una prevalencia de hasta 6 % en muestras de tanque (6,7). Las vías de contaminación pueden ser a través del ganado que actúa como reservorio, contaminaciones fecales de los pezones, superficies de la ubre, máquinas de ordeño o contaminación pospauterización en las plantas de producción (3,8).
La intoxicación por Staphylococcus spp. es una de las causas comunes de brotes de origen alimentario a nivel mundial. Desde la perspectiva de la seguridad alimentaria, su importancia se deriva de la capacidad de producir enterotoxinas (9) y su presencia en la leche y derivados lácteos se debe, principalmente, a la contaminación con casos clínicos o subclínicos de mastitis, deficiente higiene durante el proceso de obtención de la leche o proveniente de los trabajadores (10). La prevalencia se reporta hasta 89 % (11); Hill et al. (9) refieren índices de prevalencias de 13-100 % de esta bacteria en leche de tanque.
En Cuba, los reportes sobre calidad bacteriológica en leche cruda son escasos; sin embargo, Martínez et al. (12) indicaron que el 55,8 % de 360 muestras en el eslabón primario de la cadena de producción de leche fueron positivos a la presencia de Salmonella spp. En este mismo estudio, la media de los conteos de Staphylococcus coagulasa positiva fue superior a 1x103ufc/mL en todas las muestras analizadas.
La detección, el aislamiento y la identificación de diferentes tipos de microorganismos en los alimentos se realiza mediante microbiología convencional, por lo general, basado en las normas internacionales (13). Estos protocolos, aunque son baratos y simples, son laboriosos y la obtención de los resultados demora varios días (14,15,16). Recientemente se ha incrementado el uso de métodos moleculares basados en la identificación de ácidos nucleicos, como la PCR, que permite obtener los resultados en corto tiempo, por lo que resulta de vital importancia en la industria alimentaria (15).
Para la detección de Salmonella spp. y S. aureus se han descrito ensayos de PCR, tanto en formato simple como en múltiplex (16,17,18), reconocidos como métodos alternativos para el diagnóstico de estas bacterias en los alimentos (19). El presente trabajo tuvo como objetivo optimizar y evaluar un ensayo de PCR dúplex para la detección de Salmonella spp y S. aureus en leche cruda.
MATERIALES Y MÉTODOS
Cepas: se utilizaron las cepas de referencia Salmonella typhimurium ATCC 14028 y Staphylococcus aureus ATCC 29213. Las cepas se conservaron a -200C en caldo triptona soya con glicerol, según lo descrito por Nakasone et al. (20).
Condiciones de preenriquecimiento: se partió de la suspensión de concentración 1,2x109ufc/mL de cada microorganismo, se tomaron 0,25 µL y se inocularon en 10 mL de caldo nutriente (BioCen; Cuba) enriquecido con 3 % de NaCl (Merck, Alemania) y se incubaron a 35°C durante 12 horas (14).
Extracción de ADN: la extracción de ADN se realizó empleando el protocolo descrito por Ahmadi et al. (21). La calidad y la concentración del ADN se evaluaron en el equipo Colibri Microvolume Nanodrop Spectrophotometer (Titertek-Berthold, Alemania). Las muestras de ADN obtenidas se conservaron a -20ºC hasta su utilización.
Reacción de PCR: se emplearon los cebadores descritos por Kawasaki et al. (22) y Cremonesi et al. (23)(Tabla 1). Las reacciones de PCR se realizaron en un volumen final de 25 μL, que contenía 5 μL GoTaqFlexi Buffer Green 5x; 2 μLMgCl2 (25 mM); 0,5 μLdNTP (10 mM); 0,2 μLTaq Polimerasa GoTaq® Flexi DNA Polymerase5U/μL, (Promega, EE.UU), 1μL (20 mM) de cada cebador; 8,3 μL de agua libre de nucleasas y 5 μL de ADN con el siguiente programa de amplificación: 95ºC por 5 min, seguido de 30 ciclos de 95ºC por 1 min, 56ºC por 1 min, 72ºC por 1 min y una extensión final de 72ºC por 7 min, realizado en el equipo 3-Prime Personal Thermal Cycler (TECHNE; Reino Unido).
Los productos de la reacción de dúplex PCR se aplicaron en geles de agarosa (Sigma-Aldrich, EE.UU.) al 1,5 % en tampón TBE 0,5X y se corrieron a 100 Volts y 50 mA en una cámara de electroforesis horizontal Mupid®-One (Advance, Japón), durante 35 minutos. Los geles se tiñeron con bromuro de etidio (0,5 μg/mL) en tampón de corrida (TBE 0,5X) durante 15 minutos y se visualizaron en un equipo Doc-Print VX5 (Vilber Lourmat, France). En todos los casos se utilizó un marcador de peso molecular de 100 pb (Promega, EE.UU).
TABLA 1 Secuencia de los cebadores utilizados en los ensayos de PCR para la detección de Salmonella spp. y S. aureus. / Sequence of the primers used in the PCR assays for the detection of Salmonella spp. and S. aureus
| Patógeno | Cebador | Secuencia [5´- 3´] | Tm (ºC) | Gen Diana [Amplicon (pb)] | Referencia |
|---|---|---|---|---|---|
|
|
TS-11 | GTC ACG GAA GAA GAG AAA TCC GTA CG | 57,3 |
|
(22) |
| TS-5 | GGG AGT CCA GGT TGA CGG AAA ATT T | 57,7 | |||
| 23S-1200 | AGC TGT GGA TTG TCC TTT GG | 51,5 | ARNr 23S (499) | (23) | |
| 23S-1698 | TCG CTC GCT CAC CTT AGA AT | 51,8 |
Optimización de los parámetros críticos del ensayo de PCR dúplex: se realizaron PCR simples con cada pareja de cebadores en un gradiente de temperatura ± 10ºC (Tabla 2) y se determinó la temperatura óptima de unión de los cebadores (Ta) del PCR dúplex. Para determinar la concentración óptima de los cebadores se evaluó el siguiente rango de concentraciones: 0,1 µM, 0,2 µM, 0,4 µM, 0,5 µM, 0,6 µM, 0,8 µM y 1 µM para cada cebador y las concentraciones de MgCl2, comprendidas entre 0,5 mM y 3,5 mM con incrementos de 0,5 mM.
TABLA 2 Temperaturas evaluadas con cada pareja de cebadores. / Temperatures evaluated with each pair of primers
| Rango Temperatura (± 10ºC) | |
|---|---|
|
|
42°C, 42.2°C, 43.3°C, 45.1°C, 47.4°C, 50°C, 52.8°C, 55.5°C, 58°C, 60.1°C, 61.7°C y 62.6°C |
|
|
47°C, 47.3°C, 48.4°C, 50.2°C, 52.5°C, 55.1°C, 57.8°C, 60.5°C, 63°C, 65.1°C, 66.7°C y 67.5°C |
Especificidad analítica: para evaluar la especificidad analítica se empleó ADN de E. coli O157:H7 ATCC 35150, Streptococcus agalactiae ATCC 27591, Salmonella typhimurium. ATCC 14028 y S. aureus ATCC 29213, Salmonella enteritidis ATCC 13076, Bacillus cereus ATCC 11778 y Listeria monocytogenes ATCC 14119.
Límite de detección (LD): para determinar el LD del ensayo de PCR dúplex se realizaron diluciones seriadas 1/10 (100 ng/μL, 10 ng/μL, 1 ng/μL, 100 pg/μL, 10 pg/μL, 1 pg/μL, 100 fg/μL, 10 fg/μL, 1fg/μL) de ADN extraído de las cepas de referencia Salmonella typhimurium ATCC 14028 y Staphylococcus aureus ATCC 29213.
Detección de Salmonella spp. y S. aureus en muestras de leche cruda: se colectaron 60 muestras de leche cruda según la Norma IDF 50/ISO 707:2012, previamente caracterizadas por la Prueba de California (CMT) y el Tiempo de Reducción del Azul de Metileno (TRAM), de acuerdo a los requisitos microbiológicos de la NC-ISO 448:2006. Las muestras se analizaron por el método de PCR dúplex optimizado. La prevalencia se calculó a través de una aproximación Bayesiana basada en la distribución Beta, beta (s + 1; n-s + 1), donde: s = positivos; n = muestras.
RESULTADOS
El método de extracción permitió obtener la concentración suficiente de ADN y con la calidad requerida para desarrollar el ensayo de PCR. Se amplificaron fragmentos con la talla esperada: 499pb y 375pb para Staphylococcus aureus y Salmonella spp., respectivamente.
En el proceso se obtuvieron productos de PCR de la talla esperada, sin la presencia de amplificaciones inespecíficas. Se seleccionó 0,6 µM como el valor óptimo de concentración de cebadores en 25 µL (Figura 1). La temperatura óptima de alineamiento se seleccionó 60°C y la concentración óptima de MgCl2 fue de 2 mM.

FIGURA 1 Resultados de la determinación de la concentración óptima de cebadores para el ensayo de PCR dúplex en gel de agarosa 1,5 %. M: Marcador de peso molecular 100 pb; 1 - 2: 0,6 µM; 3-4: 0,8 µM; 5-6:1 µM; 7: C- (agua libre de nucleasas). / Results of the determination of the optimal concentration of primers for the 1.5 % agarose gel duplex PCR assay. M: 100 bp molecular weight marker; 1-2: 0.6 µM; 3-4: 0,8 µM; 5-6:1 µM; 7: C- (nuclease-free water)
El ensayo de PCR dúplex optimizado demostró ser específico para el diagnóstico de las bacterias en estudio, ya que solo se obtuvo amplificación frente al ADN de Staphylococcus aureus y Salmonella spp. (Figura 2). La mínima concentración de ADN genómico requerida para la reacción de PCR dúplex fue 1 pg/μL (Figura 3).
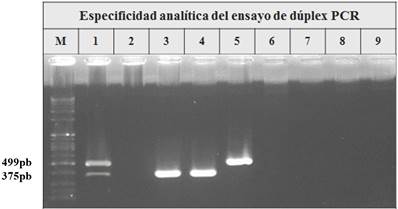
FIGURA 2 Especificidad analítica del ensayo de PCR dúplex. Electroforesis en gel de agarosa 1,5 %. M: Marcador de peso molecular 100 pb1: C+ dúplex PCR S. enteritidis ATCC 13076 y S. aureus ATCC 29213; 2: C- (agua libre de nucleasas); 3: S. enteritidis ATCC 13076; 4: S. typhimurium ATCC 14028; 5: S. aureus ATCC 29213; 6: E. coli O157:H7 ATCC 35150; 7: Streptococcus agalactiae ATCC 2748; 8: L. monocytogenes ATCC 14119; 9: Bacillus cereus ATCC 11778. / Analytical specificity of the duplex PCR assay. Electrophoresis in 1,5 % agarose gel. M: 100 bp1 molecular weight marker: C + duplex PCR S. enteritidis ATCC 13076 and S. aureus ATCC 29213; 2: C- (nuclease-free water); 3: S. enteritidis ATCC 13076; 4: S. typhimurium ATCC 14028; 5: S. aureus ATCC 29213; 6: E.coli O157: H7 ATCC 35150; 7: Streptococcus agalactiae ATCC 2748; 8: L. monocytogenes ATCC 14119; 9: Bacillus cereus ATCC 11778.
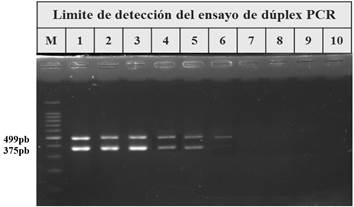
FIGURA 3 Resultados de la determinación del límite de detección del ensayo de dPCR en gel de agarosa 1,5 %. M: Marcador de peso molecular 100 pb; 1:100ng/μL; 2: 10 ng/μL; 3:1 ng/μL; 4: 100 pg/μL; 5:10 pg/μL; 6: 1 pg/μL; 100 fg/μL; 8: 10 fg/μL;9: 1 fg/μL; 10: C- (agua libre de nucleasas)./ Results of the determination of the detection limit of the dPCR assay in 1.5 % agarose gel. M: 100 bp molecular weight marker; 1: 100ng / μL; 2: 10 ng / μL; 3: 1 ng / μL; 4: 100 pg / μL; 5:10 pg / μL; 6: 1 pg / μL; 100 fg / μL; 8: 10 fg / μL; 9: 1 fg / μL; 10: C- (nuclease-free water).
Se obtuvieron 28/60 muestras positivas (46,7 %; IC 95 %: 34 - 59,3 %) a la presencia de Staphylococcus aureus y no se detectó alguna muestra positiva a la presencia de Salmonella spp. (Figura 4).
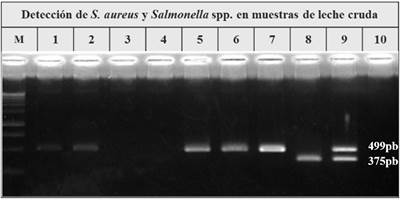
FIGURA 4 Marcador de peso molecular 100 pb (Promega); 1 - 6: Muestras de leche cruda; 7: C+ S. aureus; 8: C+ S. Enteritidis; 9: C+ dúplex PCR; 10:C- (agua libre de nucleasas)./ Hundred bp molecular weight marker (Promega); 1 - 6: Samples of raw milk, 7: C + S. aureus; 8: C + S. Enteritidis; 9: C + duplex PCR; 10: C- (nuclease-free water)
DISCUSIÓN
La PCR facilita y agiliza la detección de los microorganismos patógenos, pero su eficiencia tiende a disminuir si se aplica en muestras de alimentos directamente (24). Para aumentar la eficiencia del ensayo es recomendable realizar un paso de preenriquecimiento, que garantice la revitalización de los microrganismos y permita que las células bacterianas comiencen el proceso de multiplicación normal y disminuyan o eliminen las sustancias inhibidoras de la PCR, provenientes del alimento (25). Este paso posibilita aplicar el método de PCR en diferentes matrices, indistintamente.
El proceso de extracción debe ser eficiente para romper la pared celular sin dañar al ADN (26). Las concentraciones de ADN obtenidas en el presente estudio son similares a las descritas en estudios realizados por Cremonesi et al. (27), quienes utilizaron un protocolo de extracción de ADN basado en la utilización de tiocianato de guanidina, centrifugación y tratamiento térmico.
Es muy importante determinar la concentración óptima de los cebadores para obtener amplificaciones confiables de todos los productos de PCR esperados (28). Dicha concentración de cebadores no debe ser superior a 0,5 µM, ya que en los ensayos de PCR multiplex pueden ocurrir interacciones entre los mismos y afectar la sensibilidad del sistema, si existen diferencias en las concentraciones de los cebadores (29). La mayoría de los oligonucleótidos presentan una mayor actividad a temperaturas entre 56-60°C (29,30). En los ensayos de PCR, con una finalidad diagnóstica, la temperatura de alineamiento se debe establecer en 60°C o mayor, con el objetivo de incrementar la especificidad del ensayo (31).
El ensayo evaluado presentó una alta especificidad, comparable con estudios realizados por Cremonesi et al. (27). Los cebadores basados en el gen ARNr 23S, para el diagnóstico de S. aureus en muestras de leche, mostró una especificidad de 100 %, en cultivos y en leche cruda contaminada. Tsen et al. (32) reportaron la especificidad de los cebadores TS-11/ TS-5 frente a 327 cepas de Salmonella spp. de diferentes serotipos, los cuales no tuvieron reacción positiva frente al ADN de 56 cepas de otras enterobacterias. Por otra parte, Kawasaki (22) reportó una especificidad de 100 % con esta pareja de cebadores para la detección de Salmonella spp.
El ensayo de PCR dúplex tuvo un LD de hasta 1 pg/μL, resultado que coincide con estudios previos descritos por Kim et al. (26), quienes reportan un LD similar de un PCR múltiplex para el diagnóstico de cinco patógenos en leche (Listeria monocytogenes, Escherichia coli O157:H7, Bacillus cereus, Salmonella spp. y Staphylococcus aureus).
Estudios realizados por diferentes autores refieren que la presencia de Salmonella spp. en muestras de leche se puede encontrar en un rango desde “no detectada” hasta 8,9% (2,33,34). Las muestras de leche cruda analizadas por el método de PCR dúplex no fueron positivas a la presencia de esta bacteria patógena Está descrito que la presencia de este microorganismo en muestras de leche cruda depende de diferentes factores, como son la circulación de la bacteria en el ambiente, el tamaño y la salud de los animales y las condiciones de manejo del rebaño (9). Es importante tener en cuenta que la probabilidad de detectar este microorganismo pudo haber estado influenciado, además, por el número de muestras analizadas; por lo que se recomienda profundizar en estos aspectos para estudios posteriores.
La proporción de muestras positivas a S. aureus fue similar a la encontrada por Jørgensen et al. (35) y Hill et al.(9), quienes reportan valores de hasta 60 % de S. aureus en muestras de leche. La presencia de esta bacteria en leche puede significar la afectación de los animales con mastitis bovina o deficiente higiene en la obtención de la leche (36).
La detección de microrganismos patógenos en muestras de alimentos, o en materias primas utilizadas para la fabricación de los mismos, es uno de los principales retos en la industria alimenticia actual (37). La principal contribución de este estudio fue el desarrollo de una metodología para el diagnóstico de Salmonella spp. y S. aureus a partir de precultivos de las muestras analizadas, con lo que se incrementó la sensibilidad de la técnica y se disminuyó el tiempo de diagnóstico de dos microrganismos de vital importancia para la salud pública y veterinaria, con respecto a los métodos convencionales de cultivo. Por otra parte, el empleo de un precultivo permite la posibilidad de analizar diferentes matrices con un mismo ensayo.
El ensayo de PCR dúplex evaluado constituye una herramienta que agiliza y fortalece el diagnóstico de S. aureus y Salmonella spp. en leche, lo cual permite liberar los productos alimenticios en un menor tiempo.














