Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Cultivos Tropicales
versión impresa ISSN 0258-5936
cultrop v.30 n.1 La Habana ene.-mar. 2009
Reclasificación taxonómica de dos cepas de hongos micorrízicos arbusculares
Ms.C. Yakelín Rodríguez, email:yakelin@inca.edu.cu I y Ms.C. Kalyanne FernándezI, Dr. D. van TuinenII
I Investigadoras Agregadas del departamento de Biofertilizantes y Nutrición de las Plantas, Instituto Nacional de Ciencias Agrícolas (INCA), gaveta postal 1, San José de las Lajas, La Habana, Cuba, CP 32700
II dpto. Plante-Microbe-Environnement, UMR INRA, Universidad Bioquímica de Bourgogne, Dijon Cedex, Francia.
ABSTRACT
Knowing the extraordinary biological and economic significance of arbuscular mycorrhizal fungi (AMF) and also due to the satisfactory results reached with the vast field application of two strains, INCAM-2 and INCAM-4, from AMF collection at INCA, this work was aimed at their taxonomic reclassification, taking into account the availability of a single and reliable molecular technique, as well as worldwide experts´ advice on this fungal morphologic characterization. So far, these strains have been known as Glomus clarum-INCAM-2 and Glomus fasciculatum-INCAM-4. The detailed spore observation at the microscope allowed identifying AMF INCAM-2 strain as Glomus mosseae, which was molecularly confirmed. INCAM-4 strain was just morphologically characterized, suggesting to be classified as Glomus hoi-like, since its characteristics do not coincide with any described species; however, they are similar to those of G. hoi considered as a new species, whose identity requires deeper molecular studies.
Key words: fungi, arbuscular mycorrhizae, taxonomy, fungal morphology, PCR
RESUMEN
A partir del conocimiento de la extraordinaria importancia biológica y económica que poseen los hongos micorrízicos arbusculares (HMA) y, en particular, por los resultados satisfactorios alcanzados con la vasta aplicación en campo de dos de las cepas, INCAM-2 e INCAM-4, del cepario del INCA, fue objetivo de este trabajo reclasificarlas taxonómicamente, teniendo en cuenta la disponibilidad de una técnica molecular sencilla y confiable, así como el asesoramiento de expertos a nivel mundial en la caracterización morfológica de estos hongos. Hasta el momento, estas cepas han sido conocidas como Glomus clarum-INCAM-2 y Glomus fasciculatum-INCAM-4. La observación detallada al microscopio de las esporas permitió identificar a la cepa de HMA INCAM-2 como Glomus mosseae, lo cual se confirmó molecularmente. La cepa INCAM-4 fue caracterizada solo morfológicamente, sugiriendo clasificarse como Glomus hoi-like, ya que sus características no coinciden con las de ninguna de las especies descritas, pero se asemejan a las de G. hoi, considerándose una especie nueva, cuya identidad requiere de estudios moleculares más profundos.
Palabras clave: hongos, micorrizas arbusculares, taxonomía, morfología fúngica, PCR
INTRODUCCIÓN
Tradicionalmente los estudios taxonómicos de los hongos micorrízicos arbusculares (HMA) se han basado en la morfología y apariencia de las esporas. Numerosas familias y géneros han sido distinguidos fundamentalmente por la unión de la hifa y el modo de formación de la espora, mientras que la subestructura de las paredes de las esporas jugó un papel importante en la identificación de las especies (1, 2). Sin embargo, el criterio basado solamente en su morfología no refleja la diversidad micorrízica, si consideramos que varios linajes no relacionados dentro de los Glomales forman esporas muy similares del tipo Glomus (3).
Desde principios de la década pasada, los HMA han sido objeto de investigaciones basadas en el ADN a través de la reacción en cadena de la polimerasa (PCR), donde uno de los aspectos a tratar es el concerniente a la filogenia y taxonomía de estos organismos, así como su identificación y monitoreo en el suelo. Un estudio pionero que utilizó ADN ribosomal (4) confirmó, en principio, la clasificación que incluyó un nuevo orden: Glomales y tres familias: Glomaceae, Acaulosporaceae y Gigasporaceae (5). No obstante, las secuencias de ADNr analizadas revelaron una gran diversidad genética dentro de los Glomales, tanto a nivel de familia como de género, siendo esta mucho mayor que la informada previamente, según la taxonomía basada en la morfología de las esporas (6).
A partir de estos resultados, se ha dedicado un gran esfuerzo a desarrollar técnicas moleculares que puedan permitir la identificación de especies de HMA. Muy pocos diseñaron cebadores aleatorios (7); la mayoría escogió pruebas sistemáticas con secuencias ribosomales, por ejemplo, usando cebadores específicos para una porción de la subunidad ribosomal menor 18S (4, 8, 9, 10, 11). Otros han seleccionado la subunidad ribosomal mayor (12, 13) o los espaciadores internos de transcripción: ITS (14).
Algunos propusieron un sistema de identificación usando Polimorfismo de Conformación de Cadena Simple (SSCP) (4, 12), que es menos laborioso que la secuenciación. También se ha utilizado para este fin la electroforesis en geles con gradiente de temperatura (TTGE) en combinación con las técnicas de PCR (15).
Particularmente, se han diseñado cebadores específicos para cuatro HMA (13), incluido Glomus mosseae (Nicol. & Gerd. enmendado por Gerdemann & Trappe) y se realizaron reacciones en cadena de la polimerasa anidada (NESTED PCR), procedimiento muy sensible y simple en términos de manipulación, que puede utilizar tanto esporas como micelio intrarradical en mínimas cantidades como material de partida.
Las cepas de HMA INCAM-2 e INCAM-4, pertenecientes al cepario del Instituto Nacional de Ciencias Agrícolas (INCA), han sido reconocidas hasta la actualidad como Glomus clarum y Glomus fasciculatum, respectivamente. Se consideran entre las más promisorias, dados los resultados satisfactorios que su inoculación han mostrado en distintos cultivos de interés económico y en los diferentes suelos donde se han aplicado. Sobresalen por su importancia el incremento en el rendimiento de los cultivos entre 30 y 45 %, un mejor aprovechamiento de los nutrientes del suelo y los fertilizantes químicos que se requieren en menor dosis (16), ofrecen cierta protección contra patógenos en tomate (17), aumentan la tolerancia al déficit hídrico (18), así como favorecen la aclimatización de vitroplantas de papa (19, 20).
Tomando en consideración lo expuesto anteriormente y conociendo la importancia biológica y económica, así como el amplio uso de estas dos cepas de HMA del cepario del INCA: INCAM-2 e INCAM-4, el objetivo de este trabajo fue revisar la clasificación taxonómica de las cepas, según las nuevas técnicas moleculares disponibles y el asesoramiento de expertos a nivel mundial, que permitió profundizar en la morfología de las esporas.
MATERIALES Y MÉTODOS
Aislamiento y selección de las esporas de HMA. Se estudiaron esporas de dos cepas de HMA pertenecientes al cepario del INCA: INCAM-2 e INCAM-4. Las esporas fueron extraídas de cultivos puros sobre Sorghum bicolor utilizando la técnica de tamizado húmedo y decantado (21). Con ayuda del microscopio estéreo (Stemi2000-C), se seleccionaron las esporas de ambas cepas para ser identificadas hasta el género y, de ser posible, hasta la especie.
Identificación de los HMA
Caracterización morfológica
El criterio para la caracterización morfológica de las esporas se basó principalmente en el color, la forma y el tamaño, estructura de la pared y unión a la hifa de sostenimiento (5, 22, 23). Por tanto, se realizaron preparaciones permanentes para cada tipo de esporas con polivinil-alcohol (PVLG) y polivinil-alcohol más solución de Melzer (1:1, v/v) (PVLG+ Melzer) (22).
Los resultados de esta caracterización se compararon con los del catálogo INVAM (23), de acuerdo con la colección internacional de Glomales. La caracterización de la cepa INCAM-4 fue asesorada por el Dr. Ricardo Herrera (Instituto de Ecología y Sistemática, CITMA, Cuba) y la Dra. Yolande Dalpé (Agriculture and Agri-Food, Ottawa, Canadá), expertos en la temática.
Caracterización molecular
Solo se le realizó a la cepa INCAM-2. Se tomaron de una a tres esporas jóvenes de la cepa lo más similares posibles, teniendo en cuenta su apariencia al microscopio estéreo para realizar la extracción del ADN. Las esporas se lavaron con agua destilada, se maceraron en 40 mL de solución TE (TRIS-HCl 10 mM, pH 8, EDTA 1 mM) y se calentaron a 100°C durante tres minutos en presencia de 10 mL de Chelex-100 al 20 % (BioRad); posteriormente se enfriaron en hielo. La suspensión de ADN fue separada de los fragmentos celulares por centrifugación a 12 000 g por cinco minutos. Posteriormente, se tomó una dilución 1/10 de esta suspensión para realizar la primera reacción de amplificación.
Para esta determinación se llevó a cabo la reacción en cadena de la polimerasa anidada (24) en volúmenes finales de 25 mL que contenían: 10 mM TRIS-HCl pH 9, 50 mM KCl, 1.5 mM MgCl2, 200 nM dNTP, 500 nM de cada cebador, 0.25 U Taq ADN polimerasa (Appligen Oncor, Illkirch) y 5 mL de ADN. El termociclador fue programado de la siguiente forma: ciclo de desnaturalización inicial a 95°C (tres minutos), a continuación 30 ciclos de desnaturalización a 93°C (un minuto), la unión de los cebadores a las cadenas simples de ADN a 60°C (un minuto) y extensión a 72°C (un minuto); el último ciclo fue seguido por una extensión final de 72°C durante cinco minutos. En la primera PCR se usó una combinación del cebador universal específico de eucariota LR1 (5’-GCATATCAATAAGCGGAGGA-3‘) (24) y el cebador específico de Glomeromicota FLR4 (5’-TACGTCAACATCCTTAACGAA-3‘). Para la segunda reacción de amplificación se utilizaron 5 mL del producto de la primera reacción (dilución 1/1000) siguiendo el mismo programa. En este caso, se usó uno de los cebadores anteriores (FLR4) en combinación con un cebador taxón-específico discriminante para Glomus mosseae 5.25 (5’-ATCAACCTTTTGAGCTCG-3') (24).
Los productos de ambas PCR fueron analizados mediante la electroforesis en gel de agarosa 1.4 % usando buffer de corrida TAE (TRIS 40 mM, pH 7.8, ácido acético 20 mM, EDTA 2 mM). Los fragmentos de ADN fueron visualizados tras la tinción con bromuro de etidio (25), siendo comparados con el marcador de peso molecular (Gibco- BRL) y usando como control positivo la cepa Glomus mosseae inscrita en el Banco Europeo de Glomales (BEG) con número de acceso 12. Para el control negativo se sustituyó la suspensión de ADN por agua ultrapura (MiliQ).
Resultados Y DISCUSIóN
Caracterización morfológica. Las esporas de la cepa INCAM-2 presentan las siguientes características morfológicas (Fig. 1): color amarillo-ámbar, forma globosa, tamaño entre 100-260 mm. La unión de la hifa a la espora es en forma de embudo. Presentan septo recurvo cerca de la inserción de la hifa 0-30 mm. Presentan tres capas o paredes, la más externa es mucilaginosa, hialina, frecuentemente separada en las esporas maduras y reacciona positivamente con el reactivo de Melzer (color rojizo). La segunda pared es laminada y más gruesa, y la tercera o interna es más clara.
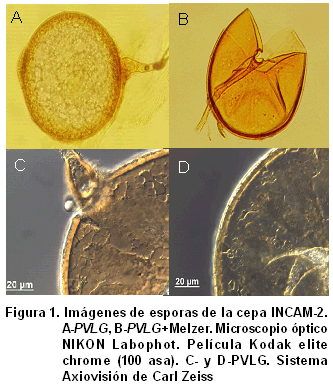
Al realizar una minuciosa comparación de estas características con las que aparecen descritas en el catálogo INVAM (23), hubo una coincidencia con la especie Glomus mosseae. Este resultado fue corroborado por Herrera1.
Al analizar la cepa INCAM-4 (Figura 2), se observaron las siguientes características morfológicas: color hialino, forma globosa a elipsoide, tamaño entre 50-120 mm. La unión de la hifa a la espora es en forma recta o cilíndrica (4.8-5.5 mm de diámetro); en ocasiones se encuentra rodeada por una capa granulada (8.0-10.5 mm) de apariencia similar a la que rodea a la espora. No se observan septos y el poro se encuentra abierto. Presentan de dos a tres capas, siendo la tinción con el reactivo de Melzer negativa. La capa más externa es hialina, 1.5-8.0 mm de grosor, más delgada o casi ausente en esporas jóvenes, la superficie es ornamentada e irregular, rodeada por células de diferentes diámetros que la conforman.
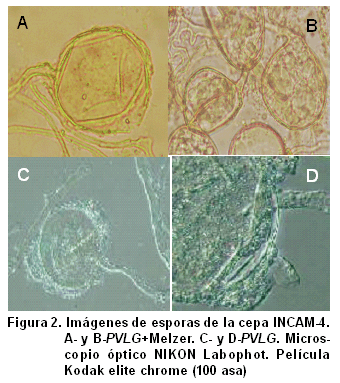
Esta capa externa no puede ser debris de suelo porque: 1) está ausente de esporas jóvenes, 2) es separable de la espora como una concha, 3) siempre es del mismo tipo de material, y 4) un material de textura similar aparece a lo largo de toda la hifa de sostenimiento. La mayoría de las veces la capa interna es única, pero en algunas esporas es doble, hialina, 1.0-1.5 mm y semi-flexible. Las esporas generalmente aparecen en forma de agregados que se ramifican de una hifa común.
La confrontación de las características morfológicas de estas esporas con las que aparecen en el catálogo INVAN (23) no coinciden con las de ninguna de las especies descritas hasta el momento, por lo que se considera una especie nueva2, pero presenta cierta semejanza con las de Glomus hoi, pudiendo sugerirse, por el momento, clasificarla como Glomus hoi-like1.
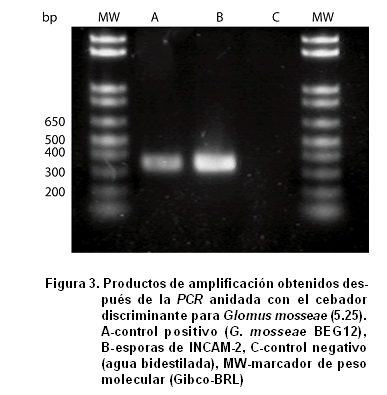
Caracterización molecular. Confirmar mediante una técnica molecular la identificación morfológica de la cepa INCAM-2 era imprescindible, para demostrar con 100 % de confiabilidad su clasificación como especie Glomus mosseae, para lo cual era factible utilizar el protocolo estandarizado de PCR anidada (24) solo para unas pocas especies de HMA de distintos géneros. En la Figura 3 aparece el gel resultante de la segunda PCR, donde se observa la coincidencia en el fragmento de ADN obtenido para las esporas de la cepa INCAM-2 con el de la especie G. mosseae BEG12 usado como control positivo, el cual posee un tamaño de 373 pares de base. Con este resultado, se puede afirmar que la cepa INCAM-2 del cepario del INCA es G. mosseae, corroborando la identificación morfológica realizada.
Para realizar la caracterización molecular de la cepa INCAM-4, se requieren protocolos más complejos que involucran generalmente cebadores menos específicos, la amplificación seguida por la clonación de los productos resultantes de la PCR y luego secuenciación de los fragmentos de ADN obtenidos para compararlos posteriormente con las secuencias registradas en las bases de datos (9, 10, 11), lo cual no ha sido posible hasta ahora. En este caso, no se puede confirmar la caracterización morfológica mediante la técnica descrita (24), debido a que esta cepa no se asemeja lo suficiente a ninguna de las especies descritas y G. hoi, que es la más cercana, no posee cebadores diseñados. También es importante tener en cuenta el tamaño de las esporas, ya que las más grandes, como G. mosseae, son más nobles para utilizar en los protocolos sencillos, mientras que el trabajo con las más pequeñas como G. hoi-like, por ejemplo, se hace más engorroso.
Los trabajos realizados demuestran el valor de los métodos moleculares de identificación, para dilucidar la composición de especies de HMA en una comunidad o incluso dentro de las raíces. No obstante, existen todavía algunos aspectos que dificultan el establecimiento de nuevos marcadores de identidad para los Glomales, como es el hecho de que numerosos microorganismos habiten las esporas de estos hongos (26). El incremento progresivo de las secuencias ribosomales no etiquetadas en las bases de datos también puede causar confusión. Otro problema que ha sido observado es la variación que todas estas secuencias muestran dentro de las esporas y morfoespecies (27). No hay una correlación evidente entre la identidad de la secuencia y de la especie, pues cada espora puede ocultar diferentes tipos de secuencias.
No existe un concepto de especie filogenética para los Glomales y algunos aspectos de su genética continúan siendo un misterio (3).
La revisión taxonómica de las cepas de HMA existentes en un banco o cepario no resulta sorprendente, si se considera que la clasificación internacional de estos hongos es sometida periódicamente a revisiones por expertos, dado el desarrollo de técnicas novedosas más confiables que ayudan a esclarecer las relaciones filogenéticas entre ellos, además de la aparición de nuevas especies no descritas. Por mencionar algunas de estas clasificaciones, está la inicial propuesta por Gerdemann y Trappe (1), que ha sido revisada y replanteada por Morton y Benny (5), Schübler et al. (8) y Morton y Redecker (28), hasta llegar a la actual de Schübler (29). Estas reclasificaciones o revisiones han conducido a erigir nuevos órdenes, familias y géneros con la descripción de nuevas especies de HMA (30, 31, 32, 33).
AGRADECIMIENTOS
Al finado Dr.C. Ricardo Herrera y a la Dra.C. Yolande Dalpé, por su colaboración en la identificación morfológica de las cepas. Al Dr.C. Eduardo Furrasola, por emitir la certificación de la cepa INCAM-4 realizada por el Dr. Herrera en el 2007.
REFERENCIAS
1. Gerdemann, J. W. y Trappe, J. M. Endogonaceae in the Pacific Northwest. Mycologia Memoirs, 1974, vol. 5, p. 1-76.
2. Morton, J. B. Taxonomy of VA mycorrhizal fungi: Classification, nomenclature and identification. Mycotaxon, 1988, vol. 32, p. 267-324.
3. Redecker, D.; Morton, J. B. y Bruns, T. D. Ancestral lineages of arbuscular mycorrhizal fungi (Glomales). Mol. Phylogen. Evol., 2000, vol. 14, p.276-284.
4. Simon, L.; Bousquet, J.; Levesque, R. C. y Lalonde, M. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature, 1993, vol. 363, p. 67-69.
5. Morton, J. B. y Benny, G. L. Revised classification of arbuscular mycorrhizal fungi (Zygomicetes): a new order, Glomales, two new suborders, Glominae and Gigasporinae, and two new families, Acaulosporaceae, with an emendation of Glomaceae. Mycotaxon, 1990, vol. 37, p. 471-491.
6. Redecker, D. Molecular identification and phylogeny of arbuscular mycorrhizal fungi. Plant and Soil, 2002, vol. 244, p. 67-73.
7. Lanfranco, L.; Wyss, P.; Marzachi, C. y Bonfante, P. Generation of RAPD-PCR primers for the identification of isolates of Glomus mosseae, an arbuscular mycorrhizal fungus. Mol. Ecol., 1995, vol. 4, p. 61–68.
8. Schübler, A.; Gehrig, H.; Schwarzott, D. y Walker, C. Analysis of partial Glomales SSU RNA gene sequences: implications for primer design and phylogeny. Mycol. Res., 2001, vol. 105, p. 5–15.
9. DeBellis, T. y Widden, P. Diversity of the small subunit ribosomal RNA gene of the arbuscular mycorrhizal fungi colonizing Clintonia borealis from a mixed-wood boreal forest. FEMS Microbiol. Ecol., 2006, vol. 58, p. 225–235.
10. Lee, J.; Lee, S. y Young, P. W. J. Improved PCR primers for the detection and identification of arbuscular mycorrhizal fungi. FEMS Microbiol. Ecol., 2008, vol. 65, p. 339-349.
11. Öpik, M.; Moora, M.; Zobel, M.; Saks, Ü.; Wheatley, R.; Wright, F. y Daniel, T. High diversity of arbuscular mycorrhizal fungi in a boreal herb-rich coniferous forest. New Phytologist, 2008, vol. 179, p. 867–876.
12. Kjøller, R. y Rosendahl, S. Detection of arbuscular mycorrhizal fungi (Glomales) in roots by nested PCR and SSCP (single stranded conformation polymorphism). Plant Soil, 2000, vol. 226, p. 189–196.
13. Tuinen, D. van; Jacquot, E.; Zhao, B.; Gallotte, A. y Gianinazzi-Pearson, V. Characterization of root colonization profiles by a microcosm community of arbuscular mycorrhizal fungi using 25S rDNA-targeted nested PCR. Mol. Ecol., 1998a, vol. 7, p.879-887.
14. Redecker, D. y Raab, P. Phylogeny of the Glomeromycota (arbuscular mycorrhizal fungi): recent developments and new gene markers. Mycologia, 2006, vol. 98, p. 885-895.
15. Cornejo, P.; Azcón-Aguilar, C.; Barea, J. M. y Ferrol, N. Temporal temperature gradient gel electrophoresis (TTGE) as a tool for the characterization of arbuscular mycorrhizal fungi. FEMS Microbiology Letters, 2004, vol. 241, p. 265-270.
16. Rivera, R. /et al./. El manejo efectivo de la simbiosis micorrízica, una vía hacia la agricultura sostenible. Estudio de caso: El Caribe. La Habana: INCA. 2003.
17. Noval, B. de la; Pérez, E.; Martínez, B.; León, O.; Martínez-Gallardo, N. y Delano-Frier, J. Exogenous systemin has a contrasting effect on disease resistance in mycorrhizal tomato (Solanum lycopersicum) plants infected with necrotrophic or hemibiotrophic pathogens. Mycorrhiza, 2007, vol. 17, no. 5, p. 449-460.
18. Fundora, L. R. La biofertilización micorrízica: Una alternativa ecológica para producir tomate en condiciones de limitados recursos hídricos. (Tesis de Maestría). UNAH. 2006.
19. Rodríguez, Y.; Quiñones, Y. y Hernández, M. M. Efecto de la inoculación con tres cepas de hongos micorrízicos arbusculares sobre la aclimatización de vitroplantas de papa (solanum tuberosum). Cultivos Tropicales, 2006, vol. 27, no. 1, p. 19-24.
20. Rodríguez, Y.; Mena, A.; Marentes, F. L. y Fernández, K. Respuesta de enzimas antioxidantes y crecimiento de vitroplantas de papa micorrizadas in vitro. Cultivos Tropicales, 2008, vol. 29, no. 1, p. 29-35.
21. Gerdemann, J. W. y Nicholson, T. H. Spore of mycorrhizae endogone species extracted from soil by wet sieving and decanting. Trans Br. Mycol. Soc. 1963, vol. 46, p. 235-244.
22. Walker, C. Taxonomic concepts in the Endogonaceae: spore wall characteristics in species descriptions. Mycotaxon, 1983, vol. 18, p. 443-455.
23. INVAM, 2008, http://www.invam.caf.wvu.edu/.
24. Tuinen, D. van; Zhao, B. y Gianinazzi-Pearson, V. PCR in studies of AM fungi: from primers to application. En: Mycorrhizal manual. Berlin Heidelberg: Springer. 1998, p. 387-399.
25. Sambrook, J.; Fritsch, E. F. y Maniatis, T. Molecular cloning: a laboratory manual. Cold Spring Harbour Laboratory Press, New Cork. 1989.
26. Mirabal, L.; Ortega, E. y Diethelm, K. Spores of the mycorrhizal fungus Glomus mosseae host yeasts that solubilize phosphate and acumulate polyphosphates. Mycorrhiza, 2008, vol. 18, p. 197-204.
27. Sanders, I. R.; Alt, M.; Groppe, K.; Boller, T. y Wiemken, A. Identification of ribosomal DNA polymorphisms among and within spores of the Glomales: Application to studies on the genetic diversity of arbuscular mycorrhizal fungal communities. New Phytol., 1995, vol. 130, p. 419–427.
28. Morton, J. B. y Redecker, D. Two new families of Glomales, Archaeosporaceae and Paraglomaceae, with two new genera Archaeospora and Paraglomus, based on concordant molecular and morphological characters. Mycologia, 2001, vol. 93, p. 181-195
29. Schübler A. 2008. http://www.lrz-muenchen.de/~schuessler/amphylo/
30. Oehl, F. y Sieverding, E. Pacispora, a new vesicular-arbuscular mycorrhizal fungal genus in the Glomeromycetes. J. Appl. Bot. Food Qual., 2004, vol. 78, p. 72-82.
31. Walker, C.; Blaszkowski, J.; Schwarzott, D. y Schußler, A. Gerdemannia gen. nov., a genus separated from Glomus, and Gerdemanniaceae fam. nov., a new family in the Glomeromycota. Mycol Res., 2004, vol. 108, p. 707-718.
32. Sieverding, E. y Ole, F. Revision of Entrophospora and description of Kuklospora and Intraspora, two new genera in the arbuscular mycorrhizal Glomeromycetes. J. Appl. Bot. and Food Qual., 2006, vol. 80, p. 69-81.
33. Spain, J. L.; Sieverding, E. y Oehl, F. Appendicispora: a new genus in the arbuscular mycorrhiza-forming Glomeromycetes, with a discussion of the genus Archaeospora. Mycotaxon, 2006, vol. 97, p. 163-182.
Recibido: 3 de noviembre de 2008
Aceptado: 12 de enero de 2009













