Cultivos Tropicales
ISSN 0258-5936
Identificación del gen Ty.3, de resistencia a begomovirus, en accesiones de Solanum lycopersicum L.
Identification of the gene Ty.3, begomovirus resistance in accessions of Solanum lycopersicum L.
Francisco Dueñas Hurtado1, Marta Álvarez Gil1, Carlos Moya López1, Yamila Martínez Zubiaur2
1 Instituto Nacional de Ciencias Agrícolas (INCA).
2 Centro Nacional de Sanidad Agropecuaria (CENSA).
RESUMEN
La enfermedad del rizado amarillo del tomate, cuyo agente causal es el Tomato yellow leaf curl virus (TYLCV) constituye, uno de los principales problemas para el cultivo del tomate en las regiones productoras de Cuba y el mundo. En la actualidad se han descrito varios genes relacionados con la resistencia. Los genes Ty.1, Ty.2 y Ty.3 son los que más se han empleado en los programas de mejoramiento genético de la especie. En Cuba, solo se cuenta con una variedad portadora del gen Ty.1 y otras accesiones portadoras del gen Ty.2. La amplificación del gen Ty.3 se realizó por RCP y se utilizaron los cebadores específicos FLUW.25R y FLUW.25F. En este trabajo se identificó la presencia de la banda relacionada con el patrón resistente del gen Ty.3 en uno de las seis accesiones caracterizadas.
Palabras claves: geminivirus rizado amarillo tomate, resistencia genética.
ABSTRACT
Tomato yellow leaf curl disease is one of the most important problems affecting tomato cultivation in most part of the Cuban growing areas and in other countries. The casual agent is the Tomato yellow leaf curl virus (TYLCV). At the moment several genes related with the resistance have been described. The genes Ty.1, Ty.2 and Ty.3 are those that more they have been used in the plant breeding programs of the species. In Cuba, Vyta is only variety that had Ty.1 gene and other accessions carrying the Ty.2 gene. The amplification of the gene Ty.3 was carried out for PCR and were used the specific primers FLUW.25R and FLUW.25F. In this work the presence of the band related with the resistant pattern of the gene Ty.3 was identified in one that six characterized accessions.
Key word: tomato yellow leaf curl geminivirus, genetic resistance.
INTRODUCCIÓN
La enfermedad del encrespamiento y amarillamiento de la hoja, causada por Tomato yellow leaf curl virus (TYLCV), constituye la mayor afectación del cultivo del tomate (Solanum lycopersicum L.) a nivel internacional (1). En Cuba, las pérdidas por esta virosis representan hasta el 100% de la producción, a nivel nacional, cuando se emplean cultivares susceptibles (2).
La mayoría de los trabajos de mejoramiento del tomate con el objetivo de obtener plantas resistentes a los begomovirus se han realizado en el Viejo Mundo, a pesar de que esta especie vegetal es oriunda de América donde, además, se ha encontrado la mayoría de los geminivirus que azotan a las plantaciones (1).
Hasta la actualidad, con relación a los programas de mejora genética a TYLCV, se ha trabajado en la incorporación de genes de resistencia en tomate, los que han sido detectados en accesiones de especies silvestres, que van desde las más cercanas hasta las más alejadas de S. lycopersicum. L (3). Esto ha permitido que, a nivel internacional se esté utilizando la combinación de varios de estos genes en un mismo material vegetal, en función de lograr una resistencia más duradera (4).
En Cuba solo se cuenta con el cultivar Vyta, portador del gen Ty.1 (5) en las áreas de producción, mientras que a nivel internacional diversos genotipos que portan los genes Ty.2, Ty.3, Ty.4, Ty.5, tcm.1 y tgr.1 de resistencia a begomovirus están siendo utilizados en los programas de mejoramiento para la enfermedad (6, 7, 8, 9, 10, 11).
El Ty.3 es un gen de efecto mayor que está siendo empleado por los programas de mejoramiento genético de la Florida, por el espectro de resistencia que ofrece a begomovirus de genoma monopartito y bipartito que afectan al cultivo (7).
El locus de resistencia a begomovirus, Ty.3, se localizó sobre el brazo largo del cromosoma 6. Este locus se introgresó en líneas avanzadas de tomate, en la Florida, EE.UU., provenientes de un cruzamiento con S. chilense, accesiones LA2779 y LA1932 (7, 12). Los autores identificaron un segmento largo (27 cM), introgresado de LA2779 y uno más corto (6cM), introgresado de LA1932 donde con el estudio de una progenie F2, se pudo evidenciar que el locus Ty.3 explicaba el 65% de la varianza para la resistencia al TYLCV, y el 30% para la resistencia al Virus del moteado del tomate (ToMoV).
En Cuba se desconoce de la presencia de accesiones que porten este gen de resistencia y que sean utilizadas para el mejoramiento genético de la enfermedad. Teniendo en cuenta los antecedentes expuestos, la necesidad de contar con diversas fuentes genéticas para contrarrestar los efectos nocivos del virus y el espectro de resistencia que ofrece Ty.3, se plantea como objetivo identificar la presencia de este gen de resistencia a Begomovirus y determinar el grado de homocigosis en estos cultivares a evaluar, lo cual sería de gran importancia para incorporar a la estrategia de mejoramiento genético del cultivo en Cuba.
MATERIALES Y MÉTODOS
El material vegetal empleado en la investigación (Tabla I), fueron líneas obtenidas a partir de materiales híbridos con niveles de resistencia a TYLCV, provenientes del Volcani Center del Israel (13). Se empleó el cultivar Campbell 28 como testigo susceptible.
Las semillas, seis de cada accesión y del cultivar Campbell 28, fueron sembradas en bandejas de poliestireno que contenían un sustrato compuesto por suelo Ferralítico Rojo Compactado: cachaza: zeolita, en proporción 1:2:1, siendo la capacidad de cada alveolo de 32,5 cm3. Las plántulas se mantuvieron en casa de posturas, de manera protegida hasta alcanzar el estadio fenológico de cuatro hojas verdaderas.
Para detectar la presencia del gen Ty.3, el ADN se extrajo a partir de 5g de cada material vegetal. Las muestras se maceraron en mortero con ayuda de nitrógeno líquido siguiendo la metodología de Dellaporta y col., (15). Se procesaron un total de tres réplicas con tres repeticiones para cada caso y la calidad del ADN se determinó con una lectura a los 260 nm en un espectrofotómetro (Ultroespec Plus Spectrophotometer, Pharmacia LKB).
Para su identificación se emplearon los cebadores, mezclas de reacción y el programa específico para este gen, utilizando un termociclador (MJ Research, Inc.) y los cebadores específicos FLUW.25F y FLUW.25R para su identificación (16).
El programa de amplificación consistió en un paso inicial de desnaturalización a 940C durante 3 min., seguido por 35 ciclos de reacción (30s a 940C de desnaturalización, 1 min. a 530C de anillamiento de los cebadores y 1min. a 720C de extensión), seguido por un paso de extensión final durante 10 min. a 720C. Las reacciones de amplificación se ajustaron a un volumen final de 25ul. Se utilizó 1U de la enzima Taq ADN Polimerasa, con 2,5mM de cada dNTP, 2,5mM de tampón de reacción 10X (Promega), 1,5mM de MgCl2 (Promega), 2,5µM de cada cebador y 15ng de la muestra.
Los productos obtenidos de la reacción en cadena de la polimerasa (RCP), se analizaron en geles de agarosa al 1,5%, utilizándose como patrón de peso molecular un marcador de 100pb (Gibco). Los geles se observaron en un tras iluminador UV.
RESULTADOS Y DISCUSIÓN
En todas las accesiones evaluadas se expresó una homocigosis recesiva (patrón susceptible), para la presencia del Ty.3 (Tabla II); solamente el genotipo STY4, mostró la presencia del gen (patrón resistente), en condiciones de homocigosis dominante el cual se observó en los patrones electroforéticos para todas las plantas evaluadas de este material (Figura 1). La banda que se relacionó con el patrón resistente presentó un tamaño de 640pb. y la del patrón susceptible un tamaño de 480pb. aproximadamente, resultado que coincidió con los obtenidos por Salus y col., (16), en la comprobación de la presencia del locus Ty.3 en líneas e híbridos de tomate obtenidos por el programa de mejoramiento a Begomovirus en Guatemala.
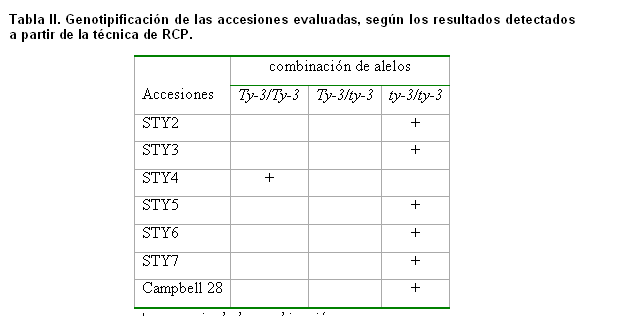

El patrón susceptible del cultivar Campbell 28 comprobó la susceptibilidad de este material a TYLCV y su empleo acertado como control susceptible en los programas de mejoramiento a la enfermedad en el país. Este material no solo carece de este gen sino de otros genes (Ty.1, Ty.2, Ty.4 y Ty.5), descritos y utilizados para la resistencia al TYLCV (5, 6, 7, 8, 9, 10, 17).
A diferencia del fenotipo que mostró el cultivar STY6 en los análisis moleculares y utilizándose una misma combinación de cebadores para la amplificación del gen, se encontró en una línea seleccionada en Marruecos, obtenida a partir de la cruza de STY6 con el híbrido Daniella, que presentó el gen Ty.3 en heterocigosis (16). Este hallazgo pudo estar favorecido por la combinación de esta línea con el híbrido, pues estudios anteriores, a estos resultados enunciaron que la resistencia de STY6 estaba mediada por la acción combinada de varios genes de resistencia introgresados a partir de S. peruvianum (13). Esta es una accesión que ha tenido un buen comportamiento ante los aislados de begomovirus presentes en Guatemala, y que ha sido utilizada por los programas de mejoramiento genético de la hortaliza en este país que es afectado por, al menos, siete begomovirus de genomas bipartitos (18).
Esta accesión, además, ha sido utilizada en los trabajos de resistencia a begomovirus, presentes en Brasil (19), donde en todas las plantas inoculadas no se observó afectaciones provocadas por begomovirus, luego de ser infectadas con mosca blanca (Bemisia tabaci Gen.), lo cual indicó que constituía una fuente de resistencia a emplear ante los aislados circulantes en ese país.
El fenotipo susceptible para el gen Ty.3 en la accesión STY7, se corresponde con los estudios de genética de la resistencia de este material al ser inoculado con el aislado de Israel del TYLCV.
Esta accesión proviene de la especie silvestre S. peruvianum y presentó una resistencia determinada por una dominancia parcial y oligogénica. Los resultados obtenidos sugirieron el modelo de dos genes, uno parcialmente dominante (AA) y otro recesivo (bb), donde cada uno puede contribuir a la resistencia, pero ambos genes controlados de manera dominante y epistática por un tercer gen recesivo (cc) (20). Investigaciones recientes, describieron que la resistencia a TYLCV, en STY7, esta mediada por un QTL mayor el cual llamaron Ty.5, localizado en el cromosoma 4 de la especie. La acción de este QTL es modificada, a su vez, por otros QTL menores situados en los cromosomas 1, 7 y 11 (9), siendo diferentes a los encontrados en los cromosomas 1 y 11 ya descritos por su resistencia al TYLCV (5, 21).
Los resultados de la RCP para la accesión STY5 no mostraron la presencia del gen. Un estudio genético en una línea F6 derivada del híbrido comercial Tyking, manifestó resistencia a un begomovirus bipartito en Brasil Tomato chlorotic mottle virus, la que estuvo condicionada por un solo gen recesivo, del cual se desconoce su localización en el genoma y fue llamado tcm.1 (10), argumento que pudiera explicar la no presencia de Ty.3 en este material. Otras investigaciones destacaron, que una línea portadora del gen tcm.1 mostró un buen comportamiento ante el TYLCV, aislado de Israel (21), lo que amplía el espectro de resistencia de este gen.
Las accesiones STY2 y STY3 mostraron el mismo patrón susceptible que las accesiones STY5, STY6 y STY7. Estos genotipos provienen de otras fuentes de resistencia (Tabla I), y han sido utilizados por los programas de mejoramiento en el Volcani Center desde la validación de sus líneas resistentes a TYLCV.IL (14) hasta la confección de una escala internacional de severidad para la evaluación de los síntomas causados por este begomovirus (13).
El locus Ty.3, también se ha encontrado en otras especies silvestres así como, en líneas avanzadas derivadas de las especies S. peruvianum y S. habrochaites, que presentaron un alto nivel de resistencia a begomovirus bipartitos en Guatemala (7).
El incremento del nivel de resistencia a begomovirus a nivel internacional se podrá obtener por la combinación de diferentes genes de resistencia en un solo cultivar; para lograr el éxito, es necesario combinar los genes de resistencia a distintos virus. Por ello, es importante distinguir los genes en los genotipos resistentes, lo cual puede efectuarse por la caracterización mediante marcadores moleculares (4, 21).
El desarrollo de marcadores relacionados con los genes de resistencia descritos para la especie viral TYLCV y basados en la técnica de RCP, permite la selección asistida de aquellos híbridos, líneas y cultivares portadores de estos genes en los estados de homocigosis adecuados para su utilización por los programas de mejoramiento genético. Esta técnica permite a un costo muy bajo, con un alto nivel de reproducibilidad y baja complejidad el avance por selección asistida de aquellos materiales élites que combinen más de dos genes de resistencia.
La detección del gen Ty.3 en una de las accesiones caracterizadas, propiciará su aprovechamiento en los programas de mejoramiento genético de la hortaliza para begomovirus en el país y en la consolidación del programa de manejo integrado de plagas (MIP), que se desarrolla en Cuba.
REFRENCIAS
1. Barbieri, M., Acciarri, N., Sabatini, E., Sardo, L., Accotto, G. P., Pecchioni, N. Introgression of resistance to two Mediterranean virus species causing tomato yellow leaf curl virus into a valuable traditional tomato variety. 2010. Journal of Plant Pathology. 92 (2), p. 485.493.
2. Gómez, O., Piñón, M., Martínez, Y., Quiñones, M., Fonseca, D., Laterrot, H. Breeding for resistance to begomovirus in tropic.adapted tomato genotypes. Plant Breeding, 2004. 123: 275.279.
3. Czosnek, H., Lapidot. M., Polston. J., Maxwell. D. Tomato Yellow Leaf Curl Virus Disease. Management, molecular biology, breeding for resistance. Netherlands: Springer. 2007, p. 439. ISBN: 978.1.4020.4768.8.
4. Lapidot, M. y J.E. Polston. Resistance to Tomato yellow leaf curl virus in tomato. En: Natural Resistance Mechanism of Plant Viruses. Netherlands: Springer. 2007, p. 503.520.
5. Piñón, M., Gómez, O., Cornide, M. T. RFLP analysis of Cuban tomato breeding lines with resistance to Tomato yellow leaf curl virus. 2005. Acta Hort. 695:273.276.
6. Hanson, P.M., S.K. Green y G. Kuo. Ty.2, a gene on chromosome 11 conditioning geminivirus resistance in tomato. Rept. Tomato Genet Coop Rep. 2006, 56: 17.18.
7. Ji, Y. F., Schuster, D. J., Scott, J. W. Ty.3, a begomovirus resistance locus near the Tomato yellow leaf curl virus resistance locus Ty.1 on chromosome 6 of tomato. Molecular Breeding. 2007, vol.20, no.3. p. 271.284.
8. Ji Y., Scott J.W., Schuster D.J., Maxwell D.P. Molecular mapping of Ty.4, a new Tomato yellow leaf curl virus resistance locus on chromosome 3 of tomato. Journal of the American Society for Horticultural Science. 2009. 134: 281.288.
9. Anbinder, I., Reuveni. M., Azari, R., Paran, I., Nahon, S., Shlomo, H., Chen, L., Lapidot, M., Levin, I. Moleculer dissection of Tomato leaf curl virus resistance in tomato line TY172 derived from Solanum peruvianum. Theor.Appl.Genet. 2009, 119: 519.530.
10. Giordano, L.B., Silva.Lobo, V.L., Santana, F.M., Fonseca, M.E.N., Boiteux, L.S. Inheritance of resistance to the bipartite Tomato chlorotic mottle begomovirus derived from Lycopersicon esculentum cv. ‘Tyking’. Euphytica, 2005, 143: 27–33.
11. Xue.Yu B., M.R. Thomas, M.S. Rasheed, M. Saeed, P. Hanson, P.J. de Barro y M.A. Rezaian. A recessive allele (tgr.1) conditioning tomato resistance to geminivirus infection is associated with impaired viral movement. Phytopathology, 2007, 97 (8):930.937.
12. Ji, Y., y J.W. Scott. Development of breeder friendly markers for begomovirus resistance genes derived from S. chilense. Proc Tomato Breeders Roundtable, Tampa, FL, USA. 2006, roundtable06.ifas.ufl.edu/schedule.htm.
13. Lapidot, M., Ben.Joseph, R., Cohen, L., Machbash, Z., Levy, D. Development of scale for evaluation of Tomato yellow leaf curl virus resistance level in tomato plants. Virology, 2006. Vol. 96, no. 12, p. 1404.1408.
14. Lapidot, M., Friedmann, M., Lachman, O., Yehezkel, A., Nahon, S., Cohen, S., y Pilowsky, M. Comparison of reistance level to Tomato yellow leaf curl virus among comercial cultivars and breeding lines. Plant Disease. 1997. 81: 1425.1428.
15. Dellaporta, S. L., Wood. J., Hicks. J. B. A plant DNA minipreparation: Version II. Plant Molecular Biology Reporter. 1983. v. 1, p. 19.21.
16. Salus, M. S., Martin, C. T., Maxwell, D. P. PCR protocol for detection of introgression at 25 cM (Ty.3 locus). UW.Madison Team. 2006, p. 1.3.
17. Boiteux, L.S., Oliveira, V.R., Silva, C.H., Makishima, N., Inoue.Nagata, A.K, Fonseca, M.E.N., Giordano, L.B. Reaction of tomato hybrids carrying the Ty.1 locus to Brazilian bipartite begomovirus species. Hotricultura Brasileira. 2007, v. 25, no. 1, p. 20.23.
18. Mejía, L., Teni. R. E., Vidavski. F., Czosnek. H., Lapidot. M., Nakhla. M. K., Maxwell. D. P. Evaluation of tomato germplasm and selection of breeding lines for resistance to begomoviruses in Guatemala. Acta Hort. 2005, 695:251.255.
19. Martín. F., da Graça, S., Willians, A., Moreira, D.J., de Brito, G. Sources of resistance in Lycopersicon spp. To bipartite whitefly.transmitted geminivirus from Brazil. Euphytica. 2001. vol 122 no. 1: 45.51.
20. Friedmann, M., Lapidot, M., Cohen, S., y Pilowsky, M. A novel source of resistance to Tomato Yellow Leaf Curl Virus exhibiting a symptomless reaction to viral Infection. Journal of the American Society for Horticultural Science. 1998, 123 (6):1004.1007.
21. García, E. La resistencia como estrategia de control de virus causantes de amarillamiento en tomate. (Tesis de Doctorado). Universidad de Málaga. Facultad de Ciencias. Departamento de Biología Celular, Genética y Fisiología. 2008. 233 p.
Recibido 10/09/2010, aceptado 2/06/2011.














