Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Cultivos Tropicales
versión impresa ISSN 0258-5936
cultrop vol.35 no.4 La Habana oct.-dic. 2014
Polimorfismo bioquímico en cultivares de fresa (Fragaria ananassa Duch.)
Biochemical polymorphism in cultivars of strawberry (Fragaria ananassa Duch.)
Ms.C. Argelys Kessel Dominí,I Ms.C. Regla M. Lara Rodríguez,II Dra.C. María M. Hernández Espinosa,III Dr.C. Orlando Coto ArbeloIV
IAspirante a Investigador del Departamento Genética y Mejoramiento de Plantas, Instituto Nacional de Ciencias Agrícolas (INCA), gaveta postal 1, San José de las Lajas, Mayabeque, Cuba. CP 32 700.
IIEspecialista del Departamento Genética y Mejoramiento de Plantas, Instituto Nacional de Ciencias Agrícolas (INCA), gaveta postal 1, San José de las Lajas, Mayabeque, Cuba. CP 32 700.
IIIInvestigador Titular del Departamento Genética y Mejoramiento de Plantas, Instituto Nacional de Ciencias Agrícolas (INCA), gaveta postal 1, San José de las Lajas, Mayabeque, Cuba. CP 32 700.
IVInvestigador Titular del Instituto de Investigaciones en Fruticultura Tropical (IIFT), La Habana, Cuba.
RESUMEN
La caracterización genética de los cultivares de fresa (Fragaria ananassa Duch.) existentes en Cuba resulta de gran interés ya que permite obtener información básica necesaria para el diseño de estrategias para la conservación de este germoplasma y para futuros programas de mejora del cultivo en el país, con el fin de seleccionar cultivares mejor adaptados a nuestras condiciones edafoclimáticas. El presente trabajo tuvo como objetivo determinar la variabilidad, en siete genotipos de fresa cultivados en diferentes localidades de la isla mediante el empleo de marcadores bioquímicos. El análisis se realizó para los sistemas isoenzimáticos esterasas, anhidrasa carbónica, fosfatasas ácidas y malato deshidrogenasa. El grado de polimorfismo total para los sistemas estudiados fue elevado, de un 89 %, siendo el sistema malato deshidrogenasa el más polimórfico con un 100 %. El mayor nivel de polimorfismo, entre cultivares, se observó en el cultivar ‘Misionaria’ con un 81,2 % y el menor en el cultivar de Villa Clara con un 32,4 %. Se construyó una matriz de presencia-aunsencia de bandas, la que se procesó por el paquete del programa estadístico Past versión 2,14; se empleó el índice de Jaccard y el método UPGMA para obtener el dendograma. Estos resultados permitieron conocer el grado de variabilidad presente en los cultivares de fresa existentes en Cuba, información que podrá ser aprovechada por los programas de mejoramiento genético del cultivo.
Palabras clave: fresa, polimorfismo, cultivar.
ABSTRACT
Genetic characterization strawberry cultivars (Fragaria ananassa Duch.) existing in Cuba is of great interest because it allows to get basic information necessary for the design of conservation strategies of this germplasm for future crop improvement programs in the country, in order to select better adapted cultivars to our soil and climate. The aim of this study is to determine the variability in seven strawberry genotypes grown in different parts of the island by using biochemical markers. The analysis was performed for esterase, carbonic anhydrase, acid phosphatase and malate dehydrogenase isoenzyme systems. The total polymorphism degree of studied systems was high, at 89 %, been the most polymorphic malate dehydrogenase with 100 %. The highest level of polymorphism among cultivars was observed in the cultivar ‘Missionary’ with 81,2 % and the lowest in cultivar of Villa Clara with 32,4 %. Built a matrix absence-presence of bands, which was processed by the Past statistical software package version 2,14, was used Jaccard index and the UPGMA method to obtain the dendrogram. These results allowed to determine the degree of variability present in the strawberry cultivars exist in Cuba, information that can be exploited by breeding programs of the crop.
Key words: strawberry, polymorphism, cultivar.
INTRODUCCIÓN
La producción de fresas en Cuba se inició hace algunos años con el propósito de la comercialización a los polos turísticos de fruta fresca o materia prima y como resultado de ello, se definió un grupo de cultivares adaptables al clima cubano, los cuales fueron, principalmente: ‘Misionaria’, ‘Oso Grande’, ‘Rabunda’, ‘Parker’ y ‘Chandler’ (1). Sin embargo, la propagación de la fruta en las últimas décadas ha estado limitada por la incidencia de altas temperaturas y el ataque de plagas que han conllevado a prácticamente la desaparición del cultivo en la isla (2). Por lo que resulta indispensable realizar acciones encaminadas a obtener cultivares mejor adaptados a las condiciones edafoclimáticas cubanas con el fin de satisfacer el creciente mercado del turismo en fronteras.
En el Instituto de Investigaciones en Fruticultura Tropical (IIFT) existen conservados algunos de los genotipos antes mencionados; además, se han prospectado en diferentes localidades del país, cultivares de fresa que no han sido caracterizados; por lo que el estudio de estos materiales, permitirá obtener un estimado de la variabilidad existente en Cuba, además de brindarnos información básica necesaria para iniciar un programa de mejoramiento en el cultivo (2).
En la fresa, el uso de las isoenzimas ha estado dirigido principalmente a la identificación de cultivares y a la caracterización de la diversidad genética presente entre la fresa norteamericana y europea. El número de sistemas usados en este tipo de trabajos ha sido bastante reducido, debido a que algunos sistemas no presentan polimorfismo y otros presentan inconsistencia en los patrones isoenzimáticos obtenidos, los sistemas más usados han sido glucosa fosfato isomerasa, leucina amino pectidasa y fosfoglucomutasa (3, 4, 5). Además, se han realizado otros estudios con la superóxido dismutasa y las peroxidasas que son enzimas que están implicadas en la protección de plantas contra daños causados por estrés (6, 7).
En Cuba se han empleado otros sistemas enzimáticos como, las esterasas, anhidrasa carbónica y fosfatasas ácidas, que han sido considerados marcadores bioquímicos de gran importancia en la identificación del genoma y se recomiendan en la caracterización del banco de germoplasma de cultivares del género Fragaria (8). Es por ello, que el presente trabajo tuvo como objetivo determinar la variabilidad genética en siete genotipos de fresa cultivados en diferentes localidades de la isla mediante el empleo de marcadores bioquímicos.
MATERIALES Y MÉTODOS
Material vegetal
Se evaluaron siete genotipos de fresa (Fragaria ananassa Duch.), de ellos tres han sido cultivados en diferentes localidades del país y se encuentran conservados en el Banco de Germoplasma del Instituto en Investigaciones en Fruticultura Tropical (IIFT), ellos son: ‘Misionaria’ o fresa criolla, ‘Oso Grande’ y ‘Rabunda’; el resto fueron recolectados en fincas de productores: ‘Parker’, ‘Chandler’, ‘Villa Clara’ y la ‘Gran Piedra’ (Santiago de Cuba). Estos dos últimos cultivares fueron nombrados de acuerdo al lugar donde se colectaron.
Análisis isoenzimático de los cultivares de fresa
Para el análisis bioquímico se obtuvieron los extractos a partir de 0,5 g de hojas sanas en crecimiento activo de plantas jóvenes de cada cultivar, los cuales se encontraban plantados en la finca “Las Papas”, área experimental del Instituto Nacional de Ciencias Agrícolas (INCA). Se empleó como tampón de extracción 0,05 M de Tris-HCl, 0,007 M de ácido cítrico, PEG 1 % y 40 µL de beta-mercaptoetanol. Luego del macerado se centrifugó a 10 000 revoluciones por minuto durante 10 minutos y se conservó a -20 °C hasta su utilización. Las corridas electroforéticas se realizaron en gel de poliacrilamida (PAGE) al 10 %, empleando un equipo de corrida en lámina vertical y un sistema de tampones discontinuos, con tampón de corrida Tris- glicina pH 8,3. El tiempo de corrida estuvo determinado por el desplazamiento de la banda de Kholrauch hasta aproximadamente 6,0 cm del inicio. Se añadió en cada pocillo 20 µL de muestra. La intensidad de corriente empleada fue de 50 mA (9).
Se analizaron un total de cuatro sistemas isoenzimáticos con sus métodos de tinción específicosA (10). Después de teñidos los geles, fueron lavados con agua destilada estéril y se conservaron con una solución de ácido acético al 10 % hasta el momento que se midieron las movilidades electroforéticas (Tabla I) (9).
Análisis estadístico de los datos obtenidos
Los datos obtenidos a partir de los análisis moleculares se tabularon según la presencia (1) o ausencia (0) de bandas. Las similitudes genéticas entre los cultivares de fresa fueron estimadas según el coeficiente de Jaccard, por el paquete de programas estadísticos Past versión 2.14 (11). Los dendogramas se construyeron empleando el método de UPGMA.
RESULTADOS Y DISCUSIÓN
Análisis isoenzimático de los cultivares de fresa
En la Figura 1 se muestra el diagrama electroforético para las isoenzimas fosfatasas ácidas, con un total de once bandas, dos de ellas monomórficas, lo que evidenció un 81,8 % de polimorfismo enzimático, con la presencia de siete zimotipos propios para cada genotipo estudiado (Tabla II). La banda de menor migración anódica solo se observó para los cultivares ‘Misionaria’ y ‘Parker’ y la de mayor migración para ‘Rabunda’, ‘Chandler’ y ‘Misionaria’, la banda de 0,33 unidades sólo estuvo presente en ‘Misionaria’ y ‘Rabunda’ y la de 0,43 en ‘Oso Grande’ y el cultivar recolectado en Villa Clara.
Los altos niveles de polimorfismo enzimático obtenidos en nuestra investigación para este sistema, corresponden con los informados en la literatura en otros cultivos, así como en el estudio de la diversidad de clones de boniato de interés comercialB y en el análisis de especies silvestres de tabacoC.
Vale destacar, que las fosfatasas ácidas juegan un papel importante en la hidrólisis del fósforo orgánico y, por ello, en el ciclo biogeoquímico de este elemento de importancia en la nutrición de las plantas. La actividad de estas enzimas se ha asociado con la capacidad de algunos genotipos de tolerar la baja disponibilidad de fósforo y el estrés producido por agotamiento de sales y agua (12).
Estas enzimas constituyen un ejemplo ideal de marcador ligado a un carácter de resistencia. El locus (Aps-1) que codifica para la fosfatasa ácida-1 ha sido usado como marcador isoenzimático en cultivares de tomate (Solanum lycopercum L.), permitiendo la selección asistida a nemátodos del género Meloidogyne, sin necesidad de infectar el suelo (13).
En el sistema anhidrasa carbónica (Figura 2) se visualizaron once bandas en total y solo la banda de 0,22 unidades resultó común para todos los genotipos, por lo que su polimorfismo fue del 90,9 % (Tabla II). Las bandas de menor y mayor migración anódica (0,04 y 0,90 unidades) no se observaron en el cultivar ‘Parker’. Se detectaron siete patrones de bandas exclusivos para cada cultivar y ‘Misionaria’ fue la que presentó mayor número de isoformas.
Esta gran heterogeneidad electroforética para este sistema, se ha encontrado en otros trabajos de caracterización y detección de la variabilidad genética como en especies silvestres del género Nicotiana (14) y en especies del género Lilium (15).
De acuerdo con estos resultados, podemos plantear que este sistema resultó apropiado para la detección de variabilidad entre genotipos en las especies referidas, aunque se conoce que la activación o inhibición de bandas está influenciada por el estadio de desarrollo fisiológico del material vegetalC.
La enzima anhidrasa carbónica ha sido detectada en un gran número de plantas tanto monocotiledóneas como dicotiledóneas. La mayoría de los informes en plantas se refieren a la función que realizan las anhidrasas carbónicas en los tejidos verdes durante la fotosíntesis, así como con la respuesta al enraizamiento y constituyendo un marcador para este proceso (16).
En el sistema esterasas (Figura 3) se detectaron doce bandas electroforéticas para un 83,3 % de polimorfismo total y se mostraron siete zimotipos. La banda de menor migración anódica se observó en todos los genotipos; sin embargo, la banda de mayor migración solo se presentó para los cultivares ‘Parker’ y ‘Chandler’. Se apreciaron cuatro bandas propias (0,05; 0,35; 0,38 y 0,63 unidades), las cuales permitieron identificar a los cultivares ‘Misionaria’, ‘Villa Clara’, ‘Rabunda’ y ‘Chandler’ del resto.
Es válido destacar que la información obtenida a partir de este sistema isoenzimático, resulta importante, debido a que otros autores emplearon las esterasas para la identificación de cultivares de fresa y no obtuvieron patrones de bandas consistentes (3); sin embargo, en otros estudios realizados en nuestro país (8) con el empleo de este sistema se alcanzaron los niveles más altos de polimorfismo, por lo que pudiera considerarse un marcador bioquímico esencial en la identificación del genoma y poder recomendarlo en la caracterización de cultivares de fresa, en las condiciones evaluadas.
Además, cabe resaltar que en otros cultivos se ha encontrado un marcado polimorfismo para este sistema isoenzimático. En la evaluación de la diversidad de clones de boniato (Ipomoea batatas (L))B se obtuvo patrones de bandas exclusivos y se pudo diferenciar cada clon por separado. Otros autores obtuvieron resultados similares en el estudio de la diversidad de especies silvestres del género Nicotiana (14) y en la caracterización de 23 accesiones de leucaena (Leucaena spp), donde se obtuvo un 92 % de polimorfismo (17).
Estas enzimas abundan mucho en las plantas y tienen una función muy importante en los procesos fotosintéticos. Están constituidas por un grupo complejo de proteínas asociadas con proteínas intracelulares específicas y se caracterizan por la estabilidad en la expresión enzimática, lo que las hace un sistema importante para la realización de estudios fitogenéticos. Su empleo ha sido decisivo en la caracterización de los bancos de germoplasma de diversas especies en el país (15, 18).
El alto polimorfismo expresado para las isoenzimas fosfatasas ácidas, anhidrasa carbónica y esterasas difiere de los informados por otros autores (4, 5) en el análisis de la diversidad genética en especies silvestres y comerciales de Fragaria, donde con el empleo de estos sistemas no detectaron información. Las diferencias encontradas pueden deberse a que las condiciones de cultivo y época en la que se realizó la investigación fueron diferentes y se conoce que las isoenzimas son marcadores de expresión génica, por lo que están influidas por las condiciones ambientales.
Los resultados del análisis de las isoenzimas malato deshidrogenasa se observan en la Figura 4. Se detectó un total de ocho bandas, todas polimórficas, para un 100 % de polimorfismo (Tabla II). En este sistema se formaron seis zimotipos, donde los cultivares ‘Rabunda’ y ‘Oso Grande’ mostraron patrones de bandas idénticos. Se visualizaron dos bandas propias (0,13 y 0,40 unidades) que identificaron a los cultivares ‘Misionaria’ y ‘Parker’. ‘Misionaria’ fue el cultivar de mayor número de bandas y la de menor fue el genotipo recolectado en Villa Clara.
Es de señalar que ha sido muy poco informada la obtención de altos niveles de polimorfismo con el uso de este sistema isoenzimático en diferentes especies vegetales, (17). Sin embargo, otros autores (19), cuando estudiaron los patrones de bandeo isoenzimático de malato deshidrogenasa en cultivares de Tigridia pavónica (L. f.) DC, lograron diferenciar el mayor número de ellos.
Conjuntamente, se ha informado que en condiciones normales, la actividad enzimática de este sistema desaparece en etapas muy tempranas del desarrollo de las plantas, a pesar de que puede inducirse cuando se presentan condiciones de anaerobiosisA. Además, en los cloroplastos hay una forma adicional de MDH (malato deshidrogenasa) dependiente del NADP, que es esencial en la fotosíntesis (20).
Un resumen cuantitativo del resultado de los sistemas isoenzimáticos estudiados, se muestra en la Tabla II. Como se ha mencionado anteriormente, se observa que de un total de 42 bandas analizadas, 37 de ellas fueron polimórficas, para un 89 % de polimorfismo total. Este elevado polimorfismo se puso de manifiesto principalmente en los sistemas malato deshidrogenasa y anhidrasa carbónica. Se obtuvieron un total de 28 zimotipos, de los cuales 27 fueron exclusivos, para un 96,43 %, lo cual está muy relacionado con el grado de variabilidad genética de estos cultivares, para estos sistemas isoenzimáticos. Deben destacarse los sistemas fosfatasa ácida, anhidrasa carbónica y esterasas, los cuales permitieron la caracterización individual de cada genotipo, ratificando que en esta colección de trabajo no se encuentran duplicados y que estos sistemas isoenzimáticos constituyen marcadores potentes en la caracterización genético-bioquímica de cultivares del género Fragaria.
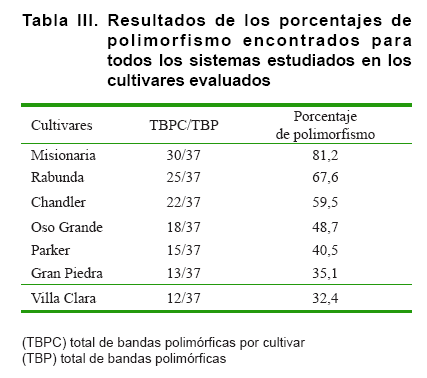
En la Tabla III se refleja el polimorfismo encontrado para cada cultivar cuando se realiza un análisis integral de los resultados aportados a partir de los cuatro sistemas isoenzimáticos empleados. El mayor nivel de polimorfismo lo presentó ‘Misionaria’ con un 81,2 %, mientras que el menor polimorfismo se observó en el genotipo recolectado en ‘Villa Clara’. De ahí que podemos proponer a ‘Misionaria’ como cultivar con mayores y mejores potencialidades para ser utilizado en programas de mejoramiento genético, el mismo presenta variaciones en su expresión de ADN, lo que lo hace adecuado para incrementar la variabilidad en los cultivares comerciales de Fragaria ananassa Duch. y de esta manera disminuir la erosión genética.
Al analizar los resultados del análisis de conglomerado de los siete cultivares del género Fragaria, a partir de los cuatro sistemas isoenzimáticos empleados (Figura 5), se pudo apreciar la formación de un grupo conformado por ‘Misionaria’ y ‘Rabunda’, el resto de los cultivares no se agruparon.
A pesar del desarrollo alcanzado en los estudios genéticos por los marcadores moleculares basados en el ADN, los análisis isoenzimáticos han demostrado mantener su utilidad en la determinación de la diversidad genética en poblaciones, géneros y especies. Las técnicas isoenzimáticas son relativamente económicas, prácticas y factibles de utilizar, aún cuando se analiza un gran número de muestras, ya que mediante la comparación de los patrones de bandas de las isoenzimas analizadas, se puede proporcionar una estimación del grado de relación genética entre los genotipos analizados (9).
Es de señalar que se han realizado otros estudios donde se han empleado otros sistemas como, la superóxido dismutasa con el objetivo de comprobar como aumenta su actividad ante las afectaciones causadas por el hongo Mycosphaerella fragariae, por lo que se considera que esta enzima está asociada a la resistencia a la mancha foliar en la fresa (6). De igual forma se ha determinado el aumento de los niveles de peroxidasas en hojas de fresa ante el estrés provocado por altas temperaturas (7), es por ello que recomendamos emplear estos sistemas en programas de mejoramiento del cultivo para la identificación de cultivares o especies resistentes o suceptibles a enfermedades y estrés.
Los resultados confirman la validez y utilidad de la aplicación de los marcadores bioquímicos utilizados, al permitir la caracterización de los cultivares estudiados y evaluarlos de acuerdo a sus afinidades genéticas. Esta información puede ser utilizada en los programas de mejoramiento genético de la especie comercial Fragaria ananassa Duch. Sin embargo, a pesar de la utilidad demostrada por los marcadores bioquímicos en la caracterización de la variabilidad genética, hay que tener en cuenta las limitaciones de los mismos. Por todo ello es recomendable complementar los estudios bioquímicos con análisis mediante marcadores moleculares de ADN y de esta manera profundizar en el conocimiento de la variabilidad genética existente entre las especies del género Fragaria.
REFERENCIAS
1. Clavijo, R.; Beltrán, A.; Llauger, R.; Rodríguez, A.; Farrés, E.; García, M. U. y Placeres, J. Apuntes sobre el cultivo de la fresa (Fragaria x ananassa Duch.). Citrifrut, julio-diciembre, 2010, vol. 27, no. 2, pp. 67-70. ISSN 1607-5072.
2. Kessel, A. Mejora genética de la fresa (Fragaria ananassa Duch.) a través de métodos biotecnológicos. Cultivos Tropicales, 2012, vol. 33, no. 3, pp. 34-41. ISSN 1819-4087.
3. Bell, J. A. y Simpson, W. The use of isoenzyme polymorphisms as an aid for cultivar identification in strawberry. Euphytica, 1994, vol. 77, pp. 113-117. ISSN 1573-5060.
4. Becerra, V.; Paredes, M. y Romero, A. Biochemical and molecular diversity in Chilean strawberries (Fragaria chiloensis L. Duch.) and its implication for genetic improvement of the species. Agricultura Técnica, 2001, vol. 61, no. 4, pp. 413-428. ISSN 0365-2807.
5. Becerra, V. y Paredes, M. Biochemical and molecular diversity in the chilean strawberry and its implications for plant breeding. Hort. Science, 2005, vol. 40, no. 6, pp. 1642-1643. ISSN 2327-9834.
6. Moghaddam, B. E.; Charles, M. T.; Carisse, O. y Khanizadeh, S. Superoxide dismutase responses of strawberry cultivars to infection by Mycosphaerella fragariae. Journal of Plant Physiology, 2006, vol. 163, no. 2, pp. 147-153. ISSN 0176-1617.
7. Gulen, A. and Eris, A. Effect of heat stress on peroxidase activity and total protein content in strawberry plants. Plant Science, 2004, vol. 166, no. 3, pp. 739-744. ISSN 0168-9452.
8. Kessel, A.; Lara, R. M., Hernández, M. M.; Hernández, Y.; Coto, O. y Téllez, Georvis . Caracterización morfoagronómica y bioquímica de cultivares de fresa existentes en Cuba. En Memorias: Congreso Internacional de Biotecnología de las Plantas IBP. Villa Clara. 2012. ISBN 978-959-16-2045-3.
9. González, C. Detección del polimorfismo genético mediante marcadores bioquímicos en plantas. En: Cornide, M. T. Marcadores Moleculares, nuevos horizontes en la genética y la selección de plantas. (Ed) Felix Varela, La Habana. 2002, pp. 36-66.
10. Wendel, J. F. y Weeden, N. F. Visualization and interpretation of plant isozymes. E. Soltis y P. M. Soltis Editores. “Isozymes in Plant Biology”. Dioscorides Press, 1989, pp. 5-34. ISSN 1516-8913.
11. Hammer, Ø.; Harper, D. A. T. y Ryan, P. D. PAST: Paleontological Statistics Software Package for Education and Data Analysis. Palaeontologia Electronica, 2001, vol. 4, no. 1, 9 pp. ISSN 1094-8074.
12. Castilla, Y.; González, M. E. y Xiqués, S. Micropropagación de plantas de clavel español (Dianthus caryophyllus L.) con el empleo de Biobrás-16: determinación de estabilidad y/o variabilidad genética. En Memorias: Congreso Internacional de Biotecnología de las Plantas IBP, Villa Clara, 2012. ISBN 978-959-16-2045-3.
13. Lara, R. M.; Álvarez, M.; Florido, M.; Dueñas, F.; Plana, D. y Bao, L. Empleo de la selección asistida por marcadores para la resistencia a nemátodos en el cultivo del tomate. En: Memorias I Congreso Cubano de Horticultura, La Habana, 2012. ISBN 978-959-71111-59-7.
14. Valdés, M.; González, C.; Lara, M. R.; Hernández, Y.; Pavón, M. I.; Hernández, R. M.; Cabrera, M. y Torrecilla, G. Diversidad genética de especies silvestres del género Nicotiana I: caracterización mediante marcadores bioquímicos. Protección Vegetal, 2010, vol. 25, no. 2, pp, 88-97. ISSN 2224-4697.
15. Lara, R. M.; Álvarez, M. A.; Florido, M. Dueñas, F. y Bao, L. Caracterización isoenzimática de 10 especies del género Lilium. En Memorias: Congreso Internacional de Biotecnología de las Plantas IBP. Villa Clara. 2012. ISBN 978-959-16-2045-3.
16. Hernández, R. M.; Diosdado, E.; Cabrera, J. C. y Coll, F. Efecto de los biorreguladores del crecimiento en la embriogénesis somática de mandarina Cleopatra (Citrus reshni Hort. ex Tan.). Cultivos Tropicales, 2010, vol. 31, no. 3, pp. 32-38. ISSN 1819-4087.
17. Wencomo, H. B.; Álvarez, A.; Coto, O.; Díaz, M. y Ortiz, R. Caracterización morfoagronómica e isoenzimática de 23 accesiones de Leucaena spp. Pastos y Forrajes, 2011, vol. 34, no. 4, pp. 413-432. ISSN 2078-8452.
18. Castillo, J. G.; Estévez, A.; Salomón, J. L.; Vargas, D.; Hernández, M. M.; Pérez, A.; Borrás, H.; López, Y. y Arencibia, A. D. Determinación de la estabilidad genética de en cuatro especies del banco de germoplasma de papa en Cuba. Cultivos Tropicales, 2010, vol. 31, no. 3, pp. 51-57. ISSN 1819-4087.
19. Arzate, A. M.; Hoyos, A.; Vázquez, L. M. y Gutiérrez, M. de Guadalupe. Caractercterización isoenzimática de nueve variedades de Tigridia pavónica (L.f.) DC. Agrociencia, 2008, vol.42, no. 5, pp. 519-528. ISSN 1405-3195.
20. Wikipedia. Malato Deshidrogenasa (NADP+). Español. Internet, wikipedia, [Consultado 7 de septiembre 2012]. Disponible en: <http://www.es.wikipedia.org/wiki/Malato_deshidrogenasa_(NADP%2B)> .
Recibido: 9 de julio de 2013
Aceptado: 3 de octubre de 2013
Ms.C. Argelys Kessel Domini, Aspirante a Investigador del Departamento Genética y Mejoramiento de Plantas, Instituto Nacional de Ciencias Agrícolas (INCA), gaveta postal 1, San José de las Lajas, Mayabeque, Cuba. CP 32 700. Email: argelys@inca.edu.cu