Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.50 n.2 Ciudad de la Habana Mayo-ago. 1998
Instituto de Medicina Tropical "Pedro Kourí"
Caracterización intratípica de Poliovirus por la técnica de reacción en cadena de la polimerasa
Resumen
Se introdujo la técnica de la reacción en cadena de la polimerasa para la caracterización intratípica de Poliovirus. Se usaron cebadores que sólo promueven la ampliación de las cepas vacunales de Sabin, comprobada por corrida electroforética de los productos de ADN amplificados (Sabin 1-97 pb, Sabin 2-71 pb, Sabin 3-44 pb) y cuya especificidad se verificó satisfactoriamente. Se estudiaron por esta técnica 23 cepas cubanas de Poliovirus aisladas e identificadas en el Laboratorio de Enterovirus del Instituto de Medicina Tropical "Pedro Kourí" de 1993 a 1994, y todas resultaron ser del tipo vacunal. Se observó cómo el Poliovirus vacunal de Sabin puede ser causa de meningoencefalitis viral como complicación neurológica más leve. Este estudio aportó una evidencia más a favor de la no circulación del Poliovirus salvaje en Cuba.
Descriptores DeCS: POLIOVIRUS/aislamiento & purificación; POLIOVIRUS/genética; REACCION EN CADENA POR POLIMERASA/métodos; ARN VIRAL/aislamiento & purificación.
Con la era de la vacunación antipoliomielítica, la incidencia de parálisis fláccida aguda (PFA) declinó de forma drástica en todo el mundo. Hoy en muchos países, el Poliovirus salvaje ha sido remplazado por las cepas vacunales atenuadas, pero sigue siendo causa de parálisis en 1 de cada 1 000 niños nacidos en algunos países subdesarrollados. Las Organizaciones Mundial y Panamericana de la Salud (OMS/OPS) se han propuesto para el año 2000 la total erradicación del Poliovirus salvaje en el mundo,1 y junto a la labor preventiva de la vacunación y la vigilancia epidemiológica, se suma la investigación virológica con el fin de definir la naturaleza salvaje o vacunal del agente causal.
El diagnóstico tradicional del Poliovirus es el aislamiento en cultivos celulares; luego, la identificación y diferenciación intratípica por técnicas serológicas, físico-químicas y de inmunoensayo han sido poco definitorias y engorrosas ya que algunos Poliovirus salvajes poseen propiedades similares a los virus vacunales.2 El avance de la biología molecular ha permitido la introducción de varios métodos diferenciales para la investigación básica y el diagnóstico rápido certero. Éste es el caso de la técnica de reacción en cadena de la polimerasa (RCP) que en la actualidad desempeña una importante función por sus reconocidas sensibilidad, especificidad y simplicidad.3
En 1994, la OMS otorgó a Cuba el Certificado de Erradicación de la Poliomielitis, pero desafortu-nadamente el Poliovirus salvaje está presente en otras áreas del mundo, y las importaciones representan un riesgo de introducir al agente en la comunidad.
En el presente estudio nos propusimos contribuir a la vigilancia virológica del Poliovirus salvaje al introducir la técnica de RCP en la caracterización de los Poliovirus y aplicarla en la diferenciación intratípica de 23 cepas aisladas de 1993 a 1994, para corroborar la no circulación en Cuba de la forma salvaje.
Métodos
Muestras y controles. La muestra comprendió 23 cepas de Poliovirus aisladas de 1993 a 1994 a partir de muestras clínicas de pacientes con PFA y sus contactos asintomáticos, así como de otros casos con enfermedades neurológicas (síndrome de Guillain-Barré-meningoencefalitis viral y neuropatía epidémica) e identificadas por neutralización (Nt) mediante el esquema de Lim-Benyesh-Melnick (LBM),4 así como con monosueros antipoliovirus y sus combinaciones, producidos en el Laboratorio de Enterovirus del Instituto de Medicina Tropical "Pedro Kourí" (Lab EV-IPK) (tabla).
TABLA. Cepas de Poliovirus aisladas sujetas a caracterización intratípica por reacción en cadena de la polimerasa
|
|
|
|
|
| Producto de amplificación | ||
| Cepa | Año | Muestra | Clasificación clínica | Serotipo LBM y monosuero | Sabin 1 97 pb | Sabin 2 71 pb | Sabin 3 44 pb |
| 87 | 1993 | HF | PFA | P1 | + | - | - |
| 610 | 1993 | HF | PNPE | P3 | - | - | + |
| 81 | 1994 | HF | Contacto de PFA | P2 | - | + | - |
| 89 | 1994 | HF | PNPE | P2 | - | + | - |
| 94 | 1994 | HF | PNPE | P2 | - | + | - |
| 103 | 1994 | HF | Sínd. G. Barré | P2 | - | + | - |
| 106 | 1994 | HF | Contacto de PFA | P1 | + | - | - |
| 114 | 1994 | HF | Contacto de PFA | P2 | - | + | - |
| 120 | 1994 | HF | Contacto de PFA | P1 | + | - | - |
| 122 | 1994 | HF | Contacto de PFA | P1 | + | - | - |
| 130 | 1994 | HF | MEV | P1 | + | - | - |
| 168 | 1994 | HF | Contacto de PFA | P3 | - | - | + |
| 195 | 1994 | HF | Contacto de PFA | P1 | + | - | - |
| 199 | 1994 | HF | Contacto de PFA | P2 | - | + | - |
| 200 | 1994 | HF | Contacto de PFA | P3 | - | - | + |
| 211 | 1994 | HF | Contacto de PFA | P1 | + | - | - |
| 214 | 1994 | HF | Contacto de PFA | P3 | - | - | + |
| 233 | 1994 | HF | Contacto de PFA | P3 | - | - | + |
| 237 | 1994 | hf | Contacto de PFA | P2 | - | + | - |
| 241 | 1994 | HF | Contacto de PFA | P3 | - | - | + |
| 255 | 1994 | HF | MEV | P1 | + | - | - |
| 257 | 1994 | HF | MEV | P1 | + | - | - |
| 282 | 1994 | HF | MEV | P1 | + | - | - |
Fuente: Laboratorio de Enterovirus del Instituto de Medicina Tropical "Pedro Kourí" de 1993 a 1994.
HF: Muestra de heces fecales. PFA: Paciente con parálisis fláccida aguda. PNPE: Paciente con polineuropatía epidémica. Sínd. G. Barré: Paciente con diagnóstico de Síndrome de Guillain-Barré. MEV: Paciente con meningoencefalitis viral. Serotipo LBM y monosueros: Serotipo viral según esquema de Lim-Benyesh-Melnick, así como monosueros antipoliovirus y sus combinaciones
Se tomaron como controles positivos: 1) cepas de referencia de Poliovirus vacunales Sabin tipos 1, 2 y 3 recibidas del National Institute for Biological Standard and Control, de Inglaterra, 2) 1 cepa de referencia de Poliovirus salvaje tipo 1: cepa PV-1/Mahoney/USA 42, cuya zona a amplificar es idéntica a la homóloga vacunal, donada gentilmente por el Instituto de Poliomielitis y Virus de Encefalitis de Moscú. Los controles negativos fueron: 1) 1 cepa de Coxsackievirus B-4(#488 de 1994) aislada en el Lab EV-IPK a partir de las heces fecales de un caso de PFA e identificada por Nt mediante el esquema de LBM, 2) 2 cepas de referencia de Polivirus tipo 2 salvajes: cepa PV-2/MEF-1 Moscú y cepa PV-2/MEF-1 Brasil, donadas por el Instituto de Poliomielitis y Virus de Encefalitis de Moscú y el Laboratorio de Enterovirus de FIOCRUZ, Brasil, de forma respectiva, 3) cultivo de células de línea de riñón de mono verde africano Cercopithecus aetiops adulto normal (Vero), no infectadas.
Oligonucleótidos sintéticos. Se usaron 3 pares de oligonucleótidos, diseñados por Yang y otros en 19913 y sintetizados en el Centro de Ingeniería Genética y Biotecnología de Cuba; éstos sólo promueven la amplificación de cepas Sabin y sus progenitores, se excluye la amplificación cruzada con genotipos de Poliovirus salvajes. El producto de la RCP tiene una talla de 97, 71 y 44 pb para los Poliovirus Sabin 1, 2 y 3, respectivamente. A continuación presentamos sus secuencias nucleotídicas:
| Código | Posición | Secuencia |
| Sabin 1/RCP-1 | (2584-2601) | 5TCCACTGGCTTCAGTGTT 3 |
| Sabin 1/RCP-2 | (2505-2523) | 5AGGTCAGATGCTTGAAAGC 3 |
| Sabin 2/RCP-1 | (2580-2595) | 5CGGCTTGTGTCCAGGC 3 |
| Sabin 2/RCP-2 | (2525-2544) | 5CCGTTGAAGGGATTACTAAA 3 |
| Sabin 3/RCP-1 | (2562-2580) | 5TAAGCTATCCTGTTGCTTC 3 |
| Sabin 3/RCP-2 | (2537-2553) | 5AGGGCGCCCTAACTTTG 3 |
Los números entre paréntesis indican el intervalo de la región del genoma que se va a amplificar, que codifica para parte de la proteína VP1, siguiendo el consenso del sistema de numeración de Toyoda y otros en 1984.5
Preparación del ARN viral. Las cepas y controles se sometieron a 3 ciclos consecutivos de congelación-descongelación (de -70 oC a temperatura ambiente) para favorecer la lisis celular. La extracción de ácidos nucleicos se realizó según el protocolo descrito por Abraham y otros en 1993,6 luego el ARN precipitado se resuspendió en 10 mL de agua libre de RNasas (tridestilada con dietilpirocarbonato al 0,01 %). La concentración de ácidos nucleicos y de contaminantes (proteínas, restos de fenol) se determinó mediante la lectura en un espectrofotómetro, según el esquema de trabajo de Davis y otros en 1986.7
Reacción de amplificación. La transcripción reversa (TR) y la RCP se llevaron a cabo en un solo paso. Se tomaron 10 m L del ácido ribonucleico (ARN) extraído y se agregaron en 90 mL de una mezcla de reacción 10 x (Tris-HCl 10 mM pH8,9; MgCl2 15 mM; KCl 80 mM; albúmina sérica bovina a 0,5 mg/mL; colato de sodio al 0,1 % y Tritón X-100 al 0,1 %) con 200mM de cada deoxinucleótido trifosfatado (dATP, dGTP, dCTP, dGTP), dithiothreito l 0,01 M; MgCl2 0,25 mM; 50 pmol de cada cebador; 20 unidades de enzima transcriptasa reversa del virus de la mieloblastosis aviaria (Boehringer Mamheim) y 2,5 unidades de enzima TaqDNA polimerasa (Promega). La TR-RCP fue programada en un termociclador (PTC 100TM. MJ Research, Inc. Watertown Mass.) realizándose la TR a 42 oC durante 30 min. Luego la mezcla se sometió a 95 oC por 5 min (inactivación de la transcriptasa reversa y desnaturalización de ADN complementario) y 30 ciclos de 94 oC, 45 s (desnaturalización); 60 oC, 45 s (hibridación) y 72 oC, 60 s (extensión). Después del último ciclo, la reacción se mantuvo a 72 oC durante 5 min (última extensión). Terminada la TR-RCP, el producto se conservó a -20 oC hasta su análisis.
Análisis del producto de la TR-RCP. Se llevó a cabo según el método de Ausubel y otros en 1989.8
Resultados
La purificación del ARN fue efectiva ya que su análisis demostró la presencia de ácidos nucleicos con valores aproximados a los 30 ng/mL. También se detectaron impurezas que aparentemente no interfirieron en los resultados de la RCP.
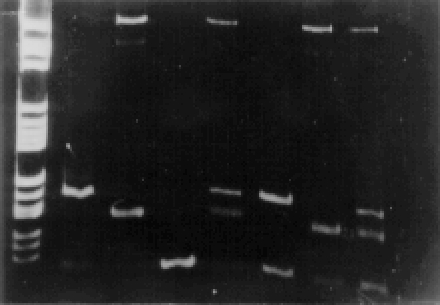
Al ARN aislado de cada una de las 3 cepas de referencia de los Poliovirus vacunales de Sabin tipos 1, 2 y 3 se le aplicó la TR-RCP con los 3 pares de cebadores. Se obtuvieron productos de ADN iguales a las tallas esperadas (97, 71 y 44 pb) para cada serotipo, de acuerdo con el patrón de peso molecular. La especificidad también fue probada mezclando los ARN de cada serotipo en diferentes combinaciones (P1+P2; P1+P3; P2+P3; P1+P2+P3) y luego de la TR-RCP se observó que, aun estando presentes los 3 pares de cebadores, sólo se produjo amplificación cuando el ARN de cada serotipo estuvo incluido en la mezcla de reacción (fig.). En algunas reacciones de amplificación se generaron bandas de ADN inespecíficas, quizás por el elevado número de moldes de las cepas, éstas no influyeron en la correcta interpretación de los resultados pues poseían pesos moleculares superiores a los 587 pb. Por su parte, los controles se comportaron según lo esperado: la cepa salvaje tipo 1, PV1 Mahoney/USA 42 mostró una banda correspondiente a los 97 pb, ya que la zona a amplificar es idéntica a su homóloga vacunal.9 Las cepas salvajes tipo 2 no mostraron bandas ya que éstas tienen marcadas diferencias con los cebadores correspondientes (9 divergencias con Sabin 2/RCP-1 y 8 divergencias con Sabin 2/RCP-2) que impiden la hibridación y por tanto la amplificación.10 La TR-RCP aplicada a otros Enterovirus mostró la especificidad de los cebadores al no producirse amplificación con el Coxsackievirus B4, el cual es un productor de PFa, y tiene una homología de 55 % (la mayor descrita) con los Poliovirus Sabin en la zona del genoma que codifica para la proteína VP1. La TR-RCP del control celular y el agua también fueron negativas, por lo que se deduce que no hubo hibridaciones inespecíficas ni contaminaciones, se excluye así la posibilidad de resultados falsos positivos. En la tabla también se observan los resultados de la TR-RCP aplicada al estudio de cepas. Todas las muestras estudiadas fueron positivas a Poliovirus vacunal Sabin, existiendo correlación con el serotipo definido previamente por pruebas de Nt.
Fig. Especificidad de los cebadores. PM: marcador de peso molecular (plásmido pBR-322 digerido con la enzima Hae III, Boehringer Mamheim). P1, P2, P3, P12, P13, P23, P123: productos de amplificación de las cepas Sabin y sus combinaciones. N: control negativo del agua destilada.
Discusión
La selección de un adecuado juego de cebadores que permita la amplificación específica de secuencias de cepas Sabin, y que excluya la amplificación cruzada con genotipos de Poliovirus salvajes, requiere la cuidadosa consideración de la frecuencia y de los mecanismos de evolución de diferentes regiones genómicas. Los sitios seleccionados deben mantener de forma sustancial su secuencia, a pesar de los cambios evolutivos, los cuales deben obedecer a impulsos genéticos, más que en respuesta a presiones selectivas. Para la diferenciación intratípica de Poliovirus, los intervalos genómicos con secuencias genotipo-específicas son: 1) codones de sitios antigénicos de neutralización,11,12 2) residuos 640-740 de la región 5NTR,13-15 3) codones 2-32 (serotipos 1 y 2) y 2-30 (serotipo 3) del dominio aminoterminal de VP1.5 Decidimos seleccionar estos últimos, ya que su secuencia nucleotídica y aminoacídica varía ampliamente entre los diferentes genotipos; este intervalo es flanqueado por secuencias de VP2, VP3 y VP1 que codifican para sitios antigénicos tipoespecíficos, por lo que la identificación basada en la TR-RCP se correlaciona con el serotipo. Además, el dominio VP1 aminoterminal se pliega en el interior del virión, no forma parte de sus antígenos inductores de anticuerpos neutralizantes y por tanto, es poco influenciado por la fuerte presión selectiva que éstos ejercen.16 Desde el punto de vista técnico, la selección de intervalos genómicos cortos contribuye a la sensibilidad analítica del sistema, ya que éstos son transcritos con más eficiencia en los pasos iniciales de la reacción en cadena.17
Con los resultados obtenidos en los ensayos de especificidad aplicados a las cepas controles se podría pensar que los genotipos de Poliovirus salvajes podrían hibridar con estos cebadores (como ocurrió con la cepa Mahoney) y llevar a resultados equívocos. A pesar de que se reconoce que en la naturaleza circulan numerosos virus salvajes con divergencias de hasta un 23 % en la región de la cápside, Yang y otros con los mismos cebadores estudiaron 30 aislamientos salvajes de América, Asia y África, observaron que, a pesar de las diferencias, no se obtenía amplificación y que los únicos Poliovirus "salvajes" cuya secuencia fue amplificada constituyeron cepas de referencia relacionadas de forma genética con las cepas vacunales inactivadas de Salk y atenuadas de Sabin.16 De esta forma se puede concluir que los aislamientos de Poliovirus "no Sabin" pueden ser discriminados por exclusión. Por otra parte, los aislamientos asociados con la vacunación pueden diferir de forma marcada de la cepa parental, ya que el paso y multiplicación en el intestino puede provocar frecuentes cambios genéticos y antigénicos.18 No obstante, los resultados obtenidos con las 23 cepas cubanas, las cuales proceden directamente del tracto digestivo, sugieren que los cambios que surjan no interfieren con la adecuada identificación con estos cebadores, debido a la internalización de la zona escogida. Por tanto reafirmamos la validez de la identificación y diferenciación intratípica por TR-RCP con los iniciadores de Yang.
Damos especial valor al resultado obtenido con la cepa 87/1993, la cual es hasta hoy la única del serotipo 1 aislada en Cuba a partir de un caso de PFA; esto se debe a que la reversión a la neurovirulencia requiere la acumulación de varias mutaciones nucleotídicas en zonas muy específicas del genoma. Todo esto hace que el Poliovirus vacunal de Sabin tipo 1 acapare sólo el 10 % de los casos de poliomielitis asociada con la vacunación.19,20 Otro dato de interés es observar que el Poliovirus vacunal es causa de casos esporádicos de meningoencefalitis viral como complicación neurológica más leve, debido a la amplia circulación del agente durante el período de campaña de vacunación antipoliomielítica con la vacuna oral, lo cual no ha sido reportado por otros autores. Es de destacar que con nuestro estudio aportamos un argumento más a favor de la no circulación de la forma salvaje en Cuba.
SUMMARY
The polimerase chain reaction techniques was introduced for the intratypic characterization of Poliovirus. Primers were used only to promote the amplification of the Sabin vaccine strains proved by electrophoretic run of the amplified DNA products (Sabin 1 - 97 pb, Sabin 2 - 71 pb, Sabin 3 - 44 pb) and whose specificity was satisfactorily verified. 23 Cuban poliovirus strains isolated and identified at the Laboratory of Enterovirus of the "Pedro Kourí" Tropical Medicine Institute from 1993 to 1994 were studied by this technique. All of them were of the vaccine type. It was observed how the Sabin vaccine poliovirus may be the cause of viral meningoencephalitis as a milder neurological complication. Tghis study provided one more evidence about the non circulation of the wild poliovirus in Cuba.
Subject headings: POLIOVIRUSES/isolation & purification; POLIOVIRUSES/genetics; POLYMERASE CHAIN REACTION/methods; RNA, VIRAL/isolation & purification.
REFERENCIAS BIBLIOGRÁFICAS
- Pan American Health Organization. Director announces. Campaign to erradicate poliomyelitis from the Americas by 1990. Bull Pan Am Health Organ 1985;19:213-5.
- Ciarnic R, Couillin P, Blonddel B, Cabu N, Boue A, Horodniceanu F. Natural variation of poliovirus neutralization epitopes. Infect Inmunol 1983;41:1217-25.
- Chen Fu Yang, Lina De, Holloway BP, Pallansch MA, Kew OM. Detection and identification of vaccine related poliovirus by the polimerase chain reaction. Virus Res 1991;20:159-79.
- Melnick JL, Wimber IL. Lyophilized combination of the enterovirus equine antisera. New LBM pools prepared from reserves of antisera stored frozen for two decades. Bull World Health Organ 1984;63:543-50.
- Toyoda H, Kohara M, Katoaka Y, Saganuma T, Omala T, Imura N, et al. Complete nucleotide sequence of all three poliovirus serotipe genomes: implication for genetic relationship, gene function and antigenic determinants. J Mol Biol 1984;174:561-85.
- Abrahan R, Chonmaitree T, Mc Combs J, Prabhakar B, Lo Verde PT, Ogra PL. Rapid detection of Poliovirus by reverse transcription and polimerase chain amplification: application for differenciation between Poliovirus and nonpoliovirus enteroviruses. J Clin Mic 1993;31(2):395-9.
- Davis LG, Dibner MD, Battey JF. Optical density. Analytical measurements. En: Basic methods in molecular biology. New York: Elsevier Science, 1986;327-8.
- Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA, et al. Preparation and analysis of DNA. En: Current protocols in molecular biology. New York: John Wiley, 1989; vol 1.
- Nomoto A, Omata T, Toyoda H, Kuge S, Horie H, Kataoka Y, et al. Complete nucleotide sequence of the attenuated poliovirus Sabin 1 strain genoma. Proc Natl Acad Sci USA 1982;79:5793-7.
- Kew OM, Nottay BK, Rico-Hesse RR, Pallansch MA. Molecular epidemiology of wild Poliovirus transmission. En: Kurstak E, Marusyk RG, Murphy FA, Van Regenmortel MHV, eds. Applied virology research. New York:Plenum, 1990; vol2:199-221.
- Minor PD, Ferguson M, Icenogle JP. Antigenic and molecular evolution of the vaccine strain of type 3 poliovirus during the period of excretion by a primary vaccine. J Gen Virol 1986;67:693-706.
- Page GS, Mosser AG, Hogle JM, Filman DJ, Rueckert RR, Chow M. Three-dimensional structure of Poliovirus serotype 1 neutralizing determinants. J Virol 1988;62:1781-94.
- Monica N la, Meriam C, Racaniello VR. Mapping of sequences required for mouse neurovirulence of Poliovirus type 2 Lansing. J Virol 1986;57:515-25.
- Hughes PJ, Evans DMA, Minor PD, Schild GC, Almond JW, Stanway G. The nucleotide sequence of type 3 Poliovirus isoleted during a recent outbreak of poliomielitis in Finland. J Gen Virol 1986;67:2093-2102.
- Pöyry T, Kinnunen L, Kew O, Hovi T. Type 3 poliovirus/Finland 1904 is genetically related to common Mediterranean strains. J Gen Virol 1990;71:2535-41.
- Chen Fu Yang, Lina De, Holloway BP, Pallansch MA, Kew OM. Detection and identification of vaccine related poliovirus by the polimerase chain reaction. Virus Res 1991;20:159-79.
- Berger CR, Wallace DM, Puskas RS, Eschenfeldt WH. Reverse transcriptase and its associated ribonuclease H. Interplay of two enzime activities controls the yield singled-stranded complementary deoxyribonucleid acid. Biochemistry 1983;22:2365-72.
- Contreras G, Dimok K, Furesz J, Gardell C, Hazlett D, Karpinski K, et al. Genetic characterization of Sabin types 1 and 3 poliovaccine virus following serial passage in the human intestinal tract. Biologicals 1992;20:15-26.
- Hellen CUT, Wimmer E. Enterovirus genetics. En: Rotbart HA, ed. Human enterovirus infections. Washington D.C: American Society for Microbiology 1995;25-53.
- Bouchard MJ, Lam DH, Racaniello VR. Determinants of attenuation and temperature sensitivity in the type 1 Poliovirus Sabin vaccine. J Virol 1995;69(8):4972-8.
Recibido: 29 de enero de 1997. Aprobado: 1 de diciembre de 1997.
Dra. Ivonne Ávalos Redón. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana, Cuba.
- Especialista de I Grado en Microbiología.
- Doctor en Ciencias Médicas. Especialista de II Grado en Microbiología. Investigador Titular.
- Master en Virología. Licenciado en Microbiología.
- Técnica Sanitaria "A".
- Master en Virología. Licenciada en Microbiología. Investigadora Agregada.
- Master en Virología. Licenciada en Microbiología. Investigadora Auxiliar.
- Licenciada en Microbiología.