Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.50 n.3 Ciudad de la Habana sep.-dic. 1998
Instituto de Medicina Tropical "Pedro Kourí"
Aplicación de la técnica de reacción en cadena de la polimerasa para la detección de secuencias de Papillomavirus humano
Resumen
Se aplicó la técnica de reacción en cadena de la polimerasa para la detección de secuencias de Papillomavirus humano (PVH) mediante controles de líneas celulares de cáncer cervical y tejidos obtenidos por biopsia con diagnóstico clínico positivo a PVH. Se utilizó un juego de oligonucleótidos consenso, que son complementarios a una región altamente conservada dentro del marco de lectura abierta E1 del genoma viral de los PVH que afectan la mucosa cervical. Con este juego de cebadores fue posible amplificar secuencias de ácido desoxirribonucleico (ADN) correspondientes a los PVH 6 y 11, considerados dentro del grupo de bajo riesgo y de los PVH 16, 18, 31 y 33 comprendidos en el grupo de alto riego. El estudio de la sensibilidad de la técnica de amplificación arrojó como resultado un nivel de detección de 3,5 partículas virales por cada genoma diploide celular.
Descriptores DeCS: PAPILLOMAVIRUS HUMANO/genética; REACCION EN CADENA POR POLIMERASA/métodos; ANALISIS DE SECUENCIA DE ADN/métodos; GENOMA VIRAL; ADN VIRAL/genética.
La familia Papovaviridae está constituida por un grupo heterogéneo de virus. Hasta el presente han sido identificados 77 genotipos diferentes de Papillomavirus en humanos. En la actualidad se ha podido determinar de forma parcial la secuencia de aproximadamente 30 tipos adicionales de Papillomavirus humanos (PVH), lo cual sugiere la existencia de un número total de 100 genotipos de PVH. De los 77 tipos cuyas secuencias han sido estudiadas por completo, más de 40 infectan la mucosa cervical y representan un grupo importante de patógenos que producen enfermedades de origen epitelial.1 La infección por PVH tiene una alta incidencia y demostrada asociación con el cáncer cervical.2 Una de las más fuertes evidencias de la función que desempeña este virus en la carcinogénesis lo constituye la presencia del ADN viral en más del 90 % de los carcinomas celulares escamosos del tracto genital.3,4 Se reconoce la vía sexual como la más común de diseminación del virus.5
La infección del cuello uterino por PVH puede ser asintomática o provocar una serie de manifestaciones que varían desde la presencia de condilomas benignos hasta la ocurrencia de alteraciones displásicas de diferentes grados y cáncer. La distinción de los diferentes estadios patológicos tiene un alto valor pronóstico ya que mientras los condilomas casi siempre permanecen como lesiones benignas, las neoplasias intraepiteliales cervicales (NIC) tienen una alta potencialidad de progresar al cáncer invasivo.6
Debido a la falta de un sistema celular susceptible para el aislamiento e identificación del virus y a la ausencia de pruebas serológicas adecuadas, el diagnóstico de la infección por PVH ha estado basado fundamentalmente en la detección del ácido nucleico viral.7
Desde la introducción de la reacción en cadena de la polimerasa (RCP) como nueva técnica para el diagnóstico, el estudio y análisis del ADN sufrió un vuelco tanto al nivel clínico como al investigativo. El método involucra la amplificación selectiva de secuencias específicas de ácidos nucleicos mediante síntesis enzimática, y constituye hasta el momento la técnica más sensible empleada en la detección del ADN de PVH. La RCP permite identificar un solo tipo viral, con la utilización de cebadores que se unan a secuencias específicas de éste o puede detectar un amplio espectro de tipos mediante cebadores generales o consenso.8
El presente trabajo tiene como objetivo aplicar la tecnología de la RCP para la detección de ADN de PVH empleando un juego de oligonucleótidos consenso correspondiente a una región altamente conservada dentro del genoma viral (región E1). Con estos cebadores nos propusimos determinar la sensibilidad y factibilidad de la técnica para amplificar secuencias de los tipos de PVH que se presentan con mayor frecuencia en el tracto genital. Contorni y Leoncini9 publicaron las secuencias de estos cebadores para su empleo en la amplificación de muestras parafinadas. Sin embargo, en nuestras condiciones, nos trazamos introducir la modificación de aplicar esta metodología a la detección de secuencias de PVH en muestras frescas de tejidos endocervicales obtenidos por biopsia con un diagnóstico clínico sugestivo de infección por PVH.
Métodos
Cultivos celulares y clones de PVH. Se emplearon las líneas de cáncer cervical humano Siha, Hela y CaSki obtenidas de la American Type Culture Collection. La línea Siha contiene una copia de PVH 16 por cada genoma celular, la Hela contiene entre 10 y 50 copias de PVH 18 y la CaSki entre 400 y 500 copias de PVH 16. Los cultivos se realizaron en medio Eagle suplementado con suero de ternera fetal al 10 % y se incubaron a 37 oC hasta que el crecimiento celular se hizo confluente. Estas líneas celulares fueron sometidas a extracción de ADN y fueron utilizadas como controles positivos para ser empleados en la reacción de amplificación.
Los genomas completos de PVH 6, 11, 16 y 18 clonados en el plásmido pBR 322 fueron donados gentilmente por el Departamento de Genética del Hospital Clinicoquirúrgico "Hermanos Ameijeiras". La extracción de las bandas de PVH empleadas en la reacción de amplificación se realizó por digestión con Bam HI (Amersham) para los tipos 6, 11 y 16, y con Eco RI (Amersham) para el tipo 18. Las bandas fueron purificadas mediante la técnica de agarosa de bajo punto de fusión.10
Tejidos endocervicales obtenidos por biopsia. Se estudiaron para la reacción de amplificación 10 muestras de tejidos endocervicales obtenidos por biopsia a partir de pacientes que asistieron a la consulta de patología de cuello del Hospital Ginecoobstétrico "Eusebio Hernández". El empleo de biopsias frescas constituye una modificación del protocolo original de Contorni y Leoncini,9 quienes describieron esta metodología para ser empleada en muestras parafinadas. El criterio de selección de este tipo de muestra fue la presencia de alteraciones citológicas que revelaran la presencia de infección por PVH y que comprendieron coilocitosis abundante y NIC de diferentes grados,11 incluidas dentro de las categorías 3, 4, 5 y 6 del sistema de clasificación establecido por la OMS en 1984. Además, estas muestras presentaban un diagnóstico clínico por colposcopia típico de infección por PVH. Las biopsias fueron transportadas a 4 oC en tubos de congelación con una solución salina fosfatada al 0,01 % de duodecil sulfato de sodio (PBS-SDS) y se almacenaron a -70 oC hasta su procesamiento.
Extracción de ADN de los cultivos celulares. Cuando el crecimiento se hizo confluente, las células fueron desprendidas por tripsinización y lavadas con PBS 1X. Después se centrifugaron a 3 500 rpm (centrífuga GR. 4.11, Jouan) y el sedimento se resuspendió en 500 mL de buffer lisis (Tris 10 mM pH 8, EDTA 10 mM, NaCl 10 mM, SDS 0,5 %).
Los detritos celulares se eliminaron centrifugando a 12 000 rpm (centrífuga D7200, Hettich) y el sobrenadante fue tratado durante 1 h a 56 oC con 30 U de proteinasa K (25 mg/mL); seguidamente se realizaron 2 extracciones de proteínas con fenol y fenol-cloroformo-alcohol isoamílico (25:24:1) con sus respectivas centrifugaciones a 12 000 rpm. El ADN se precipitó con 2,5 volúmenes de etanol absoluto y 1/10 de volumen de acetato de sodio 3 M pH 5,3 toda la noche a -20 oC.
Al día siguiente se centrifugó durante 30 min a 12 000 rpm, el precipitado se lavó con etanol al 70 %, se secó al vacío (Speed vac CHIST 1131, Blanc Labo) y fue resuspendido en 20 mL de agua bidestilada estéril. La calidad del proceso de extracción se determinó mediante una electroforesis en gel de agarosa al 0,8 % en tampón Tris-Borato-EDTA (TBE) y la visualización se realizó por tinción con bromuro de etidio en un transiluminador (Macrovue 2011, LKB). La concentración de ADN en todos los casos se determinó en un espectrofotómetro (1135, Unican).10
Extracción de ADN de los tejidos endocervicales. Los fragmentos de tejidos de aproximadamente 3 mm3 se resuspendieron en 500 mL de buffer lisis (Tris 10 mM pH 8, EDTA 10 mM, NaCl 10 mM, SDS 0,5 %). Para lograr la ruptura del tejido se utilizaron 20 mL de SDS (10 %) y 40 mL de proteinasa K (25 mg/mL), y se incubaron entre 2 y 3 h a 56 oC. Las proteínas celulares se eliminaron por extracción volumen/volumen con fenol-cloroformo-alcohol isoamílico (25:24:1) y la precipitación del ADN se efectuó con etanol absoluto y acetato de sodio. El ADN fue resuspendido en 20 mL de agua bidestilada estéril y la calidad del proceso de extracción se determinó de igual forma que en los cultivos celulares.
Reacción en cadena de la polimerasa. Los cebadores utilizados son complementarios a una secuencia nucleotídica del ORF E1 que aparece con un alto nivel de conservación entre los PVH asociados con las lesiones de la mucosa cervical. Sus secuencias son consideradas "secuencias consenso" ya que son capaces de amplificar regiones del genoma de 6 tipos virales muy frecuentes en el tracto genital (PVH 6, 11, 16, 18, 31 y 33) y generan un fragmento cuya talla oscila entre 577 y 594 pb en dependencia del tipo de PVH que se somete a la reacción de amplificación.9
La reacción de amplificación se realizó utilizando 1 ng del genoma de los tipos de PVH 6, 11, 16 y 18; 10 ng de ADN de los cultivos celulares Hela y CaSki; 100 ng de ADN extraído de la línea celular Siha y 100 ng de ADN proveniente de las biopsias endocervicales en un volumen final de 100 mL. Se incluyeron como controles negativos 10 ng de ADN de fibroblastos humanos, agua bidestilada estéril, así como 100 ng de ADN proveniente de una muestra de tejido endocervical negativa a PVH por la técnica de Southern blot.
La mezcla de reacción estuvo constituida por 10 mL del buffer de amplificación 10X (KCl 500 mM, Tris-HCl 100 mM pH 8,3; MgCl2 15 mM, gelatina 0,01 %), 200 mM de cada dNTP, 2,5 unidades de Taq. polimerasa (IPK), 50 pM de cada cebador y agua bidestilada estéril; posteriormente se procedió a efectuar la reacción de amplificación.9
Para la detección del producto amplificado se analizaron 10 mL de cada producto de reacción en una electroforesis en gel de agarosa al 1,2 % en tampón TBE. La visualización se realizó por coloración con bromuro de etidio.
Sensibilidad de la reacción de amplificación. La sensibilidad del ensayo para este juego de oligos es un aspecto que incluimos a la metodología original descrita por Contorni y Leoncini. Para ello se realizaron diluciones del genoma de PVH 18 en 100 ng de ADN de placenta humana. Las diluciones abarcaron un amplio rango de concentraciones del genoma del virus (1 ng, 100 pg, 10 pg, 1 pg, 100 fg, 10 fg , 1 fg y 0,1 fg/mL) y se amplificaron y visualizaron bajo las mismas condiciones y parámetros descritos anteriormente.
Los fragmentos originados a partir de la banda de PVH 18 fueron sometidos a una hibridación Southern blot. La sonda utilizada fue el genoma completo de PVH 18 marcado mediante el método de random primer o marcaje al azar.10 Se emplearon 20 unidades de la enzima Klenow (Promega) para marcar 100 ng de ADN de PVH 18 en presencia de 50 mCi de a P32dATP (Amersham).12
Análisis de restricción. Los productos amplificados a partir del ADN de las líneas celulares y de las bandas de los PVH 6, 11, 16 y 18 fueron tratados con fenol-cloroformo-alcohol isoamílico (25:24:1), precipitados con etanol absoluto y resuspendidos en un volumen final de 50 mL. Para la digestión enzimática se adicionaron 2 unidades de la enzima Alu I (Promega) y 5 mL del buffer de digestión H 10X (Promega), se incubó a 37 oC durante 2 h. Los patrones de digestión se analizaron en electroforesis en gel de agarosa al 1,2 % en tampón TBE, a 80 V durante 1 h.9
Resultados
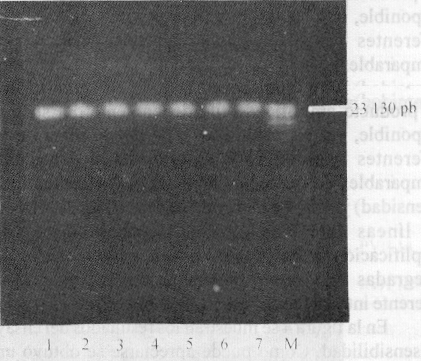
La extracción de ADN a partir de tejidos endocervicales obtenidos por biopsias resultó exitosa empleando la metodología descrita, al obtener un ácido nucleico con calidad y concentración adecuadas para su empleo óptimo en la RCP. En todos los casos la con-centración del ADN extraído fue de aproximadamente 200 ng/mL (fig. 1).
Fig. 1. Extracción de ADN a partir de tejidos endocervicales obtenidos por biopsia y de tejidos de líneas celulares de cáncer cervical. En todos los casos la concentración del ADN extraído de estas muestras empleadas como controles positivos fue de aproximadamente 200 ng/mL. Los carriles 1, 2, 3 y 4 corresponden a ADN de tejidos endocervicales de pacientes; y los carriles 5, 6 y 7 corresponden a ADN de tejidos de líneas celulares, Siha, Hela y CaSki, de forma respectiva M ( l ADN Hind III).
La RCP de los genomas completos de PVH 6, 11, 16 y 18 reveló la presencia de una banda con igual intensidad y pequeñas diferencias en la talla para cada genotipo viral (PVH 6 y 11-577 pb; PVH 16-583 pb; PVH 18-595 pb) al ser analizados 10 mL del producto amplificado en una electroforesis en gel de agarosa al 2 % (fig. 2).
Fig. 2. Productos amplificados a partir de los genomas completos de PVH 6, 11, 16 y 18. Las bandas amplificadas oscilan en un rango entre 577 y 595 pb. M(pBR 322 Hae III).
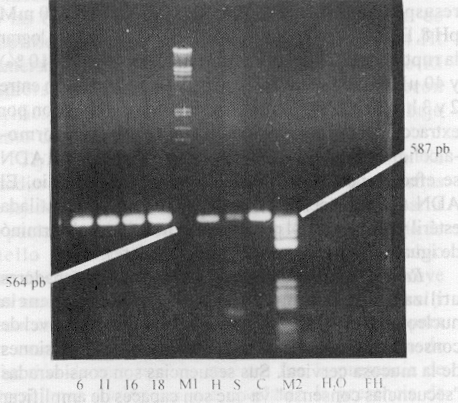
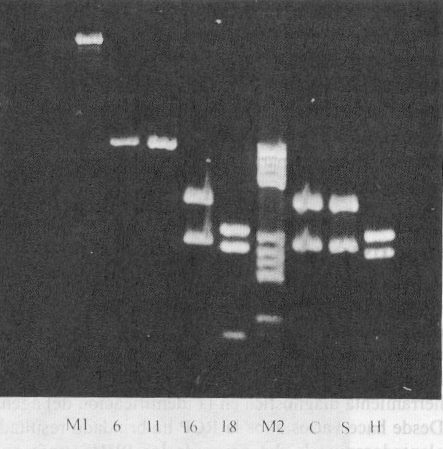
Los productos amplificados a partir de 10 ng de ADN proveniente de las líneas celulares Hela y CaSki y de 100 ng de ADN de Siha rindieron bandas cuyas tallas se corresponden con las de los genotipos virales integrados a sus respectivos cromosomas (fig. 3). La cantidad final de producto amplificado fue dependiente del sustrato disponible, ya que al utilizar 1 ng del genoma de los diferentes tipos de PVH se obtuvieron cantidades comparables de productos de la RCP (bandas de igual intensidad) y por otra parte, al emplear 10 ng de ADN de las líneas celulares, se obtuvieron productos de amplificación proporcionales al número de copias virales integradas a los cromosomas celulares (bandas con diferente intensidad; en orden creciente: Hela y CaSki).
Fig. 3. Productos amplificados a partir de los genomas de PVH 6, 11, 16 y 18 y del ADN extraído de las líneas celulares Siha (S), Hela (H) y CasKi (C). M1 (l Hind III/EcoR 1), M2 (pBR 322/Hae III), FH (fibroblastos humanos).
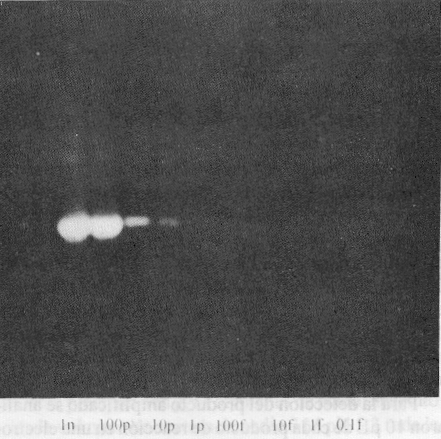
En la figura 4 se muestran los resultados del ensayo de sensibilidad. Como puede apreciarse se obtuvo una amplificación evidente hasta la cuarta dilución del genoma de PVH 18, correspondiente a 1 pg de ADN viral diluido en 100 ng de ADN de placenta humana, lo que significa que la técnica utilizada es capaz de detectar 3,5 genomas virales por cada genoma diploide celular.
Fig. 4. Resultados del ensayo de sensibilidad en el que se emplearon diluciones del genoma de PVH 18. Las diluciones seriadas de base 10 se realizaron desde 1ng hasta 0,1 fg. Se obtuvo amplificación hasta la cuarta dilución (1 pg).
La hibridación a que fueron sometidos los productos de la reacción de amplificación permitió detectar la presencia del ADN viral hasta 1 pg (fig. 5).
Fig. 5. Resultados del Southern blot a que fueron sometidos los productos amplificados en el ensayo de sensibilidad. Se detecta una señal de amplificación hasta 1 pg (cuarta dilución).
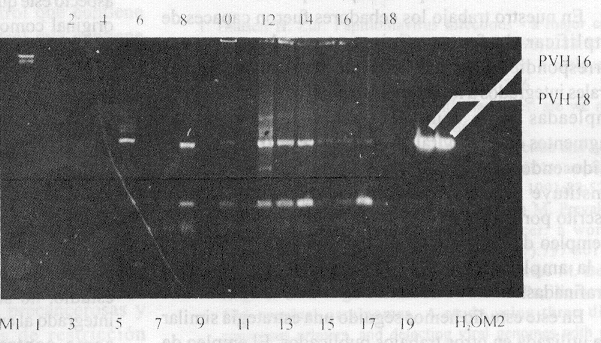
La aplicación de la RCP a la detección del genoma de PVH en tejidos de biopsias endocervicales y el posterior análisis de los productos amplificados mediante una electroforesis en gel de agarosa al 1,2 %, reveló la presencia de una simple banda con la talla esperada en los 10 casos empleados como controles positivos. Las diferencias que existen en la intensidad y talla de las bandas obtenidas se deben a que las concentraciones de ADN de cada muestra difieren en cada uno de los casos (fig. 6).
Fig. 6. Aplicación de la RCP a la detección de genomas de PVH a partir de ADN de tejidos endocervicales obtenidos por biopsia. Los carriles 3, 5, 8, 10, 12, 13, 14, 15, 16 y 17 se corresponden con los 10 casos empleados como controles positivos, los carriles 1, 2, 4, 6, 7, 9 11 y 18 se corresponden con los controles negativos.
El análisis de restricción con Alu I de los productos amplificados a partir del genoma de los PVH del grupo de bajo riesgo (tipos 6 y 11) muestra el mismo patrón de restricción (un fragmento único de 555 pb), mientras que la digestión de los productos de la RCP de los PVH del grupo de alto riesgo mostró un patrón característico para cada tipo: PVH 16 (343, 240 pb); PVH 18 (267, 238, 90 pb). Las líneas celulares Siha y CaSki contienen integrado en sus cromosomas el genoma de PVH 16, y la línea Hela contiene copias de PVH 18. La digestión con Alu I de los productos de la RCP a que fueron sometidos los ADN de estas líneas, mostró patrones de restricción coincidentes con los obtenidos para cada tipo viral integrado a sus cromosomas (fig. 7).
Fig. 7. Digestión con Alu I de los productos amplificados a partir de los genomas completos de PVH 6, PVH 11, PVH 16 y PVH 18, y de las líneas celulares Siha (S), Hela (H) y CaSki (K). M1 (l ADN/Hind III/ EcoRI), M2 (pBr 322/Hae III).
Discusión
Muchas evidencias apoyan que los PVH pueden desempeñar una función importante en el desarrollo del cáncer cervical, y que la detección del ADN viral en las lesiones constituya actualmente la más importante herramienta diagnóstica en la identificación del agente. Desde hace varios años la RCP ha brindado resultados alentadores en la detección de los PVH, y ofrece la posibilidad de identificar y tipificar el virus de una forma rápida y fácil.8
En los primeros estudios realizados utilizando la RCP se emplearon diferentes juegos de cebadores específicos para cada tipo viral, pero el posterior desarrollo de los protocolos de trabajo trajo como resultado el empleo de un solo juego de oligonucleótidos para detectar un amplio espectro de tipos virales.13,14
De los 30 tipos de PVH que son capaces de infectar la mucosa cervical, los tipos 6 y 11 son los más comunes en los condilomas acuminados y en las NIC de bajo grado, el tipo 16 es el más frecuente en las lesiones malignas y el PVH 18 es el que más se detecta en los carcinomas cervicales con un curso clínico agresivo.15
En nuestro estudio, el empleo de un juego de oligos consenso diseñados a partir de una región altamente conservada del ORF E1, nos permitió la amplificación de un fragmento de 577 a 595 pb del genoma de los 6 tipos de PVH mucosotrópicos más frecuentes en la población mundial, que constituye un sustrato adecuado para la tipificación de éstos mediante enzimas de restricción.9 La comparación de los fragmentos amplificados con el patrón de peso molecular permite comprobar que cada producto de la RCP presenta la talla adecuada. El empleo de un solo juego de cebadores en la detección de 4 tipos de PVH simplifica el trabajo en el muestreo masivo, minimiza la manipulación excesiva y el alto costo del diagnóstico que implicaría el uso de cebadores específicos para la identificación de cada tipo viral por separado.
En nuestro trabajo los cebadores fueron capaces de amplificar de forma eficiente tanto los genomas correspondientes a cada clon de PVH, los genomas virales integrados a los cromosomas de las líneas celulares empleadas como controles positivos; así como los fragmentos de ADN viral presentes en las 10 muestras de tejido endocervical. El empleo de biopsias frescas constituye una modificación del protocolo original descrito por Contorni y Leoncini,9 quienes describieron el empleo de estos oligos consenso para ser empleados en la amplificación de ADN de PVH en muestras parafinadas.
En este estudio hemos seguido una estrategia similar a la utilizada en otros trabajos publicados. El empleo de un juego de oligos consenso diseñados a partir del análisis de secuencias del ORF L1, permitió detectar 11 genotipos de PVH asociados con lesiones de la mucosa cervical.16
En 1991, se reporta un trabajo que se apoyó en las secuencias conservadas del ORF E6 para utilizar un juego de cebadores en la amplificación de 5 genotipos virales que fueron tipificados por Southern Blot.17 Dos años más tarde un grupo de investigadores desarrolló un protocolo que permitió la detección de 33 tipos de PVH cutáneos y genitales a partir de la amplificación de una secuencia conservada del ORF E1.14 En otro trabajo se analizó el ORF L1 para la síntesis de un juego de cebadores consenso que permitió la diferenciación en grupos con diferentes comportamientos biológicos de 18 tipos de PVH.8
El análisis computadorizado de las secuencias de ADN de los diferentes tipos de PVH revela que las regiones más conservadas se localizan en los ORF L1 y E1. Durante la integración del genoma viral al cromosoma celular se producen deleciones en la región E1/E2 que inducen a muchos investigadores a la utilización de secuencias de los ORF L1 y E6 que no son clivadas durante la integración.18 En nuestro trabajo se utilizan oligonucleótidos correspondientes a la región 5del ORF E1, que se mantiene intacta después del proceso de integración y posibilita la detección de PVH mediante la RCP sin que se produzcan resultados falsos positivos.
Se plantea que el contenido de ADN de una célula humana es de aproximadamente 5 pg, pudiéndose inferir que en 100 ng de ADN de placenta existen cerca de 20 000 células. También se conoce que en 1 pg de ADN de PVH hay involucrados unos 70 000 genomas virales.14,16 La relación que se establece entre los planteamientos anteriores y los resultados del ensayo de sensibilidad (70 000/20 000) permiten concluir que nuestro sistema es capaz de detectar 3,5 partículas virales por cada genoma diploide celular.
Los resultados obtenidos en el ensayo de sensibilidad, aspecto éste que también ha sido agregado a la metodología original como una modificación, son comparables a los reportados por otros investigadores. En un estudio en el que se aplicó la RCP para la identificación de tipos cutáneos y genitales de PVH se demostró que el método podía detectar entre 0,3 y 3 partículas virales por cada genoma diploide celular. Otro trabajo realizado en el que se utilizaron cebadores consenso para la detección de un amplio espectro de tipos de PVH, reportó un nivel de detección de 0,1 a 1 pg de ADN de PVH.14,16
La detección del ADN de PVH en 10 biopsias endocervicales demuestra que la RCP descrita en este estudio, no sólo detecta el genoma viral purificado o integrado al cromosoma de las líneas celulares empleadas como controles; sino que es capaz de amplificar secuencias de PVH de forma eficiente en muestras clínicas de pacientes con alteraciones citológicas donde el ADN viral puede encontrarse integrado por completo, parcialmente o en forma episomal.
La experiencia adquirida hasta el momento por la gran cantidad de investigaciones realizadas en torno a la función de los PVH en la génesis del cáncer cervicouterino, destaca la diferencia notable del porcentaje de positividad a PVH que existe entre mujeres con alteraciones citológicas y las que no presentan ningún tipo de afectación celular.
De manera general, la prevalencia de PVH asociado con alteraciones cervicales alcanza valores entre 50 y 90 % en mujeres con alteraciones citológicas, mientras que sólo se reportan valores del 15 al 20 % cuando se analizan mujeres con el cérvix normal.17 Al comprobar la utilidad de esta técnica en los controles utilizados queda demostrada la factibilidad de su uso para estudiar la prevalencia de PVH en un gran número de muestras clínicas a partir de tejidos endocervicales obtenidos por biopsia.
A pesar de que el Southern Blot es considerada la técnica de referencia en el diagnóstico de PVH, la RCP constituye un método ideal para la identificación del genoma viral de un modo rápido y relativamente fácil en aquellas muestras que rinden pequeñas cantidades de ácido nucleico. El empleo de biopsias endocervicales como controles en nuestro trabajo imposibilitó la utilización del Southern Blot en la detección del virus, ya que se requieren entre 5 y 10 mg de ADN para obtener resultados satisfactorios.19
Por la alta sensibilidad de la técnica, las contaminaciones pueden constituir un serio problema y dar lugar a resultados falsos positivos. La incorporación de controles negativos en las reacciones de amplificación permite un mejor análisis frente a la presencia de resultados falsos positivos causados por contaminaciones o a amplificaciones originadas por una hibridación inespecífica de los cebadores en la RCP (control que contiene agua en lugar de ADN, control que contiene ADN de fibroblastos humanos y control que contiene ADN extraído de una muestra de tejido endocervical con citología normal y negativa a PVH por Southern blot).
El tratamiento de los productos de la RCP con Alu I permite clasificar los PVH que aparecen con mayor frecuencia en las lesiones genitales en 2 grupos con diferentes comportamientos biológicos (alto y bajo riesgos). Para los tipos 6 y 11 que son los más comunes en los condilomas acuminados y las NIC de bajo grado se obtuvo una banda de 555 pb que permite incluirlos dentro del grupo de bajo riesgo. Para los PVH 16 y 18 que son los tipos más frecuentes en las lesiones precancerosas y cancerosas se obtuvieron patrones de restricción característicos, que no sólo posibilitan su clasificación dentro del grupo de alto riesgo, sino también su tipificación individual.20 Los resultados que se obtuvieron en este trabajo al digerir los productos amplificados tanto de los genomas de PVH como del ADN de las líneas celulares pueden ser utilizados en la tipificación de PVH presentes en muestras clínicas de tejidos endocervicales. Los patrones de digestión que se obtuvieron para cada tipo viral coinciden con los reportados en un ensayo similar realizado para la detección de PVH en tejidos endocervicales embebidos en parafina.9
Existen estudios donde la tipificación se realiza empleando varias enzimas de restricción. En estos casos la complejidad del análisis de los patrones de digestión, así como el alto costo de las enzimas, permite corroborar las ventajas de la tipificación utilizando una sola enzima de restricción con la que se obtienen resultados fáciles de interpretar porque los patrones de restricción para los PVH de alto y bajo riesgos difieren notablemente.4
Summary
The polymerase chain reaction technique was applied to detect sequences of human Papillomavirus (HPV) by controls of cellular lines of cervical cancer and of tissues obtained through biopsy with a HPV-positive clinical diagnosis. A set of consensus oligonucleotides, which are complementary to a highly conserved region within the open reading frame El of the viral genome of HPV affecting the cervical mucosa, was used. With these primers it was possible to amplify DNA sequences corresponding to HPV 6 and 11, considered in the low risk group, and to HPV 16, 18, 31 and 33, included in the high risk group. The study of the sensitivity of the amplification technique showed a level of detection of 3,5 viral particles per each cellular diploid genome.
Subject headings: PAPILLOMAVIRUS, HUMAN/genetics; POLYMERASE CHAIN REACTION/methods; SEQUENCE ANALYSIS DNA/methods; GENOME, VIRAL; DNA, VIRAL/genetics.
Referencias Bibliográficas
- Hausen H. Zur: Papillomavirus infections - a major cause of human cancers. Biochim Biophis Acta 1996;1288:55-78.
- Franco EL. Viral etiology of cervical cancer: a critique of the evidence. Rev Infec Dis 1991;13:1195-206.
- Roman A, Fife KA. Human papillomaviruses: are we ready to type? J Clin Microbiol 1993;2:166:190.
- Yoshikawa H, Kawama T, Kitagawa K. Detection and typing of multiple genital human papillomaviruses by DNA amplification with consensus primers. Jpn J Cancer Res 1991;82:524-31.
- Bosch FX, Manos MM, Muñoz N, Sherman M. Prevalence of human Papillomavirus in cervical cancer: a worldwide perspective. J Nat Cancer Inst 1995;11(87):797-801.
- Karlsen F, Kristensen G, Holm R, Chitemerese M, Berner A, Hagmar BM. High incidence of human Papillomavirus in 146 cervical carcinomas. A study using three different consensus primers, and detecting viral genomes with putative deletions. Eur J Cancer 1995;9(31):1511-6.
- Venuti A, Badaracco G, Marcante ML. Detection and typing of human Papillomavirus by single hibridization. J Virol Methods 1994;51:115-24.
- Jacobs MV, Roda Husman AM de, Brule AJ van der. Group- -specific between high and low risk human Papillomavirus genotypes by general primer mediated PCR and two cocktails of oligonucleotide probes. J Clin Microbiol 1995;3(4):901-5.
- Contorni M, Leoncini P. Typing of human Papillomavirus DNAs by restriction endonuclease differenciation mapping of the PCR product. J Virol Methods 1993;41:29-39.
- Maniatis T, Fritsch EF, Sambrok J. Molecular cloning: a laboratory manual. New York: Cold Spring Harbor Laboratory, 1982:93-466.
- Papanicolau GN. A new procedure for staining vaginal smears. Science 1942;45:438-9.
- Renart J, Reiser JJ, Stark GR. Protocols for Nucleic Acid Blotting and Hybridization, Proc Natl Acad Sci USA 1987;76:3116-20.
- Evander M, Wadell G. A general pair of primer for amplification and detection of genital human Papillomavirus types. J Virol 1982;44:393-400.
- Tieben LM, Schegget J, Minaar RP. Detection of cutaneous and genital HPV types in clinical samples by PCR using consensus primer. J Virol Methods 1993;42:265-80.
- Lorinez AT, Reid R, Jenison AB. Human Papillomavirus infection of the cervix: relative risk associations of 15 common anogenital types. Obstet Gynecol 1992;79:328-37.
- Snidjers P, Brule AJ van der, Schrjnemakres H. The use of general primers in the polymerase chain reaction permits the detection of a broad spectrum of human Papillomavirus genotypes. J Gen Virol 1990;1032:19-37.
- Tham KW, Chow TK, Lim-Tam SK. Diagnostic sensitivity of a polymerase chain reaction and Southern blot hybridization for the detection of human Papillomavirus DNA in biopsy specimens from cervical lesions. Am J Clin Pathol 1991;95(5):638-46.
- Romancruck H, Howley PM. Disruption of either the E1 or the E2 regulatory gene of human Papillomavirus type 16 increases viral immortalization capacity. Proc Natl Acad Sci USA 1992;89:3159-63.
- Southern EM. Detection of specific sequences among DNA fragments separated by gel electrophoresis. J Mol Biol 1975;98:503-17.
- Howley PM, Schegel R. The human Papillomavirus an over view. Am J Med 1988;85:155-8.
Recibido: 28 de agosto de 1997. Aprobado: 4 de enero de 1998.
Lic. Yudira Soto. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana, Cuba.
- Master en Ciencias. Licenciada en Microbiología. Aspirante a Investigadora. Instituto de Medicina Tropical "Pedro Kourí" (IPK).
- Master en Ciencias. Licenciada en Microbiología. Investigadora Agregada. IPK.
- Especialista de I Grado en Ginecología y Obstetricia. Hospital Ginecoobstétrico "Eusebio Hernández".
- Licenciado en Microbiología. IPK.
- Licenciada en Bioquímica. Aspirante a Investigadora. IPK.
- Técnica en Procesos biológicos. IPK.