Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.52 n.1 Ciudad de la Habana ene.-abr. 2000
Instituto de Medicina Tropical "Pedro Kourí"
Detección rápida de Enterovirus mediante un método directo de reacción en cadena de la polimerasa
Resumen
Se desarrolló un método directo y económico de reacción en cadena de la polimerasa para la detección de Enterovirus, que no requiere pasos previos de extracción de ácido ribonucleico (ARN), a partir de sobrenadantes de cultivos celulares infectados. El sistema se desarrolló mediante cebadores universales del género Enterovirus y cebadores específicos de la cepa vacunal Sabin 1. Los resultados obtenidos demuestran que la ausencia de métodos de extracción y purificación de ARN previos a la reacción no afectan la sensibilidad y especificidad del sistema, lo que posibilita que pueda ser utilizado para la detección rápida de genomas de Enterovirus e identificación de cepas vacunales de poliovirus.
Descriptores DeCS: REACCION EN CADENA POR POLIMERASA/métodos; ENTEROVIRUS/aislamiento y purificación; ENTEROVIRUS/genética
El género Enterovirus (EV) agrupa a 68 serotipos, la mayoría de ellos son importantes patógenos humanos, responsables de una amplia variedad de enfermedades.1 Los Poliovirus, miembros prototipos del género, son causantes de la poliomielitis la cual ha tenido como principales víctimas a los niños. Desde el descubrimiento de las vacunas a finales de los años 50 y principio de los 60, la incidencia de esta enfermedad ha disminuido drásticamente y en la actualidad el Poliovirus salvaje ha sido reemplazado por las cepas atenuadas derivadas de la vacuna.2 Los EV no poliovirus (Coxsackievirus A y B, Echovirus y Enterovirus 68 al 71) son responsables de más de 5 a 10 000 000 de infecciones sintomáticas cada año en el mundo y por esta razón constituyen los agentes etiológicos más comunes de las meningitis.3
El diagnóstico de laboratorio de estos agentes virales es de gran importancia porque las infecciones que producen son clínicamente imposibles de distinguir de las causadas por patógenos bacte-rianos y otros virus.4
Desde su descubrimiento en 1954 el aislamiento en cultivo celular se mantiene como la técnica de oro para el diagnóstico de los EV. Sin embargo, con esta técnica un aislamiento puede ser identificado como Enterovirus sólo por la observación del efecto citopático (ECP), lo cual requiere en muchos casos un alto nivel de experiencia.5 La identificación de los aislamientos se realiza mediante neutralización con la utilización de la mezcla de sueros de Lim Benyesh Melnick (LMB),6 que reconocen 42 serotipos de EV, pero los resultados por esta técnica demoran al menos 5 d. Para la detección e identificación rápida de los EV se han desarrollado varios sistemas de reacción en cadena de la polimerasa (RCP)7-9 pero han sido altamente costosos, lo que dificulta su aplicación en países del tercer mundo.
En el presente trabajo se propone un método directo y económico de RCP, que no requiere pasos previos de extracción de ácido ribonucleico (ARN), para la detección rápida de genomas de Enterovirus e identificación de cepas vacunales de Poliovirus a partir de sobrenadantes de cultivos celulares infectados.
Métodos
Virus (muestras): Se utilizaron 3 cepas de referencia de Poliovirus vacunales: Sabin 1, 2, 3 recibidas del National Institute for Biological Standards and Control, de Inglaterra y una cepa de un Enterovirus no Poliovirus: Coxsackievirus B-4 (# 488 de 1994) aislada en el Laboratorio de Enterovirus del Instituto de Medicina Tropical "Pedro Kourí" (IPK) a partir de las heces fecales (HF) de un caso de parálisis fláccida aguda (PFA) e identificada por neutralización mediante el esquema de LBM.6 Todos estos virus habían sido propagados en cultivo de células Vero (células de línea de riñón de mono verde africano adulto normal Cercopithecus aetiops, American Type Culture Collection).10 La cepa Sabin 1 tenía un título infectivo de 10-6 TCID50/0,1 mL. Se utilizaron además 6 aislamientos virales, de los cuales 3 fueron obtenidos de las HF de niños inmunodeficientes, después de concluida la campaña de vacunación con la vacuna oral de Poliovirus vivo atenuada e identificados por neutralización como Poliovirus tipo 1. El resto procedían de las HF de pacientes con diagnóstico clínico de meningoencefalitis viral (MEV) que no pudieron ser identificados mediante la mezcla de sueros de LBM.6
Oligonucleótidos sintéticos. Se emplearon 2 pares de cebadores:
- Cebadores diseñados por Rotbart y otros7 (EV) que hibridan con sitios altamente conservados dentro de la región 5' no codificante del genoma de los Enterovirus, y permiten la amplificación de una secuencia de 154 pb.
- Cebadores diseñados por Yang y otros8 (Sabin 1) que permiten la amplificación de una secuencia de 97 pb en la zona VP1 de la cepa vacunal Sabin 1, se excluyen la amplificación cruzada con genotipos de Poliovirus salvajes y con los Poliovirus vacunales Sabin 2 y Sabin 3.
Ambos pares de cebadores fueron sintetizados en el Centro de Ingeniería Genética y Biotecnología (CIGB) de Cuba por el método de b-cianoetil-fosforamidite.11 La secuencia nucleotídica así como su localización en el genoma viral se muestran en la tabla.
Controles negativos. En cada uno de los ensayos de amplificación se incluyeron 2 controles negativos:
- Control celular: sobrenadante de cultivos de células Vero no infectadas.
- Control de reactivos: contiene todos los componentes necesarios para la RCP, excepto la muestra.
Extracción de ARN-RCP. Se tomaron 300 mL de sobrenadante de cultivo celular y se realizó la extracción de ARN mediante el método del isotiocianato de guanidina (GnTCN) descrito por Lanciotti y otros.12 La reacción de amplificación a partir del ARN extraído se llevó a cabo con la misma metodología descrita para el método directo de RCP.
Tabla. Secuencia nucleotídica y posición en el genoma de los cebadores
| Código | Posición | Secuencia |
| Sabin 1/RCP-1 | (2584-2601) | 5' TCCACTGGCTTCAGTGTT 3 |
| 'Sabin 1/RCP-2 | (2505-2523) | 5' AGGTCAGATGCTTGAAAGC 3' |
| EV/RCP-1 | (584-603) | 5' ACCGACGAATACCACTGTTA 3' |
| EV/RCP-2 | (450-474) | 5' CCTCCGGCCCCTGAATGCGGC TAAT 3' |
RCP-1: cebadores complementarios, RCP-2: cebadores paralelos.
Método directo de RCP. Se tomó 1 mL de sobrenadante de cultivo celular y se adicionó a una mezcla de reacción, preparada en un volumen de 17 mL que contenía 100 ng de cada uno de los cebadores a utilizar, tampón de amplificación 1X (tris HCl 67 mM y pH 8,8, NH4SO4 17 mM, EDTA 6 mM, MgCl2 2mM, 2-mercaptoetanol 1mM) y 100 mM de cada deoxinucleótido trifosfatado (dATP, dGTP, dCTP, dTTP) (Pharmacia Biotech). Cada tubo de reacción se incubó durante 5 min a 95 oC en un bloque térmico (Bioblock, Francia) para provocar la liberación del ARN viral. Al concluir esta incubación los tubos se colocaron inmediatamente en hielo y se adicionaron 32 mL de la mezcla de las enzimas que contenía 5 U de inhibidor de Rnasa (Promega), 1,66 U de transcriptasa inversa AMV (Promega), 1,26 U de Taq ADN polimerasa (Promega). Los tubos fueron puestos en un termociclador (PTC 100 TM. MJ Research, Inc. Watertown Mass.) a las temperaturas de 42 oC durante 20 min, para la transcripción inversa, a lo que siguieron 30 ciclos con temperaturas de 95 0C por 45 s, 55 0C por 45 s y 70 0C durante 45 s, finalmente la mezcla de reacción se dejó durante 5 min a 70 0C.
Para la detección del producto amplificado se analizaron 10 mL de cada tubo de reacción en una electroforesis en gel de agarosa 4 % en tampón TBE (Tris-Bobato 0,09 M, EDTA 2 mM, pH 8). Se utilizó el marcador de peso molecular PCR Markers (Promega)13 que contiene 6 fragmentos de ácido desoxirribonucleico (ADN) con tallas de 50, 150, 300, 750 y 1 000 pb en igual proporción para el primer ensayo. En el resto de los experimentos se mezcló un producto de la RCP con segmentos amplificados con la utilización de los cebadores Sabin 1/RCP-1 y Sabin 1/RCP-2 con un producto de la RCP con segmentos amplificados mediante los cebadores EV/RCP-1 y EV/RCP-2 obtenidos en el primer ensayo y esta mezcla se utilizó como patrón.
Resultados
Especificidad de la RCP
La especificidad del método directo de RCP, sin pasos previos de extracción de ARN, fue comparada con el método de extracción del GnTCN, con la utilización de cada una de las cepas de Poliovirus vacunales y la cepa de Enterovirus no Poliovirus frente a los 2 pares de cebadores.
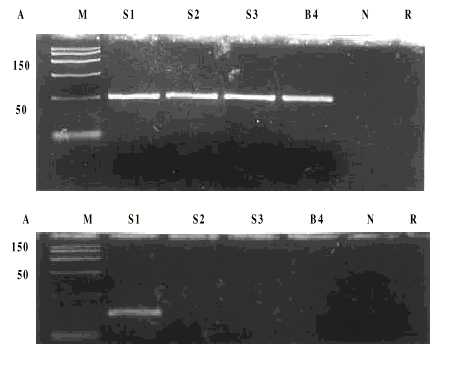
Al aplicar el método directo de RCP se observaron bandas de amplificación de 154 pb en todas las cepas estudiadas al utilizar los cebadores EV/RCP-1 y EV/RCP-2. Sin embargo, con los cebadores Sabin 1/RCP-1 y Sabin 1/RCP-2 se obtuvo un único fragmento de 97 pb en la cepa de Sabin 1. Con el método de extracción de ARN previo a la RCP se obtuvieron resultados similares (fig. 1).
A: Método directo de RCP. B: RCP con extracción previa mediante el método del GnTCN, M: marca-dor de peso molecular, S1: Sabin 1, S2: Sabin 2, S3: Sabin 3, B4: Coxsackievirus B4, N: control celular (Vero), R: control de reactivos.
Fig.1. Especificidad del método directo de reacción en cadena de la polimerasa (RCP): productos de RCP con los cebadores Sabin 1/RCP 1 y Sabin 1/RCP 2 (parte inferior).
Sensibilidad de la RCP
Para determinar si el límite de sensibilidad de la RCP con los pares de cebadores utilizados no era afectado por la ausencia de una extracción y purificación previa de ARN, se realizaron diluciones de la cepa Sabin 1 desde 1 000 TCID50 hasta 0,1 TCID50 en medio de mantenimiento de las células Vero (medio Parker 199). Fueron divididas en 2 partes, a una de ellas se le realizó extracción y purificación de ARN por el método del GnTCN y seguidamente fue sometida a la RCP, y a la otra se le aplicó el método directo de RCP.
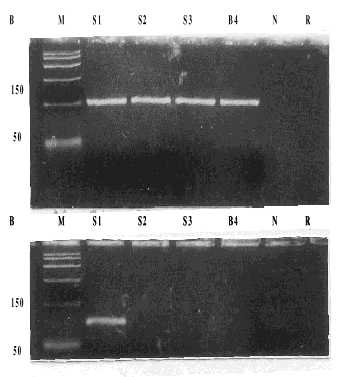
Con el método de RCP directo, sin pasos previos de extracción de ácidos nucleicos, el límite de detección fue de 10 TCID50 con los cebadores EV/RCP-1 y EV/RCP-2 y de 1 TCID50 para los cebadores Sabin 1/RCP-1 y Sabin 1/RCP-2. De forma similar, con el método de extracción y purificación previo a la RT-RCP el límite de detección fue de 10 TCID50 con los cebadores EV/RCP-1 y EV/RCP-2 y de 1TCID50 para los cebadores Sabin 1/RCP-1 Sabin 1/RCP-2 (fig. 2).
A: Método directo de RCP. B: RCP con extracción previa mediante el método del GnTCN. M: patrón de peso molecular, 1: 1 000 TCID50, 2:100 TCID50, 3: 10 TCID50, 4: 1 TCID50, 5: 0,1 TCID50, N: control celular (Vero), R: control de reactivos.
Fig. 2. Sensibilidad del método directo de la reacción en cadena de la polimerasa (RCP): productos de RCP con los cebadores Sabin 1/RCP 1 y Sabin 1/RCP 2 (parte inferior).
Utilización del método con aislamientos virales
La utilidad del método directo de RCP con los cebadores empleados fue comprobada al estudiar por este sistema 6 aislamientos virales obtenidos en el laboratorio de Enterovirus del IPK.
Se pudo corroborar que 3 aislamientos previa-mente determinados por neutralización como Poliovirus tipo 1 pertenecían a este serotipo, y se confirmó la naturaleza vacunal de los Poliovirus aislados. Se pudo confirmar además que 3 aisla-mientos de pacientes con diagnóstico clínico de MEV y que no pudieron ser identificados por neutralización mediante la mezcla de LBM correspondían a Enterovirus.
Discusión
Los autores de este trabajo desarrollaron un método directo de RCP que no requería pasos previos de extracción de ácidos nucleicos. Se pudo comprobar la especificidad de este sistema con la utilización de cebadores universales del género Enterovirus y cebadores específicos para la cepa vacunal Sabin 1 frente a las 3 cepas de Poliovirus que presentan más de 85 % de homología entre ellas y la cepa de Coxsackievirus B4 que muestra una homología en la zona VP1 de 55 % con respecto a los Poliovirus Sabin.14
Los resultados obtenidos concuerdan tanto con los de Rotbart y otros,7 Yang y otros 8 que han utilizado estos mismos cebadores en reacciones de amplificación con extracciones previas de ARN, como con los de Kilpatrick y otros15 que trabajó con un método semejante al utilizado aquí, pero con cebadores diferentes. De esta forma se demostró que la ausencia de una extracción previa de material genético no afecta la especificidad de la RCP.
Starnabach y otros16 fueron los primeros en reportar las evidencias iniciales de que la RCP puede realizarse a partir de ADN no purificado, liberado de las células por calentamiento. Sin embargo, planteó que el método puede provocar una disminución de la sensibilidad de la reacción, por la obtención de ácidos nucleicos unidos a proteínas estructurales o de fusión.
La obtención de iguales límites de sensibilidad al comparar el sistema de RCP directo con el método de extracción del GnTCN, el cual ha sido reportado por muchos autores como uno de los más potentes métodos de extracción de ácidos nucleicos,12,17-19 demuestra que la ausencia de métodos de extracción y purificación de ARN previos a la RCP no interfiere en su sensibilidad, posiblemente por la ausencia de sustancias inhibidoras de la reacción.20
Es de destacar que aunque los límites de detección fueron diferentes para el mismo molde con ambos cebadores, éstos se encontraban entre los valores reportados por diversos autores. Rotbart y otros,7 con el mismo tipo de cebadores reportaron límites de sensibilidad de 100 TCID50 en una RCP simple, a partir de una extracción de ARN basado en tratamiento con duodecil sulfato de sodio, proteinasa K y fenol-cloroformo. Por otra parte, Glimaker y otros21 al usar los cebadores de Rotbart encontraron límites de sensibilidad de 0,25 TCID50 mediante el método del GnTCN pero seguido de una RCP anidada. Yang y otros8 reportaron límites de detección similares a los encontrados en este trabajo al usar los cebadores para la cepa Sabin 1.
Por la alta sensibilidad de la RCP, los contaminantes pueden constituir un serio problema y dar lugar con facilidad a resultados positivos falsos. Las 3 fuentes fundamentales de conta-minación son: el intercambio de material genético entre las muestras trabajadas, los reactivos contaminados y la acumulación de productos de la RCP por la amplificación repetida de una misma secuencia en el laboratorio.22 En este trabajo, las posibilidades de contaminación quedan descartadas por los resultados obtenidos en los controles negativos incluidos en cada uno de los ensayos. Además, en la práctica diaria se asumió un grupo de medidas semejantes a las descritas por Kitchin y Bootman23 para la disminución de los riesgos de contaminación.
Por estas razones, los autores de este trabajo consideran que los resultados presentados aquí son confiables y de esta forma el laboratorio del IPK dispone de un sistema específico y sensible de RCP directo, que no requiere pasos previos de extracción de ácidos nucleicos, a partir de sobrenadantes de cultivos celulares infectados.
La utilización de este sistema es de gran importancia porque se produce una reducción considerable del tiempo del ensayo y permite la identificación rápida de un solo tipo viral, al emplear cebadores que se unan a secuencias específicas del mismo tipo o de un amplio espectro de éstos, si se utilizan cebadores generales o consenso. Por este sistema se evita la utilización de un engorroso método de extracción de ácidos nucleicos que demora casi 2 h cuando se quieran amplificar secuencias de ARN de Enterovirus a partir de sobrenadantes de cultivos celulares infectados para estudios de diagnóstico o investigación.
Con este método también disminuyen los riesgos de contaminación y es más factible la manipulación de un amplio número de muestras. Además, el uso de pequeñas cantidades de reactivos al aprovechar el tampón de amplificación de la reacción para la liberación del ARN viral y no emplear reactivos adicionales para la extracción del ácido nucleico, así como la utilización de sólo 1 mL de muestra y de las enzimas a bajas concentraciones de trabajo (1,66 U de transcriptasa inversa, 1,26 U de Taq DNA polimerasa), indican que este método es más económico que otros sistemas de RCP reportados, lo que permite que sea más aplicable en países del tercer mundo.
Los resultados obtenidos al aplicar este método con los aislamientos virales demuestran que el sistema debe ser empleado para la identificación y caracterización intratípica de cepas de Poliovirus derivadas de la vacuna en sustitución del método de RCP utilizado en el laboratorio del IPK.24 La técnica también puede ser empleada para la confirmación de los aislamientos de Enterovirus cuya identificación mediante la mezcla de LBM se haga difícil. Además debe ser utilizado cuando sea necesario acelerar el diagnóstico con la utilización de un solo pase de la muestra en cultivo celular, pues en la siembra primaria se hace imposible distinguir si los efectos citopáticos producidos son por causa de la multiplicación viral o por la presencia de sustancias tóxicas en la muestra inoculada.
Esta última aplicación tiene gran importancia si se tiene en cuenta que los EV son los responsables de una amplia variedad de enfermedades, y muchas de ellas son causadas por un serotipo en particular y al mismo tiempo varios serotipos pueden estar asociados con una misma enfermedad.1 Por tanto, para dar un diagnóstico lo más importante es detectar y confirmar rápido si una enfermedad con cuadro clínico sospechoso a Enterovirus es causada por estos agentes, sin necesidad de identificar un serotipo en particular.9
De acuerdo con los resultados obtenidos en el presente trabajo este método pudiera ser usado para la detección de genomas de Enterovirus directamente en muestras de pacientes. Por estas razones se recomienda en próximos estudios aplicarlo con muestras clínicas, lo que aceleraría en gran medida el diagnóstico de estos importantes agentes virales.
Summary
For the detection of Enterovirus, we devised a direct economical method of polymerase chain reaction which does not require a previous extraction of ribonucleic acid and uses infected cell culture supernatants. The system was developed on the basis of universal primers of Enterovirus and specific primers of vaccinal strain Sabin 1. The achieved results proved that the non-existance of methods of RNA extraction and purification prior to the reaction does not affect the susceptibility and specificity of the system, in the rapid detection of Enterovirus genomes and identification of vaccinal strains of poliovirus.
Subject headings: POLYMERASE CHAIN REACTION/METHODS; ENTEROVIRUS/isolation and purification; ENTEROVIRUS/genetic.
Referencias Bibliográficas
- Melnick JL. Enterovirus. Poliovirus, Coxsackievirus, Echovirus, and newer Enterovirus. En: Fields BN, Knipe DM, Howley PM, eds. Virology 3 ed. Nueva York:Raven,1996;vol2:655-712.
- Kew O, Mulders M, Lipskaya G, da Silva E, Pallansh M. Molecular epidemiology of Poliovirus. Virology 1995;6:401-14.
- Tyler K. Viral and prion disease of the nervous system. En: Harrinson TR, ed. Harrinson's Principles of Internal Medicine. 13thed. Nueva York:Mc Graw Hill, 1994;2309-12.
- Wildin S, Chonmaitree T. The importance of virology laboratory in the diagnosis and management of viral meningitis. Am J Dis Child 1987;141:454-7.
- Abraham R, Chonmaitree T, Combs J, Prabhakar B, Loverde P, Ogra P. Rapid detection of poliovirus by reverse transcription and polymerase chain reaction: aplication for differentiation between poliovirus and nonpolovirus enteroviruses. J Clin Microbiol 1993;31(2):395-9.
- Melnick JL, Wimbert IL. Liophilized combination of the enterovirus equine antisera. New LBM pools prepared from reserves of antisera stored frozen for two decades. Bull WHO 1984;63:543-50.
- Rotbart HA. Enzymatic RNA amplification of the enteroviruses. J Clin Microbiol 1990;28:438-42.
- Yang CF, Lina DE, Holloway BP, Pallansch MA, Kew OM. Detection and identification of vaccine related polioviurus by the polymerase chain reaction. Virus Res 1991;20:159-79.
- Rotbart H. Nucleic acid detection for Enterovirus. Clin Microbiol Rev 1991;4(2):156-8.
- Hay R, Caputo J, Chen TR, Macy M, Mc Clintoch P, Reidy. Catalogue of cell lines and hybridomas. 7. ed. Maryland: American type culture collections:1992:48.
- Sinha ND, Biernat J, Mc Manus J, Köster H. Polymer support oligonucleotide synthesis XVIII: use of B-cyanoethyl-N, N-dialkyamino-/N-morpholino phosphoramidite of deoxynucleosides for the synthesis of DNA fragments simplifying deprotection and isolation of the final product. Nucleic Acid Res 1984;12:4539-57.
- Lanciotti RS, Calisher CH, Gubler DJ, Chang GJ, Vordam V. Rapid detection typing of dengue viruses from clinical samples by using reverse transcriptase polymerase chain reaction. J Clin Microbiol 1992;30:545-51.
- Sambrook J, Fritsch EF, Manatis T. Molecular cloning: a laboratory manual. Nueva York:Ed. Cold Spring Harbor. Laboratory:1989.
- Palmenberg AC. Sequence aligments of picornaviral capsid proteins. En: Semiler B, Enrefeld E, eds. Molecular aspects of picornavirus infection and detection. Washinton DC: ASM Publication, 1988:211-41.
- Kilpatrick DR, Nottay B, Yang SJ, Da Silva E, Peñaranda S, Pallansch M, et al. Serotype-specific identification of polioviruses by PCR using primers containing mixed-base or deoxyinosine residues at positions of codon degeneracy. J Clin Microbiol 1998;36(2):352-7.
- Starnbach MN, Falcow S, Tomkins LS. Species specific detection of Legionella pneumophilia in water by DNA amplification and hibridization. J Clin Microbiol 1989;1257-61.
- Chomzynsky P, Sacchi N. Single step method of RNA isolation guanidium thiocyanate-phenol-cloroform extraction. Anal Biochem 1987;162:156-9.
- Casas I, Powell L, Klappe PE, Cleator GM. New method for the extraction of viral RNA DNA from cerebrospinal fluid for use in the polymerase chain reaction assay. J Virol Methods 1995;53:25-36.
- Rosario DD, Suárez CM, Rodríguez RR, Soler MN, Guzmán MGT. Identificación rápida de los serotipos del virus del dengue mediante la reacción en cadena de la polimerasa. Rev Cubana Med Trop 1996;48(3):155-69.
- Wilson IG, Cooper JE, Gilmour A. Some factors inhibiting amplification of the Staphylococcus aureus enterotoxin C1 (sec) by PCR. Int J Food Microbiol 1993;22:55-62.
- Glimaker M, Johansson B, Olcén P, Ehrnst A, Forsgren M. Detection of enteroviral RNA by polymerase chain reaction in cerebrospinal fluid from patients with aseptic meningitis. Scand J Infect Dis 1993;25:547-57.
- Sarkar G, Sommer SS. Shedding light on PCR contamination. Nature 1990;347:27.
- Kitchin PA, Bootman YS. Quality control of polymerase chain reaction. Rev Med Virol 1993;3:107-14.
- Avalos I, Sarmiento L, Más P, Mune M, Palomera R, Bello M. Caracterización intratípica de Poliovirus por la técnica de reacción en cadena de la polimerasa. Rev Cubana Med Trop 1998;50(2):89-92.
Recibido: 9 de noviembre de 1999. Aprobado: 20 de diciembre de 1999.
Lic. Luis Sarmiento Pérez. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana, Cuba. Correo electrónico: ciipk@ipk.sld.cu.
1 Máster en Virología. Licenciado en Microbiología.
2 Especialista de I Grado en Microbiología. Investigadora Agregada.
3 Licenciada en Microbiología.
4 Doctor en Ciencias Médicas. Especialista de II Grado en Microbiología. Investigador de Mérito.
5 Máster en Virología. Licenciada en Microbiología. Investigadora Auxiliar.
6 Técnica en Microbiología.
7 Especialista de I Grado en Microbiología.